Mol:FL7AACGL0104
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 55 60 0 0 0 0 0 0 0 0999 V2000 | + | 55 60 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.1189 2.3575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1189 2.3575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1189 1.5325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1189 1.5325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4045 1.1200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4045 1.1200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3100 1.5325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3100 1.5325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3100 2.3575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3100 2.3575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4045 2.7700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4045 2.7700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0245 1.1200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0245 1.1200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7390 1.5325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7390 1.5325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7390 2.3575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7390 2.3575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0245 2.7700 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 1.0245 2.7700 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5155 2.8058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5155 2.8058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2300 2.3933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2300 2.3933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9444 2.8058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9444 2.8058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9444 3.6308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9444 3.6308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2300 4.0433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2300 4.0433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5155 3.6308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5155 3.6308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5846 4.0004 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5846 4.0004 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3831 1.1606 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3831 1.1606 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6989 2.6923 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6989 2.6923 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4045 0.5576 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4045 0.5576 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6045 2.4247 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6045 2.4247 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4223 -1.1685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4223 -1.1685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2470 -1.1360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2470 -1.1360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4336 -0.3321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4336 -0.3321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0038 0.2645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0038 0.2645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1791 0.2321 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1791 0.2321 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9924 -0.5718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9924 -0.5718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4009 -0.4380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4009 -0.4380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9323 -0.7337 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9323 -0.7337 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6250 -1.8211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6250 -1.8211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8088 -1.4125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8088 -1.4125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9201 -0.7818 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9201 -0.7818 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9417 -3.4218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9417 -3.4218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7555 -3.2847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7555 -3.2847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8382 -2.4636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8382 -2.4636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3279 -1.7992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3279 -1.7992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5140 -1.9363 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5140 -1.9363 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4312 -2.7574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4312 -2.7574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8274 -2.7000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8274 -2.7000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2258 -4.0433 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2258 -4.0433 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3561 -3.6147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3561 -3.6147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3780 -2.8477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3780 -2.8477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3109 -3.1112 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3109 -3.1112 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0709 -0.3923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0709 -0.3923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3411 0.3212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3411 0.3212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8948 -0.3923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8948 -0.3923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3067 -1.1058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3067 -1.1058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1306 -1.1058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1306 -1.1058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5431 -0.3913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5431 -0.3913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3681 -0.3913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3681 -0.3913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7806 -1.1058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7806 -1.1058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3681 -1.8203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3681 -1.8203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5431 -1.8203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5431 -1.8203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6045 -1.1058 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6045 -1.1058 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7800 0.3222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7800 0.3222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 8 18 1 0 0 0 0 | + | 8 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 13 21 1 0 0 0 0 | + | 13 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 18 1 0 0 0 0 | + | 25 18 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 33 40 1 0 0 0 0 | + | 33 40 1 0 0 0 0 |
| − | 34 41 1 0 0 0 0 | + | 34 41 1 0 0 0 0 |
| − | 35 42 1 0 0 0 0 | + | 35 42 1 0 0 0 0 |
| − | 44 45 2 0 0 0 0 | + | 44 45 2 0 0 0 0 |
| − | 44 46 1 0 0 0 0 | + | 44 46 1 0 0 0 0 |
| − | 46 47 2 0 0 0 0 | + | 46 47 2 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 2 0 0 0 0 | + | 48 49 2 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 51 2 0 0 0 0 | + | 50 51 2 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 52 53 2 0 0 0 0 | + | 52 53 2 0 0 0 0 |
| − | 53 48 1 0 0 0 0 | + | 53 48 1 0 0 0 0 |
| − | 51 54 1 0 0 0 0 | + | 51 54 1 0 0 0 0 |
| − | 50 55 1 0 0 0 0 | + | 50 55 1 0 0 0 0 |
| − | 43 39 1 0 0 0 0 | + | 43 39 1 0 0 0 0 |
| − | 29 44 1 0 0 0 0 | + | 29 44 1 0 0 0 0 |
| − | 36 32 1 0 0 0 0 | + | 36 32 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AACGL0104 | + | ID FL7AACGL0104 |
| − | KNApSAcK_ID C00014760 | + | KNApSAcK_ID C00014760 |
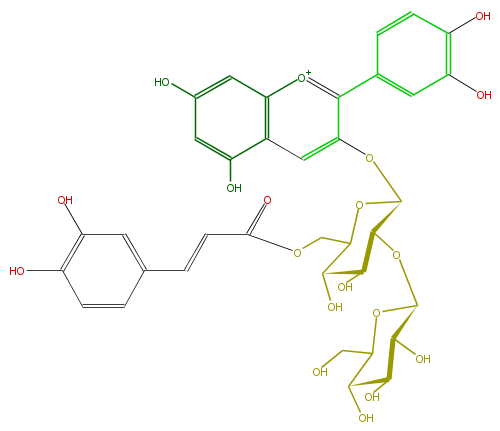
| − | NAME Cyanidin 3-(2-glucosyl-6-caffeoylglucoside) | + | NAME Cyanidin 3-(2-glucosyl-6-caffeoylglucoside) |
| − | CAS_RN 215227-67-1 | + | CAS_RN 215227-67-1 |
| − | FORMULA C36H37O19 | + | FORMULA C36H37O19 |
| − | EXACTMASS 773.192904002 | + | EXACTMASS 773.192904002 |
| − | AVERAGEMASS 773.66758 | + | AVERAGEMASS 773.66758 |
| − | SMILES O(C(=O)C=Cc(c6)ccc(c6O)O)CC(O1)C(O)C(O)C(OC(C(O)5)OC(CO)C(C(O)5)O)C1Oc(c3c(c4)cc(O)c(c4)O)cc(c2[o+1]3)c(cc(c2)O)O | + | SMILES O(C(=O)C=Cc(c6)ccc(c6O)O)CC(O1)C(O)C(O)C(OC(C(O)5)OC(CO)C(C(O)5)O)C1Oc(c3c(c4)cc(O)c(c4)O)cc(c2[o+1]3)c(cc(c2)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
55 60 0 0 0 0 0 0 0 0999 V2000
-1.1189 2.3575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1189 1.5325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4045 1.1200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3100 1.5325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3100 2.3575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4045 2.7700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0245 1.1200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7390 1.5325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7390 2.3575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0245 2.7700 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
2.5155 2.8058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2300 2.3933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9444 2.8058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9444 3.6308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2300 4.0433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5155 3.6308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5846 4.0004 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3831 1.1606 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6989 2.6923 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4045 0.5576 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6045 2.4247 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4223 -1.1685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2470 -1.1360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4336 -0.3321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0038 0.2645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1791 0.2321 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9924 -0.5718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4009 -0.4380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9323 -0.7337 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6250 -1.8211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8088 -1.4125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9201 -0.7818 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9417 -3.4218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7555 -3.2847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8382 -2.4636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3279 -1.7992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5140 -1.9363 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4312 -2.7574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8274 -2.7000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2258 -4.0433 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3561 -3.6147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3780 -2.8477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3109 -3.1112 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0709 -0.3923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3411 0.3212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8948 -0.3923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3067 -1.1058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1306 -1.1058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5431 -0.3913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3681 -0.3913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7806 -1.1058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3681 -1.8203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5431 -1.8203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6045 -1.1058 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7800 0.3222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
14 17 1 0 0 0 0
8 18 1 0 0 0 0
1 19 1 0 0 0 0
3 20 1 0 0 0 0
13 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
22 30 1 0 0 0 0
23 31 1 0 0 0 0
24 32 1 0 0 0 0
25 18 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
38 39 1 0 0 0 0
33 40 1 0 0 0 0
34 41 1 0 0 0 0
35 42 1 0 0 0 0
44 45 2 0 0 0 0
44 46 1 0 0 0 0
46 47 2 0 0 0 0
47 48 1 0 0 0 0
48 49 2 0 0 0 0
49 50 1 0 0 0 0
50 51 2 0 0 0 0
51 52 1 0 0 0 0
52 53 2 0 0 0 0
53 48 1 0 0 0 0
51 54 1 0 0 0 0
50 55 1 0 0 0 0
43 39 1 0 0 0 0
29 44 1 0 0 0 0
36 32 1 0 0 0 0
S SKP 8
ID FL7AACGL0104
KNApSAcK_ID C00014760
NAME Cyanidin 3-(2-glucosyl-6-caffeoylglucoside)
CAS_RN 215227-67-1
FORMULA C36H37O19
EXACTMASS 773.192904002
AVERAGEMASS 773.66758
SMILES O(C(=O)C=Cc(c6)ccc(c6O)O)CC(O1)C(O)C(O)C(OC(C(O)5)OC(CO)C(C(O)5)O)C1Oc(c3c(c4)cc(O)c(c4)O)cc(c2[o+1]3)c(cc(c2)O)O
M END