Mol:FL7AACGL0081
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 78 85 0 0 0 0 0 0 0 0999 V2000 | + | 78 85 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.9086 -0.5888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9086 -0.5888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9086 -1.2311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9086 -1.2311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3523 -1.5523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3523 -1.5523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2040 -1.2311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2040 -1.2311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2040 -0.5888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2040 -0.5888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3523 -0.2676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3523 -0.2676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7603 -1.5523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7603 -1.5523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3166 -1.2311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3166 -1.2311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3166 -0.5888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3166 -0.5888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7603 -0.2676 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 0.7603 -0.2676 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8727 -0.2677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8727 -0.2677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4397 -0.5950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4397 -0.5950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0067 -0.2677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0067 -0.2677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0067 0.3870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0067 0.3870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4397 0.7143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4397 0.7143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8727 0.3870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8727 0.3870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4647 -0.2677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4647 -0.2677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5735 0.7142 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5735 0.7142 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3523 -2.1944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3523 -2.1944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7327 -1.9518 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7327 -1.9518 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4397 1.3688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4397 1.3688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9933 -1.6105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9933 -1.6105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6925 -2.1315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6925 -2.1315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2710 -1.9662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2710 -1.9662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8529 -2.1315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8529 -2.1315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1538 -1.6105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1538 -1.6105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5753 -1.7758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5753 -1.7758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4346 -1.7602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4346 -1.7602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8135 -1.3632 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8135 -1.3632 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8571 -1.7990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8571 -1.7990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2186 -2.0588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2186 -2.0588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8503 -2.4234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8503 -2.4234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2186 -1.3688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2186 -1.3688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8333 -1.0140 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8333 -1.0140 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8333 -0.4005 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8333 -0.4005 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2764 -0.0790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2764 -0.0790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2764 0.5640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2764 0.5640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8333 0.8855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8333 0.8855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3902 0.5640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3902 0.5640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3902 -0.0790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3902 -0.0790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8333 1.5276 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8333 1.5276 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8616 -3.1905 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8616 -3.1905 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4905 -3.6805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4905 -3.6805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9560 -3.4726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9560 -3.4726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4402 -3.4670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4402 -3.4670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8150 -3.0922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8150 -3.0922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2826 -3.3390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2826 -3.3390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3755 -3.4872 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3755 -3.4872 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0691 -3.7086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0691 -3.7086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6497 -3.9867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6497 -3.9867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8022 -2.2992 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8022 -2.2992 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7971 -2.8245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7971 -2.8245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9462 0.8851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9462 0.8851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2321 1.5326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2321 1.5326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6033 1.0427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6033 1.0427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1378 1.2505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1378 1.2505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6535 1.2561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6535 1.2561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2788 1.6310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2788 1.6310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8111 1.3842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8111 1.3842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2817 1.2360 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2817 1.2360 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0247 1.0145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0247 1.0145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4440 0.7364 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4440 0.7364 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3067 1.8886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3067 1.8886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3067 2.5474 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3067 2.5474 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8158 2.8413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8158 2.8413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4819 3.4195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4819 3.4195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4816 2.8413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4816 2.8413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8111 3.4122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8111 3.4122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4925 3.4122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4925 3.4122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8242 2.8377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8242 2.8377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4876 2.8377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4876 2.8377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8193 3.4122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8193 3.4122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4876 3.9867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4876 3.9867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8242 3.9867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8242 3.9867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4820 3.4122 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4820 3.4122 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8189 2.2637 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8189 2.2637 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2318 -2.1447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2318 -2.1447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9462 -2.5572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9462 -2.5572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 23 20 1 0 0 0 0 | + | 23 20 1 0 0 0 0 |
| − | 31 32 2 0 0 0 0 | + | 31 32 2 0 0 0 0 |
| − | 31 33 1 0 0 0 0 | + | 31 33 1 0 0 0 0 |
| − | 33 34 2 0 0 0 0 | + | 33 34 2 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 2 0 0 0 0 | + | 35 36 2 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 2 0 0 0 0 | + | 37 38 2 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 40 35 1 0 0 0 0 | + | 40 35 1 0 0 0 0 |
| − | 38 41 1 0 0 0 0 | + | 38 41 1 0 0 0 0 |
| − | 42 43 1 1 0 0 0 | + | 42 43 1 1 0 0 0 |
| − | 43 44 1 1 0 0 0 | + | 43 44 1 1 0 0 0 |
| − | 45 44 1 1 0 0 0 | + | 45 44 1 1 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 42 1 0 0 0 0 | + | 47 42 1 0 0 0 0 |
| − | 42 48 1 0 0 0 0 | + | 42 48 1 0 0 0 0 |
| − | 43 49 1 0 0 0 0 | + | 43 49 1 0 0 0 0 |
| − | 44 50 1 0 0 0 0 | + | 44 50 1 0 0 0 0 |
| − | 31 51 1 0 0 0 0 | + | 31 51 1 0 0 0 0 |
| − | 47 52 1 0 0 0 0 | + | 47 52 1 0 0 0 0 |
| − | 52 51 1 0 0 0 0 | + | 52 51 1 0 0 0 0 |
| − | 45 19 1 0 0 0 0 | + | 45 19 1 0 0 0 0 |
| − | 39 53 1 0 0 0 0 | + | 39 53 1 0 0 0 0 |
| − | 54 55 1 1 0 0 0 | + | 54 55 1 1 0 0 0 |
| − | 55 56 1 1 0 0 0 | + | 55 56 1 1 0 0 0 |
| − | 57 56 1 1 0 0 0 | + | 57 56 1 1 0 0 0 |
| − | 57 58 1 0 0 0 0 | + | 57 58 1 0 0 0 0 |
| − | 58 59 1 0 0 0 0 | + | 58 59 1 0 0 0 0 |
| − | 59 54 1 0 0 0 0 | + | 59 54 1 0 0 0 0 |
| − | 54 60 1 0 0 0 0 | + | 54 60 1 0 0 0 0 |
| − | 55 61 1 0 0 0 0 | + | 55 61 1 0 0 0 0 |
| − | 56 62 1 0 0 0 0 | + | 56 62 1 0 0 0 0 |
| − | 57 21 1 0 0 0 0 | + | 57 21 1 0 0 0 0 |
| − | 59 63 1 0 0 0 0 | + | 59 63 1 0 0 0 0 |
| − | 63 64 1 0 0 0 0 | + | 63 64 1 0 0 0 0 |
| − | 64 65 1 0 0 0 0 | + | 64 65 1 0 0 0 0 |
| − | 65 66 2 0 0 0 0 | + | 65 66 2 0 0 0 0 |
| − | 65 67 1 0 0 0 0 | + | 65 67 1 0 0 0 0 |
| − | 67 68 2 0 0 0 0 | + | 67 68 2 0 0 0 0 |
| − | 68 69 1 0 0 0 0 | + | 68 69 1 0 0 0 0 |
| − | 69 70 2 0 0 0 0 | + | 69 70 2 0 0 0 0 |
| − | 70 71 1 0 0 0 0 | + | 70 71 1 0 0 0 0 |
| − | 71 72 2 0 0 0 0 | + | 71 72 2 0 0 0 0 |
| − | 72 73 1 0 0 0 0 | + | 72 73 1 0 0 0 0 |
| − | 73 74 2 0 0 0 0 | + | 73 74 2 0 0 0 0 |
| − | 74 69 1 0 0 0 0 | + | 74 69 1 0 0 0 0 |
| − | 72 75 1 0 0 0 0 | + | 72 75 1 0 0 0 0 |
| − | 71 76 1 0 0 0 0 | + | 71 76 1 0 0 0 0 |
| − | 25 77 1 0 0 0 0 | + | 25 77 1 0 0 0 0 |
| − | 77 78 1 0 0 0 0 | + | 77 78 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 77 78 | + | M SAL 1 2 77 78 |
| − | M SBL 1 1 84 | + | M SBL 1 1 84 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 84 -5.5787 5.2556 | + | M SBV 1 84 -5.5787 5.2556 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AACGL0081 | + | ID FL7AACGL0081 |
| − | KNApSAcK_ID C00006857 | + | KNApSAcK_ID C00006857 |
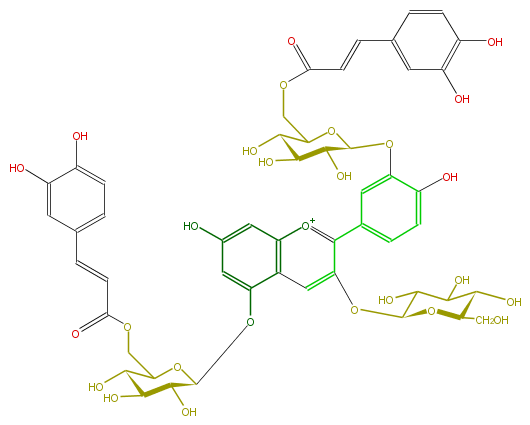
| − | NAME Gentiocyanin B;Cyanidin 3-glucoside-5,3'-di-(caffeoylglucoside) | + | NAME Gentiocyanin B;Cyanidin 3-glucoside-5,3'-di-(caffeoylglucoside) |
| − | CAS_RN 170663-60-2 | + | CAS_RN 170663-60-2 |
| − | FORMULA C51H53O27 | + | FORMULA C51H53O27 |
| − | EXACTMASS 1097.27742149 | + | EXACTMASS 1097.27742149 |
| − | AVERAGEMASS 1097.95032 | + | AVERAGEMASS 1097.95032 |
| − | SMILES Oc(c1)c(O)ccc(C=CC(OCC(O2)C(C(O)C(O)C2Oc(c3)c(ccc(c([o+1]4)c(OC(C(O)8)OC(CO)C(O)C(O)8)cc(c(OC(C6O)OC(COC(C=Cc(c7)ccc(O)c7O)=O)C(C6O)O)5)c4cc(c5)O)3)O)O)=O)1 | + | SMILES Oc(c1)c(O)ccc(C=CC(OCC(O2)C(C(O)C(O)C2Oc(c3)c(ccc(c([o+1]4)c(OC(C(O)8)OC(CO)C(O)C(O)8)cc(c(OC(C6O)OC(COC(C=Cc(c7)ccc(O)c7O)=O)C(C6O)O)5)c4cc(c5)O)3)O)O)=O)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
78 85 0 0 0 0 0 0 0 0999 V2000
-0.9086 -0.5888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9086 -1.2311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3523 -1.5523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2040 -1.2311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2040 -0.5888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3523 -0.2676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7603 -1.5523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3166 -1.2311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3166 -0.5888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7603 -0.2676 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
1.8727 -0.2677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4397 -0.5950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0067 -0.2677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0067 0.3870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4397 0.7143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8727 0.3870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4647 -0.2677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5735 0.7142 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3523 -2.1944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7327 -1.9518 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4397 1.3688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9933 -1.6105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6925 -2.1315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2710 -1.9662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8529 -2.1315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1538 -1.6105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5753 -1.7758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4346 -1.7602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8135 -1.3632 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8571 -1.7990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2186 -2.0588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8503 -2.4234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2186 -1.3688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8333 -1.0140 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8333 -0.4005 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2764 -0.0790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2764 0.5640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8333 0.8855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3902 0.5640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3902 -0.0790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8333 1.5276 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8616 -3.1905 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4905 -3.6805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9560 -3.4726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4402 -3.4670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8150 -3.0922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2826 -3.3390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3755 -3.4872 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0691 -3.7086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6497 -3.9867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8022 -2.2992 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7971 -2.8245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9462 0.8851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2321 1.5326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6033 1.0427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1378 1.2505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6535 1.2561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2788 1.6310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8111 1.3842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2817 1.2360 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0247 1.0145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4440 0.7364 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3067 1.8886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3067 2.5474 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8158 2.8413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4819 3.4195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4816 2.8413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8111 3.4122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4925 3.4122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8242 2.8377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4876 2.8377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8193 3.4122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4876 3.9867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8242 3.9867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4820 3.4122 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8189 2.2637 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2318 -2.1447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9462 -2.5572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
15 21 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
27 29 1 0 0 0 0
26 30 1 0 0 0 0
23 20 1 0 0 0 0
31 32 2 0 0 0 0
31 33 1 0 0 0 0
33 34 2 0 0 0 0
34 35 1 0 0 0 0
35 36 2 0 0 0 0
36 37 1 0 0 0 0
37 38 2 0 0 0 0
38 39 1 0 0 0 0
39 40 2 0 0 0 0
40 35 1 0 0 0 0
38 41 1 0 0 0 0
42 43 1 1 0 0 0
43 44 1 1 0 0 0
45 44 1 1 0 0 0
45 46 1 0 0 0 0
46 47 1 0 0 0 0
47 42 1 0 0 0 0
42 48 1 0 0 0 0
43 49 1 0 0 0 0
44 50 1 0 0 0 0
31 51 1 0 0 0 0
47 52 1 0 0 0 0
52 51 1 0 0 0 0
45 19 1 0 0 0 0
39 53 1 0 0 0 0
54 55 1 1 0 0 0
55 56 1 1 0 0 0
57 56 1 1 0 0 0
57 58 1 0 0 0 0
58 59 1 0 0 0 0
59 54 1 0 0 0 0
54 60 1 0 0 0 0
55 61 1 0 0 0 0
56 62 1 0 0 0 0
57 21 1 0 0 0 0
59 63 1 0 0 0 0
63 64 1 0 0 0 0
64 65 1 0 0 0 0
65 66 2 0 0 0 0
65 67 1 0 0 0 0
67 68 2 0 0 0 0
68 69 1 0 0 0 0
69 70 2 0 0 0 0
70 71 1 0 0 0 0
71 72 2 0 0 0 0
72 73 1 0 0 0 0
73 74 2 0 0 0 0
74 69 1 0 0 0 0
72 75 1 0 0 0 0
71 76 1 0 0 0 0
25 77 1 0 0 0 0
77 78 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 77 78
M SBL 1 1 84
M SMT 1 CH2OH
M SBV 1 84 -5.5787 5.2556
S SKP 8
ID FL7AACGL0081
KNApSAcK_ID C00006857
NAME Gentiocyanin B;Cyanidin 3-glucoside-5,3'-di-(caffeoylglucoside)
CAS_RN 170663-60-2
FORMULA C51H53O27
EXACTMASS 1097.27742149
AVERAGEMASS 1097.95032
SMILES Oc(c1)c(O)ccc(C=CC(OCC(O2)C(C(O)C(O)C2Oc(c3)c(ccc(c([o+1]4)c(OC(C(O)8)OC(CO)C(O)C(O)8)cc(c(OC(C6O)OC(COC(C=Cc(c7)ccc(O)c7O)=O)C(C6O)O)5)c4cc(c5)O)3)O)O)=O)1
M END