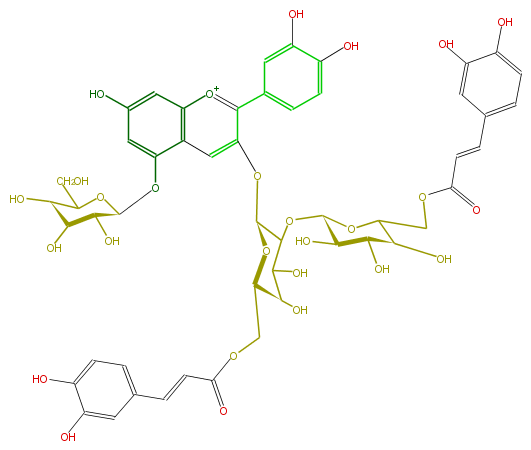
Mol:FL7AACGL0073
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 78 85 0 0 0 0 0 0 0 0999 V2000 | + | 78 85 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.6129 2.3989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6129 2.3989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6129 1.7566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6129 1.7566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0566 1.4354 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0566 1.4354 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5003 1.7566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5003 1.7566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5003 2.3989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5003 2.3989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0566 2.7201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0566 2.7201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9440 1.4354 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9440 1.4354 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3877 1.7566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3877 1.7566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3877 2.3989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3877 2.3989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9440 2.7201 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -0.9440 2.7201 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1684 2.7200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1684 2.7200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7354 2.3927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7354 2.3927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3023 2.7200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3023 2.7200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3023 3.3747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3023 3.3747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7354 3.7020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7354 3.7020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1684 3.3747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1684 3.3747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1690 2.7200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1690 2.7200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8692 3.7019 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8692 3.7019 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0566 0.7933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0566 0.7933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0284 1.0359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0284 1.0359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5069 -0.2782 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.5069 -0.2782 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.0001 0.1094 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.0001 0.1094 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.2094 -0.4886 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2094 -0.4886 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0437 -1.1866 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.0437 -1.1866 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.5069 -1.5046 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.5069 -1.5046 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.3322 -0.8932 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.3322 -0.8932 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.6816 0.1266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6816 0.1266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8337 -0.9371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8337 -0.9371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0504 -2.2629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0504 -2.2629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3514 0.2241 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 1.3514 0.2241 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 1.6549 -0.3678 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.6549 -0.3678 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.2635 -0.2468 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.2635 -0.2468 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.8143 -0.3378 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.8143 -0.3378 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.4853 0.1322 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.4853 0.1322 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.9405 -0.0430 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9405 -0.0430 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0328 -0.2891 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0328 -0.2891 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4934 -0.8523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4934 -0.8523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7675 -0.6401 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7675 -0.6401 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7354 4.3565 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7354 4.3565 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4613 -2.6212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4613 -2.6212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2154 0.5191 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -4.2154 0.5191 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -3.8442 0.0291 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.8442 0.0291 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.3097 0.2370 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.3097 0.2370 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.7939 0.2426 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.7939 0.2426 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.1687 0.6174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1687 0.6174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6363 0.3706 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -3.6363 0.3706 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -4.8063 0.5708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8063 0.5708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1946 -0.4321 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1946 -0.4321 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0034 -0.2771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0034 -0.2771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0069 0.9458 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0069 0.9458 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9138 1.4739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9138 1.4739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8957 -3.1389 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8957 -3.1389 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6715 -3.7548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6715 -3.7548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4538 -3.0405 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4538 -3.0405 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8705 -3.5370 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8705 -3.5370 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4878 -3.4281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4878 -3.4281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7015 -2.8408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7015 -2.8408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3170 -2.7323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3170 -2.7323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7188 -3.2111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7188 -3.2111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5050 -3.7983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5050 -3.7983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8895 -3.9069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8895 -3.9069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3336 -3.1027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3336 -3.1027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9062 -4.2765 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9062 -4.2765 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4701 -0.0415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4701 -0.0415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4010 0.6227 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4010 0.6227 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9766 0.8022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9766 0.8022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5077 0.3566 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5077 0.3566 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0877 1.4322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0877 1.4322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5564 1.6028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5564 1.6028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6744 2.2723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6744 2.2723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2831 2.4938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2831 2.4938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3956 3.1318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3956 3.1318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8994 3.5482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8994 3.5482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2906 3.3266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2906 3.3266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1781 2.6887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1781 2.6887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0118 4.1859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0118 4.1859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7945 3.7429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7945 3.7429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8204 -1.6856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8204 -1.6856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 24 29 1 0 0 0 0 | + | 24 29 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 31 36 1 0 0 0 0 | + | 31 36 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 27 30 1 0 0 0 0 | + | 27 30 1 0 0 0 0 |
| − | 22 20 1 0 0 0 0 | + | 22 20 1 0 0 0 0 |
| − | 15 39 1 0 0 0 0 | + | 15 39 1 0 0 0 0 |
| − | 29 40 1 0 0 0 0 | + | 29 40 1 0 0 0 0 |
| − | 41 42 1 1 0 0 0 | + | 41 42 1 1 0 0 0 |
| − | 42 43 1 1 0 0 0 | + | 42 43 1 1 0 0 0 |
| − | 44 43 1 1 0 0 0 | + | 44 43 1 1 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 41 1 0 0 0 0 | + | 46 41 1 0 0 0 0 |
| − | 41 47 1 0 0 0 0 | + | 41 47 1 0 0 0 0 |
| − | 42 48 1 0 0 0 0 | + | 42 48 1 0 0 0 0 |
| − | 43 49 1 0 0 0 0 | + | 43 49 1 0 0 0 0 |
| − | 46 50 1 0 0 0 0 | + | 46 50 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 44 19 1 0 0 0 0 | + | 44 19 1 0 0 0 0 |
| − | 40 52 1 0 0 0 0 | + | 40 52 1 0 0 0 0 |
| − | 52 53 2 0 0 0 0 | + | 52 53 2 0 0 0 0 |
| − | 52 54 1 0 0 0 0 | + | 52 54 1 0 0 0 0 |
| − | 54 55 2 0 0 0 0 | + | 54 55 2 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 56 57 2 0 0 0 0 | + | 56 57 2 0 0 0 0 |
| − | 57 58 1 0 0 0 0 | + | 57 58 1 0 0 0 0 |
| − | 58 59 2 0 0 0 0 | + | 58 59 2 0 0 0 0 |
| − | 59 60 1 0 0 0 0 | + | 59 60 1 0 0 0 0 |
| − | 60 61 2 0 0 0 0 | + | 60 61 2 0 0 0 0 |
| − | 61 56 1 0 0 0 0 | + | 61 56 1 0 0 0 0 |
| − | 59 62 1 0 0 0 0 | + | 59 62 1 0 0 0 0 |
| − | 60 63 1 0 0 0 0 | + | 60 63 1 0 0 0 0 |
| − | 34 64 1 0 0 0 0 | + | 34 64 1 0 0 0 0 |
| − | 64 65 1 0 0 0 0 | + | 64 65 1 0 0 0 0 |
| − | 66 67 2 0 0 0 0 | + | 66 67 2 0 0 0 0 |
| − | 66 68 1 0 0 0 0 | + | 66 68 1 0 0 0 0 |
| − | 68 69 2 0 0 0 0 | + | 68 69 2 0 0 0 0 |
| − | 69 70 1 0 0 0 0 | + | 69 70 1 0 0 0 0 |
| − | 66 65 1 0 0 0 0 | + | 66 65 1 0 0 0 0 |
| − | 70 71 2 0 0 0 0 | + | 70 71 2 0 0 0 0 |
| − | 71 72 1 0 0 0 0 | + | 71 72 1 0 0 0 0 |
| − | 72 73 2 0 0 0 0 | + | 72 73 2 0 0 0 0 |
| − | 73 74 1 0 0 0 0 | + | 73 74 1 0 0 0 0 |
| − | 74 75 2 0 0 0 0 | + | 74 75 2 0 0 0 0 |
| − | 75 70 1 0 0 0 0 | + | 75 70 1 0 0 0 0 |
| − | 73 76 1 0 0 0 0 | + | 73 76 1 0 0 0 0 |
| − | 74 77 1 0 0 0 0 | + | 74 77 1 0 0 0 0 |
| − | 25 78 1 0 0 0 0 | + | 25 78 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 50 51 | + | M SAL 1 2 50 51 |
| − | M SBL 1 1 54 | + | M SBL 1 1 54 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 54 -4.1012 0.8727 | + | M SVB 1 54 -4.1012 0.8727 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AACGL0073 | + | ID FL7AACGL0073 |
| − | KNApSAcK_ID C00006849 | + | KNApSAcK_ID C00006849 |
| − | NAME Cyanidin 3-(6'',6'''-dicaffeylsophoroside)-5-glucoside | + | NAME Cyanidin 3-(6'',6'''-dicaffeylsophoroside)-5-glucoside |
| − | CAS_RN 170717-21-2,36570-76-0 | + | CAS_RN 170717-21-2,36570-76-0 |
| − | FORMULA C51H53O27 | + | FORMULA C51H53O27 |
| − | EXACTMASS 1097.27742149 | + | EXACTMASS 1097.27742149 |
| − | AVERAGEMASS 1097.95032 | + | AVERAGEMASS 1097.95032 |
| − | SMILES O=C(C=Cc(c8)cc(O)c(c8)O)OC[C@H]([C@H](O)1)O[C@@H](Oc(c6c(c7)ccc(c(O)7)O)cc(c([o+1]6)5)c(cc(O)c5)O[C@H](O4)[C@H]([C@@H](O)[C@H](C4CO)O)O)C(O[C@@H](O2)[C@H](O)[C@@H]([C@@H](C2COC(=O)C=Cc(c3)cc(O)c(c3)O)O)O)C(O)1 | + | SMILES O=C(C=Cc(c8)cc(O)c(c8)O)OC[C@H]([C@H](O)1)O[C@@H](Oc(c6c(c7)ccc(c(O)7)O)cc(c([o+1]6)5)c(cc(O)c5)O[C@H](O4)[C@H]([C@@H](O)[C@H](C4CO)O)O)C(O[C@@H](O2)[C@H](O)[C@@H]([C@@H](C2COC(=O)C=Cc(c3)cc(O)c(c3)O)O)O)C(O)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
78 85 0 0 0 0 0 0 0 0999 V2000
-2.6129 2.3989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6129 1.7566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0566 1.4354 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5003 1.7566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5003 2.3989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0566 2.7201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9440 1.4354 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3877 1.7566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3877 2.3989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9440 2.7201 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
0.1684 2.7200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7354 2.3927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3023 2.7200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3023 3.3747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7354 3.7020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1684 3.3747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1690 2.7200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8692 3.7019 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0566 0.7933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0284 1.0359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5069 -0.2782 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.0001 0.1094 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.2094 -0.4886 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0437 -1.1866 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.5069 -1.5046 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.3322 -0.8932 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.6816 0.1266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8337 -0.9371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0504 -2.2629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3514 0.2241 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
1.6549 -0.3678 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.2635 -0.2468 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.8143 -0.3378 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.4853 0.1322 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.9405 -0.0430 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0328 -0.2891 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4934 -0.8523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7675 -0.6401 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7354 4.3565 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4613 -2.6212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2154 0.5191 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-3.8442 0.0291 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.3097 0.2370 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.7939 0.2426 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.1687 0.6174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6363 0.3706 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-4.8063 0.5708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1946 -0.4321 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0034 -0.2771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0069 0.9458 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9138 1.4739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8957 -3.1389 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6715 -3.7548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4538 -3.0405 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8705 -3.5370 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4878 -3.4281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7015 -2.8408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3170 -2.7323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7188 -3.2111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5050 -3.7983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8895 -3.9069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3336 -3.1027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9062 -4.2765 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4701 -0.0415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4010 0.6227 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9766 0.8022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5077 0.3566 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0877 1.4322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5564 1.6028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6744 2.2723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2831 2.4938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3956 3.1318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8994 3.5482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2906 3.3266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1781 2.6887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0118 4.1859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7945 3.7429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8204 -1.6856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
24 29 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
31 36 1 0 0 0 0
32 37 1 0 0 0 0
33 38 1 0 0 0 0
27 30 1 0 0 0 0
22 20 1 0 0 0 0
15 39 1 0 0 0 0
29 40 1 0 0 0 0
41 42 1 1 0 0 0
42 43 1 1 0 0 0
44 43 1 1 0 0 0
44 45 1 0 0 0 0
45 46 1 0 0 0 0
46 41 1 0 0 0 0
41 47 1 0 0 0 0
42 48 1 0 0 0 0
43 49 1 0 0 0 0
46 50 1 0 0 0 0
50 51 1 0 0 0 0
44 19 1 0 0 0 0
40 52 1 0 0 0 0
52 53 2 0 0 0 0
52 54 1 0 0 0 0
54 55 2 0 0 0 0
55 56 1 0 0 0 0
56 57 2 0 0 0 0
57 58 1 0 0 0 0
58 59 2 0 0 0 0
59 60 1 0 0 0 0
60 61 2 0 0 0 0
61 56 1 0 0 0 0
59 62 1 0 0 0 0
60 63 1 0 0 0 0
34 64 1 0 0 0 0
64 65 1 0 0 0 0
66 67 2 0 0 0 0
66 68 1 0 0 0 0
68 69 2 0 0 0 0
69 70 1 0 0 0 0
66 65 1 0 0 0 0
70 71 2 0 0 0 0
71 72 1 0 0 0 0
72 73 2 0 0 0 0
73 74 1 0 0 0 0
74 75 2 0 0 0 0
75 70 1 0 0 0 0
73 76 1 0 0 0 0
74 77 1 0 0 0 0
25 78 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 50 51
M SBL 1 1 54
M SMT 1 CH2OH
M SVB 1 54 -4.1012 0.8727
S SKP 8
ID FL7AACGL0073
KNApSAcK_ID C00006849
NAME Cyanidin 3-(6'',6'''-dicaffeylsophoroside)-5-glucoside
CAS_RN 170717-21-2,36570-76-0
FORMULA C51H53O27
EXACTMASS 1097.27742149
AVERAGEMASS 1097.95032
SMILES O=C(C=Cc(c8)cc(O)c(c8)O)OC[C@H]([C@H](O)1)O[C@@H](Oc(c6c(c7)ccc(c(O)7)O)cc(c([o+1]6)5)c(cc(O)c5)O[C@H](O4)[C@H]([C@@H](O)[C@H](C4CO)O)O)C(O[C@@H](O2)[C@H](O)[C@@H]([C@@H](C2COC(=O)C=Cc(c3)cc(O)c(c3)O)O)O)C(O)1
M END