Mol:FL7AACGL0047
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 55 59 0 0 0 0 0 0 0 0999 V2000 | + | 55 59 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.4676 0.5785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4676 0.5785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4676 -0.0639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4676 -0.0639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9113 -0.3850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9113 -0.3850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3550 -0.0639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3550 -0.0639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3550 0.5785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3550 0.5785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9113 0.8997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9113 0.8997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7987 -0.3850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7987 -0.3850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2424 -0.0639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2424 -0.0639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2424 0.5785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2424 0.5785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7987 0.8997 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -0.7987 0.8997 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3137 0.8996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3137 0.8996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8807 0.5722 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8807 0.5722 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4476 0.8996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4476 0.8996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4476 1.5543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4476 1.5543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8807 1.8816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8807 1.8816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3137 1.5543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3137 1.5543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0237 0.8996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0237 0.8996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0144 1.8815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0144 1.8815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9113 -1.0272 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9113 -1.0272 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1737 -0.7846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1737 -0.7846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8807 2.5361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8807 2.5361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0701 -1.3014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0701 -1.3014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6989 -1.7913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6989 -1.7913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1644 -1.5835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1644 -1.5835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6487 -1.5779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6487 -1.5779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0234 -1.2030 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0234 -1.2030 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4910 -1.4498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4910 -1.4498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5839 -1.5980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5839 -1.5980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2775 -1.8195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2775 -1.8195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8582 -2.0976 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8582 -2.0976 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9559 -0.9477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9559 -0.9477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4342 -0.4432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4342 -0.4432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1334 -0.9643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1334 -0.9643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7119 -0.7989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7119 -0.7989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2939 -0.9643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2939 -0.9643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5948 -0.4432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5948 -0.4432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0162 -0.6085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0162 -0.6085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8755 -0.5929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8755 -0.5929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2544 -0.1960 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2544 -0.1960 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2981 -0.6317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2981 -0.6317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8454 -0.9643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8454 -0.9643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1900 -1.5610 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1900 -1.5610 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7612 -1.8908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7612 -1.8908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3506 -1.5505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3506 -1.5505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7612 -2.5361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7612 -2.5361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0187 -1.9286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0187 -1.9286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0187 -2.5162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0187 -2.5162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4340 -1.6246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4340 -1.6246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9559 -0.2464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9559 -0.2464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4490 0.0383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4490 0.0383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9068 -0.2260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9068 -0.2260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4490 0.6075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4490 0.6075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9420 0.8922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9420 0.8922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4340 0.6081 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4340 0.6081 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9420 1.4600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9420 1.4600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 27 31 1 0 0 0 0 | + | 27 31 1 0 0 0 0 |
| − | 25 19 1 0 0 0 0 | + | 25 19 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 37 39 1 0 0 0 0 | + | 37 39 1 0 0 0 0 |
| − | 36 40 1 0 0 0 0 | + | 36 40 1 0 0 0 0 |
| − | 35 41 1 0 0 0 0 | + | 35 41 1 0 0 0 0 |
| − | 33 20 1 0 0 0 0 | + | 33 20 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 43 45 2 0 0 0 0 | + | 43 45 2 0 0 0 0 |
| − | 44 46 1 0 0 0 0 | + | 44 46 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 46 48 2 0 0 0 0 | + | 46 48 2 0 0 0 0 |
| − | 31 49 1 0 0 0 0 | + | 31 49 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 51 2 0 0 0 0 | + | 50 51 2 0 0 0 0 |
| − | 50 52 1 0 0 0 0 | + | 50 52 1 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 53 55 2 0 0 0 0 | + | 53 55 2 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AACGL0047 | + | ID FL7AACGL0047 |
| − | KNApSAcK_ID C00006818 | + | KNApSAcK_ID C00006818 |
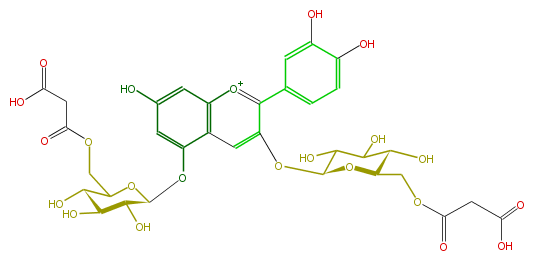
| − | NAME Cyanidin 3,5-di-(6-malonylglucoside) | + | NAME Cyanidin 3,5-di-(6-malonylglucoside) |
| − | CAS_RN - | + | CAS_RN - |
| − | FORMULA C33H35O22 | + | FORMULA C33H35O22 |
| − | EXACTMASS 783.1619978040001 | + | EXACTMASS 783.1619978040001 |
| − | AVERAGEMASS 783.6178 | + | AVERAGEMASS 783.6178 |
| − | SMILES c(c5)(O)c(cc(c5)c(c(OC(C4O)OC(COC(=O)CC(O)=O)C(C(O)4)O)1)[o+1]c(c2)c(c(OC(O3)C(O)C(O)C(O)C3COC(CC(O)=O)=O)cc(O)2)c1)O | + | SMILES c(c5)(O)c(cc(c5)c(c(OC(C4O)OC(COC(=O)CC(O)=O)C(C(O)4)O)1)[o+1]c(c2)c(c(OC(O3)C(O)C(O)C(O)C3COC(CC(O)=O)=O)cc(O)2)c1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
55 59 0 0 0 0 0 0 0 0999 V2000
-2.4676 0.5785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4676 -0.0639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9113 -0.3850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3550 -0.0639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3550 0.5785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9113 0.8997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7987 -0.3850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2424 -0.0639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2424 0.5785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7987 0.8997 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
0.3137 0.8996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8807 0.5722 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4476 0.8996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4476 1.5543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8807 1.8816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3137 1.5543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0237 0.8996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0144 1.8815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9113 -1.0272 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1737 -0.7846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8807 2.5361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0701 -1.3014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6989 -1.7913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1644 -1.5835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6487 -1.5779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0234 -1.2030 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4910 -1.4498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5839 -1.5980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2775 -1.8195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8582 -2.0976 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9559 -0.9477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4342 -0.4432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1334 -0.9643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7119 -0.7989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2939 -0.9643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5948 -0.4432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0162 -0.6085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8755 -0.5929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2544 -0.1960 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2981 -0.6317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8454 -0.9643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1900 -1.5610 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7612 -1.8908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3506 -1.5505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7612 -2.5361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0187 -1.9286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0187 -2.5162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.4340 -1.6246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9559 -0.2464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4490 0.0383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9068 -0.2260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4490 0.6075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9420 0.8922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.4340 0.6081 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9420 1.4600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
15 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
27 31 1 0 0 0 0
25 19 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
32 38 1 0 0 0 0
37 39 1 0 0 0 0
36 40 1 0 0 0 0
35 41 1 0 0 0 0
33 20 1 0 0 0 0
41 42 1 0 0 0 0
42 43 1 0 0 0 0
43 44 1 0 0 0 0
43 45 2 0 0 0 0
44 46 1 0 0 0 0
46 47 1 0 0 0 0
46 48 2 0 0 0 0
31 49 1 0 0 0 0
49 50 1 0 0 0 0
50 51 2 0 0 0 0
50 52 1 0 0 0 0
52 53 1 0 0 0 0
53 54 1 0 0 0 0
53 55 2 0 0 0 0
S SKP 8
ID FL7AACGL0047
KNApSAcK_ID C00006818
NAME Cyanidin 3,5-di-(6-malonylglucoside)
CAS_RN -
FORMULA C33H35O22
EXACTMASS 783.1619978040001
AVERAGEMASS 783.6178
SMILES c(c5)(O)c(cc(c5)c(c(OC(C4O)OC(COC(=O)CC(O)=O)C(C(O)4)O)1)[o+1]c(c2)c(c(OC(O3)C(O)C(O)C(O)C3COC(CC(O)=O)=O)cc(O)2)c1)O
M END