Mol:FL7AACGL0036
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 58 63 0 0 0 0 0 0 0 0999 V2000 | + | 58 63 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.3092 -2.1703 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3092 -2.1703 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4667 -1.8702 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.4667 -1.8702 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.1659 -2.3912 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.1659 -2.3912 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.7443 -2.2259 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7443 -2.2259 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3263 -2.3912 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.3263 -2.3912 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.6272 -1.8702 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.6272 -1.8702 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.0487 -2.0355 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 3.0487 -2.0355 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.9080 -2.0199 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9080 -2.0199 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0902 -1.5609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0902 -1.5609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0612 -2.1208 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0612 -2.1208 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1038 -4.2358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1038 -4.2358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4711 -4.1242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4711 -4.1242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2514 -3.5206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2514 -3.5206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6643 -3.0285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6643 -3.0285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2970 -3.1401 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2970 -3.1401 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5167 -3.7437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5167 -3.7437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4446 -2.4249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4446 -2.4249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8576 -1.9329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8576 -1.9329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4902 -2.0445 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4902 -2.0445 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7099 -2.6480 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -2.7099 -2.6480 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9030 -1.5526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9030 -1.5526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6791 -0.9374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6791 -0.9374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0999 -0.4359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0999 -0.4359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7446 -0.5496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7446 -0.5496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9686 -1.1647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9686 -1.1647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5477 -1.6663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5477 -1.6663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3234 -4.8392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3234 -4.8392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3992 -0.5496 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3992 -0.5496 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6192 -3.4090 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6192 -3.4090 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2201 -1.3980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2201 -1.3980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0496 -0.7152 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -0.0496 -0.7152 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.3504 -1.2362 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.3504 -1.2362 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.2281 -1.0709 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2281 -1.0709 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8101 -1.2362 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.8101 -1.2362 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.1109 -0.7152 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.1109 -0.7152 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.5324 -0.8805 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.5324 -0.8805 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.6083 -0.8649 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6083 -0.8649 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5739 -0.4059 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5739 -0.4059 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3616 -1.2362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3616 -1.2362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6131 -1.2784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6131 -1.2784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9528 1.6094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9528 1.6094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3771 0.7039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3771 0.7039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9609 0.6516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9609 0.6516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2400 1.0518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2400 1.0518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9963 1.5763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9963 1.5763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3262 2.0493 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3262 2.0493 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8998 1.9980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8998 1.9980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1435 1.4736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1435 1.4736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8136 1.0004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8136 1.0004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2292 2.4703 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2292 2.4703 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0456 2.0302 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0456 2.0302 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0172 -0.2926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0172 -0.2926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7161 1.4223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7161 1.4223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0648 0.6746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0648 0.6746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4968 -2.7126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4968 -2.7126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4968 -3.5377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4968 -3.5377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3893 2.7615 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3893 2.7615 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4775 3.7576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4775 3.7576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 1 1 0 0 0 | + | 3 4 1 1 0 0 0 |
| − | 4 5 1 1 0 0 0 | + | 4 5 1 1 0 0 0 |
| − | 6 5 1 1 0 0 0 | + | 6 5 1 1 0 0 0 |
| − | 6 7 1 0 0 0 0 | + | 6 7 1 0 0 0 0 |
| − | 7 2 1 0 0 0 0 | + | 7 2 1 0 0 0 0 |
| − | 2 8 1 0 0 0 0 | + | 2 8 1 0 0 0 0 |
| − | 7 9 1 0 0 0 0 | + | 7 9 1 0 0 0 0 |
| − | 6 10 1 0 0 0 0 | + | 6 10 1 0 0 0 0 |
| − | 3 1 1 0 0 0 0 | + | 3 1 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 11 2 0 0 0 0 | + | 16 11 2 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 19 20 2 0 0 0 0 | + | 19 20 2 0 0 0 0 |
| − | 20 15 1 0 0 0 0 | + | 20 15 1 0 0 0 0 |
| − | 19 21 1 0 0 0 0 | + | 19 21 1 0 0 0 0 |
| − | 21 22 2 0 0 0 0 | + | 21 22 2 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 2 0 0 0 0 | + | 23 24 2 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 2 0 0 0 0 | + | 25 26 2 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 11 27 1 0 0 0 0 | + | 11 27 1 0 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 13 29 1 0 0 0 0 | + | 13 29 1 0 0 0 0 |
| − | 30 18 1 0 0 0 0 | + | 30 18 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 32 30 1 0 0 0 0 | + | 32 30 1 0 0 0 0 |
| − | 39 1 1 0 0 0 0 | + | 39 1 1 0 0 0 0 |
| − | 25 40 1 0 0 0 0 | + | 25 40 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 42 43 2 0 0 0 0 | + | 42 43 2 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 2 0 0 0 0 | + | 44 45 2 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 2 0 0 0 0 | + | 46 47 2 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 2 0 0 0 0 | + | 48 49 2 0 0 0 0 |
| − | 49 44 1 0 0 0 0 | + | 49 44 1 0 0 0 0 |
| − | 47 50 1 0 0 0 0 | + | 47 50 1 0 0 0 0 |
| − | 41 51 2 0 0 0 0 | + | 41 51 2 0 0 0 0 |
| − | 41 52 1 0 0 0 0 | + | 41 52 1 0 0 0 0 |
| − | 52 35 1 0 0 0 0 | + | 52 35 1 0 0 0 0 |
| − | 48 53 1 0 0 0 0 | + | 48 53 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 5 55 1 0 0 0 0 | + | 5 55 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 46 57 1 0 0 0 0 | + | 46 57 1 0 0 0 0 |
| − | 57 58 1 0 0 0 0 | + | 57 58 1 0 0 0 0 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 55 56 | + | M SAL 3 2 55 56 |
| − | M SBL 3 1 60 | + | M SBL 3 1 60 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SVB 3 60 3.6899 -2.4043 | + | M SVB 3 60 3.6899 -2.4043 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 57 58 | + | M SAL 2 2 57 58 |
| − | M SBL 2 1 62 | + | M SBL 2 1 62 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 62 1.4831 2.5735 | + | M SVB 2 62 1.4831 2.5735 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 53 54 | + | M SAL 1 2 53 54 |
| − | M SBL 1 1 58 | + | M SBL 1 1 58 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 58 3.3381 2.2376 | + | M SVB 1 58 3.3381 2.2376 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AACGL0036 | + | ID FL7AACGL0036 |
| − | KNApSAcK_ID C00006806 | + | KNApSAcK_ID C00006806 |
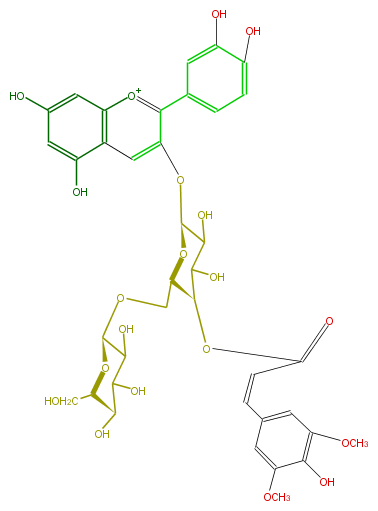
| − | NAME Alatanin 1 | + | NAME Alatanin 1 |
| − | CAS_RN 131189-50-9 | + | CAS_RN 131189-50-9 |
| − | FORMULA C38H41O20 | + | FORMULA C38H41O20 |
| − | EXACTMASS 817.219118752 | + | EXACTMASS 817.219118752 |
| − | AVERAGEMASS 817.72014 | + | AVERAGEMASS 817.72014 |
| − | SMILES c(c6)c(cc(O)c6O)c([o+1]5)c(cc(c54)c(O)cc(O)c4)O[C@@H](C3O)O[C@@H]([C@@H](C(O)3)OC(=O)C=Cc(c2)cc(c(c(OC)2)O)OC)CO[C@H](O1)C(O)C([C@@H](O)[C@@H](CO)1)O | + | SMILES c(c6)c(cc(O)c6O)c([o+1]5)c(cc(c54)c(O)cc(O)c4)O[C@@H](C3O)O[C@@H]([C@@H](C(O)3)OC(=O)C=Cc(c2)cc(c(c(OC)2)O)OC)CO[C@H](O1)C(O)C([C@@H](O)[C@@H](CO)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
58 63 0 0 0 0 0 0 0 0999 V2000
1.3092 -2.1703 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4667 -1.8702 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.1659 -2.3912 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.7443 -2.2259 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3263 -2.3912 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.6272 -1.8702 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.0487 -2.0355 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.9080 -2.0199 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0902 -1.5609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0612 -2.1208 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1038 -4.2358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4711 -4.1242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2514 -3.5206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6643 -3.0285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2970 -3.1401 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5167 -3.7437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4446 -2.4249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8576 -1.9329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4902 -2.0445 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7099 -2.6480 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
-2.9030 -1.5526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6791 -0.9374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0999 -0.4359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7446 -0.5496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9686 -1.1647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5477 -1.6663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3234 -4.8392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3992 -0.5496 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6192 -3.4090 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2201 -1.3980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0496 -0.7152 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.3504 -1.2362 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.2281 -1.0709 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8101 -1.2362 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.1109 -0.7152 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.5324 -0.8805 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.6083 -0.8649 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5739 -0.4059 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3616 -1.2362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6131 -1.2784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9528 1.6094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3771 0.7039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9609 0.6516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2400 1.0518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9963 1.5763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3262 2.0493 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8998 1.9980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1435 1.4736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8136 1.0004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2292 2.4703 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0456 2.0302 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0172 -0.2926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7161 1.4223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0648 0.6746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4968 -2.7126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4968 -3.5377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3893 2.7615 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4775 3.7576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2 3 1 0 0 0 0
3 4 1 1 0 0 0
4 5 1 1 0 0 0
6 5 1 1 0 0 0
6 7 1 0 0 0 0
7 2 1 0 0 0 0
2 8 1 0 0 0 0
7 9 1 0 0 0 0
6 10 1 0 0 0 0
3 1 1 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 11 2 0 0 0 0
14 17 1 0 0 0 0
17 18 2 0 0 0 0
18 19 1 0 0 0 0
19 20 2 0 0 0 0
20 15 1 0 0 0 0
19 21 1 0 0 0 0
21 22 2 0 0 0 0
22 23 1 0 0 0 0
23 24 2 0 0 0 0
24 25 1 0 0 0 0
25 26 2 0 0 0 0
26 21 1 0 0 0 0
11 27 1 0 0 0 0
24 28 1 0 0 0 0
13 29 1 0 0 0 0
30 18 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
36 38 1 0 0 0 0
34 39 1 0 0 0 0
32 30 1 0 0 0 0
39 1 1 0 0 0 0
25 40 1 0 0 0 0
41 42 1 0 0 0 0
42 43 2 0 0 0 0
43 44 1 0 0 0 0
44 45 2 0 0 0 0
45 46 1 0 0 0 0
46 47 2 0 0 0 0
47 48 1 0 0 0 0
48 49 2 0 0 0 0
49 44 1 0 0 0 0
47 50 1 0 0 0 0
41 51 2 0 0 0 0
41 52 1 0 0 0 0
52 35 1 0 0 0 0
48 53 1 0 0 0 0
53 54 1 0 0 0 0
5 55 1 0 0 0 0
55 56 1 0 0 0 0
46 57 1 0 0 0 0
57 58 1 0 0 0 0
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 55 56
M SBL 3 1 60
M SMT 3 CH2OH
M SVB 3 60 3.6899 -2.4043
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 57 58
M SBL 2 1 62
M SMT 2 OCH3
M SVB 2 62 1.4831 2.5735
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 53 54
M SBL 1 1 58
M SMT 1 OCH3
M SVB 1 58 3.3381 2.2376
S SKP 8
ID FL7AACGL0036
KNApSAcK_ID C00006806
NAME Alatanin 1
CAS_RN 131189-50-9
FORMULA C38H41O20
EXACTMASS 817.219118752
AVERAGEMASS 817.72014
SMILES c(c6)c(cc(O)c6O)c([o+1]5)c(cc(c54)c(O)cc(O)c4)O[C@@H](C3O)O[C@@H]([C@@H](C(O)3)OC(=O)C=Cc(c2)cc(c(c(OC)2)O)OC)CO[C@H](O1)C(O)C([C@@H](O)[C@@H](CO)1)O
M END