Mol:FL7AACGA0014
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.5717 1.9366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5717 1.9366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5717 1.1116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5717 1.1116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8572 0.6991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8572 0.6991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1428 1.1116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1428 1.1116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1428 1.9366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1428 1.9366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8572 2.3491 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8572 2.3491 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4283 0.6991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4283 0.6991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2862 1.1116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2862 1.1116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2862 1.9366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2862 1.9366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4283 2.3491 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -0.4283 2.3491 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0627 2.3849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0627 2.3849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7772 1.9724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7772 1.9724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4916 2.3849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4916 2.3849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4916 3.2099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4916 3.2099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7772 3.6224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7772 3.6224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0627 3.2099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0627 3.2099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1318 3.5795 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1318 3.5795 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9303 0.7397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9303 0.7397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1517 2.2714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1517 2.2714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8572 0.1367 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8572 0.1367 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1517 2.0038 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1517 2.0038 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9530 -1.0439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9530 -1.0439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1638 -1.2850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1638 -1.2850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2773 -0.5875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2773 -0.5875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0122 -0.2121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0122 -0.2121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2230 0.0291 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2230 0.0291 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2182 -0.6684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2182 -0.6684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7326 -0.3471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7326 -0.3471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9768 -1.7268 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9768 -1.7268 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6686 -1.4016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6686 -1.4016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5884 -1.1724 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5884 -1.1724 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3012 -0.5037 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3012 -0.5037 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9135 -2.9609 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9135 -2.9609 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1318 -3.2254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1318 -3.2254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3299 -2.5414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3299 -2.5414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0757 -2.1881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0757 -2.1881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2940 -1.9234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2940 -1.9234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1678 -2.6075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1678 -2.6075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7088 -2.2466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7088 -2.2466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2524 -2.3553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2524 -2.3553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4041 -2.9292 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4041 -2.9292 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9349 -3.6224 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9349 -3.6224 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8754 -2.9051 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8754 -2.9051 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 8 18 1 0 0 0 0 | + | 8 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 13 21 1 0 0 0 0 | + | 13 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 25 18 1 0 0 0 0 | + | 25 18 1 0 0 0 0 |
| − | 32 28 1 0 0 0 0 | + | 32 28 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 33 41 1 0 0 0 0 | + | 33 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 35 43 1 0 0 0 0 | + | 35 43 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AACGA0014 | + | ID FL7AACGA0014 |
| − | KNApSAcK_ID C00014758 | + | KNApSAcK_ID C00014758 |
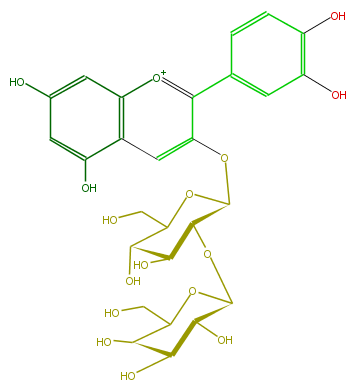
| − | NAME Cyanidin 3-(2-glucosylgalactoside) | + | NAME Cyanidin 3-(2-glucosylgalactoside) |
| − | CAS_RN 220726-09-0 | + | CAS_RN 220726-09-0 |
| − | FORMULA C27H31O16 | + | FORMULA C27H31O16 |
| − | EXACTMASS 611.161209944 | + | EXACTMASS 611.161209944 |
| − | AVERAGEMASS 611.52544 | + | AVERAGEMASS 611.52544 |
| − | SMILES Oc(c(O)1)ccc(c(c(OC(O4)C(OC(C5O)OC(C(C5O)O)CO)C(C(C4CO)O)O)3)[o+1]c(c2c3)cc(O)cc2O)c1 | + | SMILES Oc(c(O)1)ccc(c(c(OC(O4)C(OC(C5O)OC(C(C5O)O)CO)C(C(C4CO)O)O)3)[o+1]c(c2c3)cc(O)cc2O)c1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-2.5717 1.9366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5717 1.1116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8572 0.6991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1428 1.1116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1428 1.9366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8572 2.3491 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4283 0.6991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2862 1.1116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2862 1.9366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4283 2.3491 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
1.0627 2.3849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7772 1.9724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4916 2.3849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4916 3.2099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7772 3.6224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0627 3.2099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1318 3.5795 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9303 0.7397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1517 2.2714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8572 0.1367 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1517 2.0038 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9530 -1.0439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1638 -1.2850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2773 -0.5875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0122 -0.2121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2230 0.0291 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2182 -0.6684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7326 -0.3471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9768 -1.7268 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6686 -1.4016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5884 -1.1724 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3012 -0.5037 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9135 -2.9609 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1318 -3.2254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3299 -2.5414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0757 -2.1881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2940 -1.9234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1678 -2.6075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7088 -2.2466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2524 -2.3553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4041 -2.9292 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9349 -3.6224 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8754 -2.9051 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
14 17 1 0 0 0 0
8 18 1 0 0 0 0
1 19 1 0 0 0 0
3 20 1 0 0 0 0
13 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
27 28 1 0 0 0 0
22 29 1 0 0 0 0
23 30 1 0 0 0 0
24 31 1 0 0 0 0
25 18 1 0 0 0 0
32 28 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
38 39 1 0 0 0 0
39 40 1 0 0 0 0
33 41 1 0 0 0 0
34 42 1 0 0 0 0
35 43 1 0 0 0 0
36 31 1 0 0 0 0
S SKP 8
ID FL7AACGA0014
KNApSAcK_ID C00014758
NAME Cyanidin 3-(2-glucosylgalactoside)
CAS_RN 220726-09-0
FORMULA C27H31O16
EXACTMASS 611.161209944
AVERAGEMASS 611.52544
SMILES Oc(c(O)1)ccc(c(c(OC(O4)C(OC(C5O)OC(C(C5O)O)CO)C(C(C4CO)O)O)3)[o+1]c(c2c3)cc(O)cc2O)c1
M END