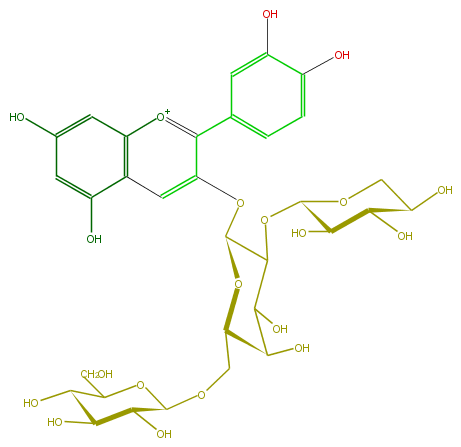
Mol:FL7AACGA0005
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 52 57 0 0 0 0 0 0 0 0999 V2000 | + | 52 57 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.4742 1.7278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4742 1.7278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4742 0.9182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4742 0.9182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7729 0.5133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7729 0.5133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0717 0.9182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0717 0.9182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0717 1.7278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0717 1.7278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7729 2.1327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7729 2.1327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3705 0.5133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3705 0.5133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6693 0.9182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6693 0.9182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6693 1.7278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6693 1.7278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3705 2.1327 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -1.3705 2.1327 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0317 2.1325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0317 2.1325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7464 1.7200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7464 1.7200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4611 2.1325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4611 2.1325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4611 2.9578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4611 2.9578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7464 3.3704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7464 3.3704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0317 2.9578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0317 2.9578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7729 -0.2961 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7729 -0.2961 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7464 4.1954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7464 4.1954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2255 0.4146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2255 0.4146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7621 -0.7602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7621 -0.7602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0842 -0.2716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0842 -0.2716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1843 -1.2112 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1843 -1.2112 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0842 -2.1566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0842 -2.1566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7621 -2.6453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7621 -2.6453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4937 -1.7056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4937 -1.7056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7114 0.0786 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7114 0.0786 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9396 -2.0953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9396 -2.0953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3779 -2.4844 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3779 -2.4844 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0098 -2.9229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0098 -2.9229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1751 2.1325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1751 2.1325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4111 0.4329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4111 0.4329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0233 -0.1793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0233 -0.1793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7755 0.2495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7755 0.2495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6412 0.2495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6412 0.2495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0291 0.8617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0291 0.8617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2768 0.4329 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2768 0.4329 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4679 -0.1807 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4679 -0.1807 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2353 0.6695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2353 0.6695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4475 -0.2427 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4475 -0.2427 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1876 -3.3589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1876 -3.3589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6853 -4.0220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6853 -4.0220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9617 -3.7407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9617 -3.7407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2637 -3.7331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2637 -3.7331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7709 -3.2258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7709 -3.2258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5099 -3.4911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5099 -3.4911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8927 -3.5938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8927 -3.5938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4248 -3.9685 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4248 -3.9685 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5467 -3.4203 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5467 -3.4203 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3768 -4.1954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3768 -4.1954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1756 3.3702 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1756 3.3702 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0063 -3.0037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0063 -3.0037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2353 -2.7235 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2353 -2.7235 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 3 17 1 0 0 0 0 | + | 3 17 1 0 0 0 0 |
| − | 15 18 1 0 0 0 0 | + | 15 18 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 25 27 1 0 0 0 0 | + | 25 27 1 0 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 21 19 1 0 0 0 0 | + | 21 19 1 0 0 0 0 |
| − | 19 8 1 0 0 0 0 | + | 19 8 1 0 0 0 0 |
| − | 1 30 1 0 0 0 0 | + | 1 30 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 34 38 1 0 0 0 0 | + | 34 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 31 26 1 0 0 0 0 | + | 31 26 1 0 0 0 0 |
| − | 40 41 1 1 0 0 0 | + | 40 41 1 1 0 0 0 |
| − | 41 42 1 1 0 0 0 | + | 41 42 1 1 0 0 0 |
| − | 43 42 1 1 0 0 0 | + | 43 42 1 1 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 40 1 0 0 0 0 | + | 45 40 1 0 0 0 0 |
| − | 40 46 1 0 0 0 0 | + | 40 46 1 0 0 0 0 |
| − | 41 47 1 0 0 0 0 | + | 41 47 1 0 0 0 0 |
| − | 29 48 1 0 0 0 0 | + | 29 48 1 0 0 0 0 |
| − | 48 43 1 0 0 0 0 | + | 48 43 1 0 0 0 0 |
| − | 42 49 1 0 0 0 0 | + | 42 49 1 0 0 0 0 |
| − | 14 50 1 0 0 0 0 | + | 14 50 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 45 51 1 0 0 0 0 | + | 45 51 1 0 0 0 0 |
| − | M CHG 1 10 1 | + | M CHG 1 10 1 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 51 52 | + | M SAL 1 2 51 52 |
| − | M SBL 1 1 57 | + | M SBL 1 1 57 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 57 0.4964 -0.4873 | + | M SBV 1 57 0.4964 -0.4873 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL7AACGA0005 | + | ID FL7AACGA0005 |
| − | FORMULA C32H39O20 | + | FORMULA C32H39O20 |
| − | EXACTMASS 743.2034686879999 | + | EXACTMASS 743.2034686879999 |
| − | AVERAGEMASS 743.64006 | + | AVERAGEMASS 743.64006 |
| − | SMILES C(C(C6O)OC(C(O)C6O)OCC(C4O)OC(C(OC(O5)C(C(C(C5)O)O)O)C4O)Oc(c3)c([o+1]c(c32)cc(cc2O)O)c(c1)cc(O)c(c1)O)O | + | SMILES C(C(C6O)OC(C(O)C6O)OCC(C4O)OC(C(OC(O5)C(C(C(C5)O)O)O)C4O)Oc(c3)c([o+1]c(c32)cc(cc2O)O)c(c1)cc(O)c(c1)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
52 57 0 0 0 0 0 0 0 0999 V2000
-3.4742 1.7278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4742 0.9182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7729 0.5133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0717 0.9182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0717 1.7278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7729 2.1327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3705 0.5133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6693 0.9182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6693 1.7278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3705 2.1327 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
0.0317 2.1325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7464 1.7200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4611 2.1325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4611 2.9578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7464 3.3704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0317 2.9578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7729 -0.2961 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7464 4.1954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2255 0.4146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7621 -0.7602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0842 -0.2716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1843 -1.2112 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0842 -2.1566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7621 -2.6453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4937 -1.7056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7114 0.0786 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9396 -2.0953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3779 -2.4844 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0098 -2.9229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1751 2.1325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4111 0.4329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0233 -0.1793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7755 0.2495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6412 0.2495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0291 0.8617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2768 0.4329 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4679 -0.1807 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2353 0.6695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4475 -0.2427 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1876 -3.3589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6853 -4.0220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9617 -3.7407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2637 -3.7331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7709 -3.2258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5099 -3.4911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8927 -3.5938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4248 -3.9685 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5467 -3.4203 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3768 -4.1954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1756 3.3702 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0063 -3.0037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2353 -2.7235 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
3 17 1 0 0 0 0
15 18 1 0 0 0 0
20 21 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
25 27 1 0 0 0 0
24 28 1 0 0 0 0
23 29 1 0 0 0 0
21 19 1 0 0 0 0
19 8 1 0 0 0 0
1 30 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
32 37 1 0 0 0 0
34 38 1 0 0 0 0
33 39 1 0 0 0 0
31 26 1 0 0 0 0
40 41 1 1 0 0 0
41 42 1 1 0 0 0
43 42 1 1 0 0 0
43 44 1 0 0 0 0
44 45 1 0 0 0 0
45 40 1 0 0 0 0
40 46 1 0 0 0 0
41 47 1 0 0 0 0
29 48 1 0 0 0 0
48 43 1 0 0 0 0
42 49 1 0 0 0 0
14 50 1 0 0 0 0
51 52 1 0 0 0 0
45 51 1 0 0 0 0
M CHG 1 10 1
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 51 52
M SBL 1 1 57
M SMT 1 ^ CH2OH
M SBV 1 57 0.4964 -0.4873
S SKP 5
ID FL7AACGA0005
FORMULA C32H39O20
EXACTMASS 743.2034686879999
AVERAGEMASS 743.64006
SMILES C(C(C6O)OC(C(O)C6O)OCC(C4O)OC(C(OC(O5)C(C(C(C5)O)O)O)C4O)Oc(c3)c([o+1]c(c32)cc(cc2O)O)c(c1)cc(O)c(c1)O)O
M END