Mol:FL7AAAGL0026
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 49 53 0 0 0 0 0 0 0 0999 V2000 | + | 49 53 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.8756 0.9284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8756 0.9284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8756 0.2861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8756 0.2861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3193 -0.0351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3193 -0.0351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7630 0.2861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7630 0.2861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7630 0.9284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7630 0.9284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3193 1.2496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3193 1.2496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2067 -0.0351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2067 -0.0351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6504 0.2861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6504 0.2861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6504 0.9284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6504 0.9284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2067 1.2496 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -1.2067 1.2496 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0943 1.2495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0943 1.2495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4727 0.9221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4727 0.9221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0396 1.2495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0396 1.2495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0396 1.9042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0396 1.9042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4727 2.2315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4727 2.2315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0943 1.9042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0943 1.9042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4317 1.2495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4317 1.2495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6065 2.2314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6065 2.2314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3193 -0.6772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3193 -0.6772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2343 -0.4346 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2343 -0.4346 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9231 -0.1346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9231 -0.1346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6223 -0.6556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6223 -0.6556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2008 -0.4903 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2008 -0.4903 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7828 -0.6556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7828 -0.6556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0836 -0.1346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0836 -0.1346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5051 -0.2998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5051 -0.2998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3644 -0.2843 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3644 -0.2843 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7433 0.1127 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7433 0.1127 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5177 -0.3852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5177 -0.3852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3343 -0.6556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3343 -0.6556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6431 -0.8483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6431 -0.8483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2719 -1.3383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2719 -1.3383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7374 -1.1304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7374 -1.1304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1264 -1.2784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1264 -1.2784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5964 -0.7500 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5964 -0.7500 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0640 -0.9968 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0640 -0.9968 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1569 -1.1450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1569 -1.1450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8505 -1.3664 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8505 -1.3664 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4311 -1.6445 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4311 -1.6445 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3204 -1.3479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3204 -1.3479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8630 -1.6612 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8630 -1.6612 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4347 -1.3312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4347 -1.3312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0514 -1.6873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0514 -1.6873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6044 -1.3680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6044 -1.3680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1569 -1.6870 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1569 -1.6870 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6044 -0.7300 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6044 -0.7300 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8630 -2.2315 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8630 -2.2315 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4625 -0.2126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4625 -0.2126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6656 -0.4261 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6656 -0.4261 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 22 20 1 0 0 0 0 | + | 22 20 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 19 1 0 0 0 0 | + | 34 19 1 0 0 0 0 |
| − | 30 40 1 0 0 0 0 | + | 30 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 2 0 0 0 0 | + | 44 45 2 0 0 0 0 |
| − | 44 46 1 0 0 0 0 | + | 44 46 1 0 0 0 0 |
| − | 41 47 2 0 0 0 0 | + | 41 47 2 0 0 0 0 |
| − | 36 48 1 0 0 0 0 | + | 36 48 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 48 49 | + | M SAL 1 2 48 49 |
| − | M SBL 1 1 52 | + | M SBL 1 1 52 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 52 -8.4674 6.4976 | + | M SBV 1 52 -8.4674 6.4976 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AAAGL0026 | + | ID FL7AAAGL0026 |
| − | KNApSAcK_ID C00006765 | + | KNApSAcK_ID C00006765 |
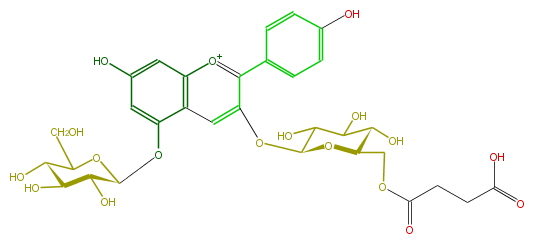
| − | NAME Pelargonidin 3-(6''-succinylglucoside)-5-glucoside | + | NAME Pelargonidin 3-(6''-succinylglucoside)-5-glucoside |
| − | CAS_RN - | + | CAS_RN - |
| − | FORMULA C31H35O18 | + | FORMULA C31H35O18 |
| − | EXACTMASS 695.182339316 | + | EXACTMASS 695.182339316 |
| − | AVERAGEMASS 695.5988 | + | AVERAGEMASS 695.5988 |
| − | SMILES c(c23)c(c(c(c5)ccc(O)c5)[o+1]c2cc(O)cc3OC(O4)C(C(O)C(O)C4CO)O)OC(C1O)OC(C(O)C(O)1)COC(CCC(O)=O)=O | + | SMILES c(c23)c(c(c(c5)ccc(O)c5)[o+1]c2cc(O)cc3OC(O4)C(C(O)C(O)C4CO)O)OC(C1O)OC(C(O)C(O)1)COC(CCC(O)=O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
49 53 0 0 0 0 0 0 0 0999 V2000
-2.8756 0.9284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8756 0.2861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3193 -0.0351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7630 0.2861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7630 0.9284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3193 1.2496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2067 -0.0351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6504 0.2861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6504 0.9284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2067 1.2496 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
-0.0943 1.2495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4727 0.9221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0396 1.2495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0396 1.9042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4727 2.2315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0943 1.9042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4317 1.2495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6065 2.2314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3193 -0.6772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2343 -0.4346 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9231 -0.1346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6223 -0.6556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2008 -0.4903 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7828 -0.6556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0836 -0.1346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5051 -0.2998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3644 -0.2843 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7433 0.1127 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5177 -0.3852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3343 -0.6556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6431 -0.8483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2719 -1.3383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7374 -1.1304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1264 -1.2784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5964 -0.7500 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0640 -0.9968 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1569 -1.1450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8505 -1.3664 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4311 -1.6445 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3204 -1.3479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8630 -1.6612 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4347 -1.3312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0514 -1.6873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6044 -1.3680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1569 -1.6870 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6044 -0.7300 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8630 -2.2315 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4625 -0.2126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6656 -0.4261 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
24 30 1 0 0 0 0
22 20 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 19 1 0 0 0 0
30 40 1 0 0 0 0
40 41 1 0 0 0 0
41 42 1 0 0 0 0
42 43 1 0 0 0 0
43 44 1 0 0 0 0
44 45 2 0 0 0 0
44 46 1 0 0 0 0
41 47 2 0 0 0 0
36 48 1 0 0 0 0
48 49 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 48 49
M SBL 1 1 52
M SMT 1 ^CH2OH
M SBV 1 52 -8.4674 6.4976
S SKP 8
ID FL7AAAGL0026
KNApSAcK_ID C00006765
NAME Pelargonidin 3-(6''-succinylglucoside)-5-glucoside
CAS_RN -
FORMULA C31H35O18
EXACTMASS 695.182339316
AVERAGEMASS 695.5988
SMILES c(c23)c(c(c(c5)ccc(O)c5)[o+1]c2cc(O)cc3OC(O4)C(C(O)C(O)C4CO)O)OC(C1O)OC(C(O)C(O)1)COC(CCC(O)=O)=O
M END