Mol:FL7AAAGL0022
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 45 49 0 0 0 0 0 0 0 0999 V2000 | + | 45 49 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.1490 0.8335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1490 0.8335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1490 0.1911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1490 0.1911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5927 -0.1301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5927 -0.1301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0364 0.1911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0364 0.1911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0364 0.8335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0364 0.8335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5927 1.1546 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5927 1.1546 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4801 -0.1301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4801 -0.1301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0762 0.1911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0762 0.1911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0762 0.8335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0762 0.8335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4801 1.1546 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -0.4801 1.1546 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6323 1.1545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6323 1.1545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1993 0.8272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1993 0.8272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7663 1.1545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7663 1.1545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7663 1.8092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7663 1.8092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1993 2.1365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1993 2.1365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6323 1.8092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6323 1.8092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7051 1.1545 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7051 1.1545 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3331 2.1365 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3331 2.1365 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5927 -0.7722 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5927 -0.7722 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4923 -0.5296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4923 -0.5296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6497 -0.2295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6497 -0.2295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3489 -0.7506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3489 -0.7506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9274 -0.5852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9274 -0.5852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5094 -0.7506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5094 -0.7506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8103 -0.2295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8103 -0.2295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2317 -0.3948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2317 -0.3948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0910 -0.3792 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0910 -0.3792 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4699 0.0177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4699 0.0177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2443 -0.4801 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2443 -0.4801 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0609 -0.7506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0609 -0.7506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9165 -0.9433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9165 -0.9433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5453 -1.4332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5453 -1.4332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0108 -1.2254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0108 -1.2254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3998 -1.3734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3998 -1.3734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8698 -0.8450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8698 -0.8450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3374 -1.0918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3374 -1.0918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4303 -1.2399 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4303 -1.2399 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1239 -1.4614 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1239 -1.4614 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7045 -1.7395 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7045 -1.7395 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2487 -1.4514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2487 -1.4514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8273 -1.6064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8273 -1.6064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4303 -1.4449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4303 -1.4449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8273 -2.1365 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8273 -2.1365 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7359 -0.3076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7359 -0.3076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9390 -0.5211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9390 -0.5211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 22 20 1 0 0 0 0 | + | 22 20 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 19 1 0 0 0 0 | + | 34 19 1 0 0 0 0 |
| − | 30 40 1 0 0 0 0 | + | 30 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 41 43 2 0 0 0 0 | + | 41 43 2 0 0 0 0 |
| − | 36 44 1 0 0 0 0 | + | 36 44 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 44 45 | + | M SAL 1 2 44 45 |
| − | M SBL 1 1 48 | + | M SBL 1 1 48 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 48 -7.7408 6.4026 | + | M SBV 1 48 -7.7408 6.4026 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AAAGL0022 | + | ID FL7AAAGL0022 |
| − | KNApSAcK_ID C00006761 | + | KNApSAcK_ID C00006761 |
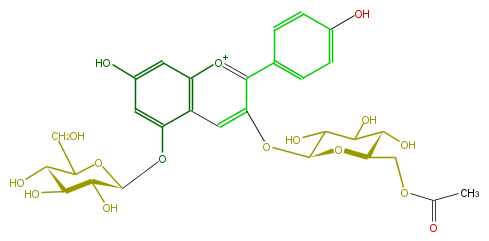
| − | NAME Pelargonidin 3-(6''-acetylglucoside)-5-glucoside | + | NAME Pelargonidin 3-(6''-acetylglucoside)-5-glucoside |
| − | CAS_RN - | + | CAS_RN - |
| − | FORMULA C29H33O16 | + | FORMULA C29H33O16 |
| − | EXACTMASS 637.176860008 | + | EXACTMASS 637.176860008 |
| − | AVERAGEMASS 637.56272 | + | AVERAGEMASS 637.56272 |
| − | SMILES OC(C1O)C(Oc(c23)cc(cc([o+1]c(c(c5)ccc(O)c5)c(OC(C(O)4)OC(COC(C)=O)C(O)C4O)c3)2)O)OC(C1O)CO | + | SMILES OC(C1O)C(Oc(c23)cc(cc([o+1]c(c(c5)ccc(O)c5)c(OC(C(O)4)OC(COC(C)=O)C(O)C4O)c3)2)O)OC(C1O)CO |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
45 49 0 0 0 0 0 0 0 0999 V2000
-2.1490 0.8335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1490 0.1911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5927 -0.1301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0364 0.1911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0364 0.8335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5927 1.1546 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4801 -0.1301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0762 0.1911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0762 0.8335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4801 1.1546 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
0.6323 1.1545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1993 0.8272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7663 1.1545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7663 1.8092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1993 2.1365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6323 1.8092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7051 1.1545 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3331 2.1365 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5927 -0.7722 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4923 -0.5296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6497 -0.2295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3489 -0.7506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9274 -0.5852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5094 -0.7506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8103 -0.2295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2317 -0.3948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0910 -0.3792 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4699 0.0177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2443 -0.4801 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0609 -0.7506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9165 -0.9433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5453 -1.4332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0108 -1.2254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3998 -1.3734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8698 -0.8450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3374 -1.0918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4303 -1.2399 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1239 -1.4614 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7045 -1.7395 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2487 -1.4514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8273 -1.6064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4303 -1.4449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8273 -2.1365 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7359 -0.3076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9390 -0.5211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
24 30 1 0 0 0 0
22 20 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 19 1 0 0 0 0
30 40 1 0 0 0 0
40 41 1 0 0 0 0
41 42 1 0 0 0 0
41 43 2 0 0 0 0
36 44 1 0 0 0 0
44 45 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 44 45
M SBL 1 1 48
M SMT 1 ^CH2OH
M SBV 1 48 -7.7408 6.4026
S SKP 8
ID FL7AAAGL0022
KNApSAcK_ID C00006761
NAME Pelargonidin 3-(6''-acetylglucoside)-5-glucoside
CAS_RN -
FORMULA C29H33O16
EXACTMASS 637.176860008
AVERAGEMASS 637.56272
SMILES OC(C1O)C(Oc(c23)cc(cc([o+1]c(c(c5)ccc(O)c5)c(OC(C(O)4)OC(COC(C)=O)C(O)C4O)c3)2)O)OC(C1O)CO
M END