Mol:FL7AAAGL0006
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 41 45 0 0 0 0 0 0 0 0999 V2000 | + | 41 45 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.5206 1.5518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5206 1.5518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5206 0.9095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5206 0.9095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9643 0.5883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9643 0.5883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4080 0.9095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4080 0.9095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4080 1.5518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4080 1.5518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9643 1.8730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9643 1.8730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8517 0.5883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8517 0.5883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2954 0.9095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2954 0.9095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2954 1.5518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2954 1.5518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8517 1.8730 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -0.8517 1.8730 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2607 1.8729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2607 1.8729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8277 1.5455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8277 1.5455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3947 1.8729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3947 1.8729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3947 2.5276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3947 2.5276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8277 2.8549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8277 2.8549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2607 2.5276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2607 2.5276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9615 2.8548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9615 2.8548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0767 1.8729 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0767 1.8729 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9643 -0.0538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9643 -0.0538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3574 0.5734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3574 0.5734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1564 -0.8560 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1564 -0.8560 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8819 -0.8560 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8819 -0.8560 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3605 -0.3516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3605 -0.3516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1823 0.3558 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1823 0.3558 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4568 0.3558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4568 0.3558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9782 -0.1486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9782 -0.1486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6453 0.2365 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6453 0.2365 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5973 -1.2969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5973 -1.2969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0334 -1.4217 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0334 -1.4217 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0767 -0.3516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0767 -0.3516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8921 -0.8000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8921 -0.8000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2207 -0.4124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2207 -0.4124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4337 -1.1579 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4337 -1.1579 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2207 -1.9078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2207 -1.9078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8921 -2.2956 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8921 -2.2956 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6791 -1.5500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6791 -1.5500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8921 -0.0982 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8921 -0.0982 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2108 -1.8570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2108 -1.8570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5691 -2.8549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5691 -2.8549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4955 -2.6581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4955 -2.6581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0788 -2.0747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0788 -2.0747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 26 21 1 1 0 0 0 | + | 26 21 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 21 28 1 0 0 0 0 | + | 21 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 32 20 1 0 0 0 0 | + | 32 20 1 0 0 0 0 |
| − | 37 25 1 0 0 0 0 | + | 37 25 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 44 -4.6557 3.3384 | + | M SBV 1 44 -4.6557 3.3384 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AAAGL0006 | + | ID FL7AAAGL0006 |
| − | KNApSAcK_ID C00006638 | + | KNApSAcK_ID C00006638 |
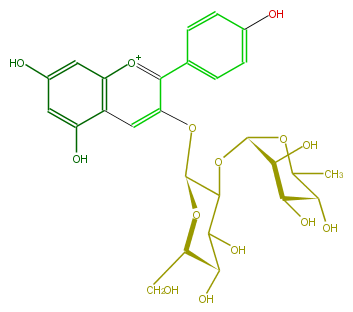
| − | NAME Pelargonidin 3-neohesperidoside;Pelargonidin 3-(2-rhamnosylglucoside) | + | NAME Pelargonidin 3-neohesperidoside;Pelargonidin 3-(2-rhamnosylglucoside) |
| − | CAS_RN 118964-12-8 | + | CAS_RN 118964-12-8 |
| − | FORMULA C27H31O14 | + | FORMULA C27H31O14 |
| − | EXACTMASS 579.1713807 | + | EXACTMASS 579.1713807 |
| − | AVERAGEMASS 579.52664 | + | AVERAGEMASS 579.52664 |
| − | SMILES O(C5CO)C(C(C(C5O)O)OC(C4O)OC(C)C(C4O)O)Oc(c2c(c3)ccc(O)c3)cc(c([o+1]2)1)c(cc(O)c1)O | + | SMILES O(C5CO)C(C(C(C5O)O)OC(C4O)OC(C)C(C4O)O)Oc(c2c(c3)ccc(O)c3)cc(c([o+1]2)1)c(cc(O)c1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
41 45 0 0 0 0 0 0 0 0999 V2000
-2.5206 1.5518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5206 0.9095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9643 0.5883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4080 0.9095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4080 1.5518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9643 1.8730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8517 0.5883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2954 0.9095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2954 1.5518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8517 1.8730 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
0.2607 1.8729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8277 1.5455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3947 1.8729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3947 2.5276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8277 2.8549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2607 2.5276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9615 2.8548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0767 1.8729 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9643 -0.0538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3574 0.5734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1564 -0.8560 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8819 -0.8560 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3605 -0.3516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1823 0.3558 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4568 0.3558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9782 -0.1486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6453 0.2365 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5973 -1.2969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0334 -1.4217 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0767 -0.3516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8921 -0.8000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2207 -0.4124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4337 -1.1579 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2207 -1.9078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8921 -2.2956 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6791 -1.5500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8921 -0.0982 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2108 -1.8570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5691 -2.8549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4955 -2.6581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0788 -2.0747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
14 17 1 0 0 0 0
1 18 1 0 0 0 0
3 19 1 0 0 0 0
8 20 1 0 0 0 0
22 21 1 1 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 1 0 0 0
26 21 1 1 0 0 0
26 27 1 0 0 0 0
21 28 1 0 0 0 0
22 29 1 0 0 0 0
23 30 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
36 38 1 0 0 0 0
35 39 1 0 0 0 0
32 20 1 0 0 0 0
37 25 1 0 0 0 0
34 40 1 0 0 0 0
40 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 44
M SMT 1 ^CH2OH
M SBV 1 44 -4.6557 3.3384
S SKP 8
ID FL7AAAGL0006
KNApSAcK_ID C00006638
NAME Pelargonidin 3-neohesperidoside;Pelargonidin 3-(2-rhamnosylglucoside)
CAS_RN 118964-12-8
FORMULA C27H31O14
EXACTMASS 579.1713807
AVERAGEMASS 579.52664
SMILES O(C5CO)C(C(C(C5O)O)OC(C4O)OC(C)C(C4O)O)Oc(c2c(c3)ccc(O)c3)cc(c([o+1]2)1)c(cc(O)c1)O
M END