Mol:FL7AAAGA0006
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 54 59 0 0 0 0 0 0 0 0999 V2000 | + | 54 59 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.3986 0.2933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3986 0.2933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7881 0.6457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7881 0.6457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7881 1.3506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7881 1.3506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3986 1.7030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3986 1.7030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0090 1.3506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0090 1.3506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0090 0.6457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0090 0.6457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1777 0.2933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1777 0.2933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5673 0.6457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5673 0.6457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5673 1.3506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5673 1.3506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1777 1.7030 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -1.1777 1.7030 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0142 1.6863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0142 1.6863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5978 1.3494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5978 1.3494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1814 1.6863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1814 1.6863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1814 2.3602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1814 2.3602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5978 2.6971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5978 2.6971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0142 2.3602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0142 2.3602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6455 2.6281 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6455 2.6281 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5084 1.6389 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5084 1.6389 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3986 -0.1688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3986 -0.1688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1298 0.2108 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1298 0.2108 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1980 -0.4486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1980 -0.4486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9050 -0.8738 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9050 -0.8738 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6817 -1.6680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6817 -1.6680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8943 -2.4651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8943 -2.4651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1873 -2.0400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1873 -2.0400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4105 -1.2457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4105 -1.2457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2799 -1.6684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2799 -1.6684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6059 -1.9944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6059 -1.9944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3345 -2.4651 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3345 -2.4651 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3633 -2.6971 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3633 -2.6971 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2845 -0.3308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2845 -0.3308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8720 -1.0453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8720 -1.0453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6653 -0.8186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6653 -0.8186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4633 -1.0278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4633 -1.0278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8759 -0.3133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8759 -0.3133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0826 -0.5399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0826 -0.5399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3756 -0.6018 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3756 -0.6018 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2430 0.0589 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2430 0.0589 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9870 -0.7254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9870 -0.7254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4024 -0.9653 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4024 -0.9653 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2381 -0.8713 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2381 -0.8713 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7779 -0.4666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7779 -0.4666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5302 -0.0376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5302 -0.0376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8073 0.4423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8073 0.4423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1503 -0.0376 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1503 -0.0376 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4560 0.4423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4560 0.4423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9257 0.9120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9257 0.9120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6000 0.7313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6000 0.7313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0937 1.2250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0937 1.2250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9130 1.8993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9130 1.8993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2386 2.0800 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2386 2.0800 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7450 1.5863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7450 1.5863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2624 2.2487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2624 2.2487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5084 1.1138 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5084 1.1138 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 3 1 0 0 0 0 | + | 10 3 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 5 18 1 0 0 0 0 | + | 5 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 21 26 1 1 0 0 0 | + | 21 26 1 1 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 23 27 1 0 0 0 0 | + | 23 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 24 29 1 0 0 0 0 | + | 24 29 1 0 0 0 0 |
| − | 25 30 1 0 0 0 0 | + | 25 30 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 35 37 1 0 0 0 0 | + | 35 37 1 0 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 26 41 1 0 0 0 0 | + | 26 41 1 0 0 0 0 |
| − | 41 32 1 0 0 0 0 | + | 41 32 1 0 0 0 0 |
| − | 31 42 1 0 0 0 0 | + | 31 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 43 45 2 0 0 0 0 | + | 43 45 2 0 0 0 0 |
| − | 44 46 2 0 0 0 0 | + | 44 46 2 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 2 0 0 0 0 | + | 47 48 2 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 51 52 2 0 0 0 0 | + | 51 52 2 0 0 0 0 |
| − | 52 47 1 0 0 0 0 | + | 52 47 1 0 0 0 0 |
| − | 50 53 1 0 0 0 0 | + | 50 53 1 0 0 0 0 |
| − | 49 54 1 0 0 0 0 | + | 49 54 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AAAGA0006 | + | ID FL7AAAGA0006 |
| − | KNApSAcK_ID C00011076 | + | KNApSAcK_ID C00011076 |
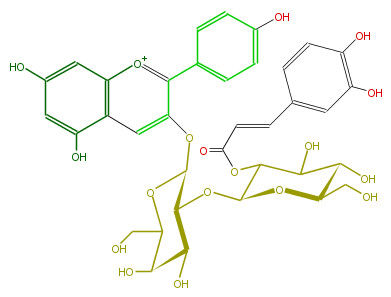
| − | NAME Pelargonidin 3-[2''-(2'''-trans-caffeoyl-beta-D-glucopyranosyl)-beta-D-galactopyranoside] | + | NAME Pelargonidin 3-[2''-(2'''-trans-caffeoyl-beta-D-glucopyranosyl)-beta-D-galactopyranoside] |
| − | CAS_RN 202338-37-2 | + | CAS_RN 202338-37-2 |
| − | FORMULA C36H37O18 | + | FORMULA C36H37O18 |
| − | EXACTMASS 757.1979893800001 | + | EXACTMASS 757.1979893800001 |
| − | AVERAGEMASS 757.66818 | + | AVERAGEMASS 757.66818 |
| − | SMILES [o+1](c2c(c6)ccc(O)c6)c(c1)c(cc(OC(C3OC(O4)C(OC(C=Cc(c5)ccc(c5O)O)=O)C(C(C4CO)O)O)OC(CO)C(C3O)O)2)c(cc(O)1)O | + | SMILES [o+1](c2c(c6)ccc(O)c6)c(c1)c(cc(OC(C3OC(O4)C(OC(C=Cc(c5)ccc(c5O)O)=O)C(C(C4CO)O)O)OC(CO)C(C3O)O)2)c(cc(O)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
54 59 0 0 0 0 0 0 0 0999 V2000
-2.3986 0.2933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7881 0.6457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7881 1.3506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3986 1.7030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0090 1.3506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0090 0.6457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1777 0.2933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5673 0.6457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5673 1.3506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1777 1.7030 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
0.0142 1.6863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5978 1.3494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1814 1.6863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1814 2.3602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5978 2.6971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0142 2.3602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6455 2.6281 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5084 1.6389 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3986 -0.1688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1298 0.2108 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1980 -0.4486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9050 -0.8738 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6817 -1.6680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8943 -2.4651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1873 -2.0400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4105 -1.2457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2799 -1.6684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6059 -1.9944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3345 -2.4651 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3633 -2.6971 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2845 -0.3308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8720 -1.0453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6653 -0.8186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4633 -1.0278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8759 -0.3133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0826 -0.5399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3756 -0.6018 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2430 0.0589 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9870 -0.7254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4024 -0.9653 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2381 -0.8713 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7779 -0.4666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5302 -0.0376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8073 0.4423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1503 -0.0376 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4560 0.4423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9257 0.9120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6000 0.7313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0937 1.2250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9130 1.8993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2386 2.0800 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7450 1.5863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2624 2.2487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5084 1.1138 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
2 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 3 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
14 17 1 0 0 0 0
5 18 1 0 0 0 0
1 19 1 0 0 0 0
8 20 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 25 1 1 0 0 0
25 26 1 1 0 0 0
21 26 1 1 0 0 0
20 21 1 0 0 0 0
23 27 1 0 0 0 0
27 28 1 0 0 0 0
24 29 1 0 0 0 0
25 30 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
35 37 1 0 0 0 0
36 38 1 0 0 0 0
34 39 1 0 0 0 0
39 40 1 0 0 0 0
26 41 1 0 0 0 0
41 32 1 0 0 0 0
31 42 1 0 0 0 0
42 43 1 0 0 0 0
43 44 1 0 0 0 0
43 45 2 0 0 0 0
44 46 2 0 0 0 0
46 47 1 0 0 0 0
47 48 2 0 0 0 0
48 49 1 0 0 0 0
49 50 2 0 0 0 0
50 51 1 0 0 0 0
51 52 2 0 0 0 0
52 47 1 0 0 0 0
50 53 1 0 0 0 0
49 54 1 0 0 0 0
S SKP 8
ID FL7AAAGA0006
KNApSAcK_ID C00011076
NAME Pelargonidin 3-[2''-(2'''-trans-caffeoyl-beta-D-glucopyranosyl)-beta-D-galactopyranoside]
CAS_RN 202338-37-2
FORMULA C36H37O18
EXACTMASS 757.1979893800001
AVERAGEMASS 757.66818
SMILES [o+1](c2c(c6)ccc(O)c6)c(c1)c(cc(OC(C3OC(O4)C(OC(C=Cc(c5)ccc(c5O)O)=O)C(C(C4CO)O)O)OC(CO)C(C3O)O)2)c(cc(O)1)O
M END