Mol:FL63ACNS0017
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.4936 0.6835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4936 0.6835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4936 0.0617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4936 0.0617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0449 -0.2492 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0449 -0.2492 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5834 0.0617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5834 0.0617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5834 0.6835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5834 0.6835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0449 0.9944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0449 0.9944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1220 -0.2492 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1220 -0.2492 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6605 0.0617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6605 0.0617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6605 0.6835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6605 0.6835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1220 0.9944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1220 0.9944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0318 0.9943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0318 0.9943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1715 -0.2333 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1715 -0.2333 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1400 0.9604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1400 0.9604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6848 0.6459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6848 0.6459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2295 0.9604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2295 0.9604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2295 1.5894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2295 1.5894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6848 1.9039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6848 1.9039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1400 1.5894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1400 1.5894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8277 1.9487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8277 1.9487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0449 -0.8707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0449 -0.8707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6848 2.5322 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6848 2.5322 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5701 0.6835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5701 0.6835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1071 0.9936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1071 0.9936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5701 0.0634 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5701 0.0634 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1071 1.6563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1071 1.6563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6810 1.9876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6810 1.9876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2549 1.6563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2549 1.6563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2549 0.9936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2549 0.9936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6810 0.6623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6810 0.6623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6810 2.6490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6810 2.6490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8277 1.9870 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8277 1.9870 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8277 0.6629 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8277 0.6629 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4730 -1.4408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4730 -1.4408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0079 -1.9059 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0079 -1.9059 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1330 -1.4408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1330 -1.4408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4819 -0.8364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4819 -0.8364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1798 -0.8364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1798 -0.8364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5288 -1.4408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5288 -1.4408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1798 -2.0452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1798 -2.0452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4819 -2.0452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4819 -2.0452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5284 -0.2327 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5284 -0.2327 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2259 -1.4408 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2259 -1.4408 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5284 -2.6490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5284 -2.6490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 1 11 1 0 0 0 0 | + | 1 11 1 0 0 0 0 |
| − | 8 12 1 1 0 0 0 | + | 8 12 1 1 0 0 0 |
| − | 9 13 1 6 0 0 0 | + | 9 13 1 6 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 19 16 1 0 0 0 0 | + | 19 16 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 17 21 1 0 0 0 0 | + | 17 21 1 0 0 0 0 |
| − | 11 22 1 0 0 0 0 | + | 11 22 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 22 24 2 0 0 0 0 | + | 22 24 2 0 0 0 0 |
| − | 23 25 2 0 0 0 0 | + | 23 25 2 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 2 0 0 0 0 | + | 26 27 2 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 2 0 0 0 0 | + | 28 29 2 0 0 0 0 |
| − | 29 23 1 0 0 0 0 | + | 29 23 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 27 31 1 0 0 0 0 | + | 27 31 1 0 0 0 0 |
| − | 28 32 1 0 0 0 0 | + | 28 32 1 0 0 0 0 |
| − | 20 33 1 0 0 0 0 | + | 20 33 1 0 0 0 0 |
| − | 33 34 2 0 0 0 0 | + | 33 34 2 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 35 36 2 0 0 0 0 | + | 35 36 2 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 2 0 0 0 0 | + | 37 38 2 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 40 35 1 0 0 0 0 | + | 40 35 1 0 0 0 0 |
| − | 37 41 1 0 0 0 0 | + | 37 41 1 0 0 0 0 |
| − | 38 42 1 0 0 0 0 | + | 38 42 1 0 0 0 0 |
| − | 39 43 1 0 0 0 0 | + | 39 43 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL63ACNS0017 | + | ID FL63ACNS0017 |
| − | KNApSAcK_ID C00008877 | + | KNApSAcK_ID C00008877 |
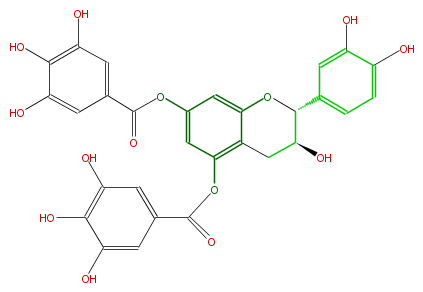
| − | NAME Catechin 5,7,-di-O-gallate | + | NAME Catechin 5,7,-di-O-gallate |
| − | CAS_RN - | + | CAS_RN - |
| − | FORMULA C29H22O14 | + | FORMULA C29H22O14 |
| − | EXACTMASS 594.100955412 | + | EXACTMASS 594.100955412 |
| − | AVERAGEMASS 594.47658 | + | AVERAGEMASS 594.47658 |
| − | SMILES c(O)(c1)c(O)ccc(C(O2)C(Cc(c(OC(=O)c(c5)cc(c(c(O)5)O)O)4)c2cc(c4)OC(=O)c(c3)cc(c(c3O)O)O)O)1 | + | SMILES c(O)(c1)c(O)ccc(C(O2)C(Cc(c(OC(=O)c(c5)cc(c(c(O)5)O)O)4)c2cc(c4)OC(=O)c(c3)cc(c(c3O)O)O)O)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-0.4936 0.6835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4936 0.0617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0449 -0.2492 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5834 0.0617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5834 0.6835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0449 0.9944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1220 -0.2492 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6605 0.0617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6605 0.6835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1220 0.9944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0318 0.9943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1715 -0.2333 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1400 0.9604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6848 0.6459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2295 0.9604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2295 1.5894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6848 1.9039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1400 1.5894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8277 1.9487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0449 -0.8707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6848 2.5322 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5701 0.6835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1071 0.9936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5701 0.0634 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1071 1.6563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6810 1.9876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2549 1.6563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2549 0.9936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6810 0.6623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6810 2.6490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8277 1.9870 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8277 0.6629 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4730 -1.4408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0079 -1.9059 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1330 -1.4408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4819 -0.8364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1798 -0.8364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5288 -1.4408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1798 -2.0452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4819 -2.0452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5284 -0.2327 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2259 -1.4408 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5284 -2.6490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
1 11 1 0 0 0 0
8 12 1 1 0 0 0
9 13 1 6 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
19 16 1 0 0 0 0
3 20 1 0 0 0 0
17 21 1 0 0 0 0
11 22 1 0 0 0 0
22 23 1 0 0 0 0
22 24 2 0 0 0 0
23 25 2 0 0 0 0
25 26 1 0 0 0 0
26 27 2 0 0 0 0
27 28 1 0 0 0 0
28 29 2 0 0 0 0
29 23 1 0 0 0 0
26 30 1 0 0 0 0
27 31 1 0 0 0 0
28 32 1 0 0 0 0
20 33 1 0 0 0 0
33 34 2 0 0 0 0
33 35 1 0 0 0 0
35 36 2 0 0 0 0
36 37 1 0 0 0 0
37 38 2 0 0 0 0
38 39 1 0 0 0 0
39 40 2 0 0 0 0
40 35 1 0 0 0 0
37 41 1 0 0 0 0
38 42 1 0 0 0 0
39 43 1 0 0 0 0
S SKP 8
ID FL63ACNS0017
KNApSAcK_ID C00008877
NAME Catechin 5,7,-di-O-gallate
CAS_RN -
FORMULA C29H22O14
EXACTMASS 594.100955412
AVERAGEMASS 594.47658
SMILES c(O)(c1)c(O)ccc(C(O2)C(Cc(c(OC(=O)c(c5)cc(c(c(O)5)O)O)4)c2cc(c4)OC(=O)c(c3)cc(c(c3O)O)O)O)1
M END