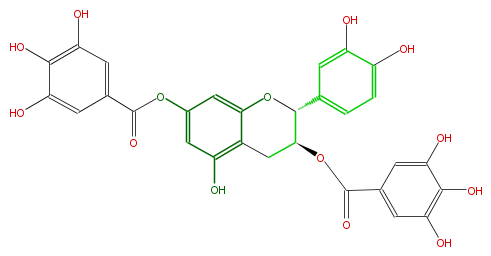
Mol:FL63ACNS0016
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.1719 0.3291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1719 0.3291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1719 -0.2928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1719 -0.2928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6334 -0.6037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6334 -0.6037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0949 -0.2928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0949 -0.2928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0949 0.3291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0949 0.3291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6334 0.6400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6334 0.6400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4437 -0.6037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4437 -0.6037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9822 -0.2928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9822 -0.2928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9822 0.3291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9822 0.3291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4437 0.6400 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4437 0.6400 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7101 0.6398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7101 0.6398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4932 -0.5878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4932 -0.5878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4617 0.6059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4617 0.6059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0064 0.2914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0064 0.2914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5512 0.6059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5512 0.6059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5512 1.2349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5512 1.2349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0064 1.5494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0064 1.5494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4617 1.2349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4617 1.2349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1494 1.5943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1494 1.5943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6334 -1.2252 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6334 -1.2252 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0064 2.1778 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0064 2.1778 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0043 -1.9204 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0043 -1.9204 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0043 -1.2591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0043 -1.2591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6867 -1.2441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6867 -1.2441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9899 -1.7694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9899 -1.7694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5964 -1.7694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5964 -1.7694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8996 -1.2441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8996 -1.2441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5964 -0.7189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5964 -0.7189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9899 -0.7189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9899 -0.7189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8996 -0.1938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8996 -0.1938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5060 -1.2441 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5060 -1.2441 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8996 -2.2945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8996 -2.2945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2484 0.3291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2484 0.3291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7854 0.6391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7854 0.6391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2484 -0.2911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2484 -0.2911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7854 1.3018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7854 1.3018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3593 1.6331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3593 1.6331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9332 1.3018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9332 1.3018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9332 0.6391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9332 0.6391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3593 0.3078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3593 0.3078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3593 2.2945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3593 2.2945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5060 1.6325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5060 1.6325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5060 0.3084 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5060 0.3084 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 1 11 1 0 0 0 0 | + | 1 11 1 0 0 0 0 |
| − | 8 12 1 1 0 0 0 | + | 8 12 1 1 0 0 0 |
| − | 9 13 1 6 0 0 0 | + | 9 13 1 6 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 19 16 1 0 0 0 0 | + | 19 16 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 17 21 1 0 0 0 0 | + | 17 21 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 2 0 0 0 0 | + | 24 25 2 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 2 0 0 0 0 | + | 26 27 2 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 2 0 0 0 0 | + | 28 29 2 0 0 0 0 |
| − | 29 24 1 0 0 0 0 | + | 29 24 1 0 0 0 0 |
| − | 28 30 1 0 0 0 0 | + | 28 30 1 0 0 0 0 |
| − | 27 31 1 0 0 0 0 | + | 27 31 1 0 0 0 0 |
| − | 26 32 1 0 0 0 0 | + | 26 32 1 0 0 0 0 |
| − | 23 22 2 0 0 0 0 | + | 23 22 2 0 0 0 0 |
| − | 23 12 1 0 0 0 0 | + | 23 12 1 0 0 0 0 |
| − | 11 33 1 0 0 0 0 | + | 11 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 33 35 2 0 0 0 0 | + | 33 35 2 0 0 0 0 |
| − | 34 36 2 0 0 0 0 | + | 34 36 2 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 2 0 0 0 0 | + | 37 38 2 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 40 34 1 0 0 0 0 | + | 40 34 1 0 0 0 0 |
| − | 37 41 1 0 0 0 0 | + | 37 41 1 0 0 0 0 |
| − | 38 42 1 0 0 0 0 | + | 38 42 1 0 0 0 0 |
| − | 39 43 1 0 0 0 0 | + | 39 43 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL63ACNS0016 | + | ID FL63ACNS0016 |
| − | KNApSAcK_ID C00008876 | + | KNApSAcK_ID C00008876 |
| − | NAME Catechin 3,7,-di-O-galate | + | NAME Catechin 3,7,-di-O-galate |
| − | CAS_RN 128397-04-6 | + | CAS_RN 128397-04-6 |
| − | FORMULA C29H22O14 | + | FORMULA C29H22O14 |
| − | EXACTMASS 594.100955412 | + | EXACTMASS 594.100955412 |
| − | AVERAGEMASS 594.47658 | + | AVERAGEMASS 594.47658 |
| − | SMILES c(c1)(c(c(cc1C(OC(C(c(c5)ccc(O)c5O)2)Cc(c4O)c(cc(c4)OC(=O)c(c3)cc(c(O)c3O)O)O2)=O)O)O)O | + | SMILES c(c1)(c(c(cc1C(OC(C(c(c5)ccc(O)c5O)2)Cc(c4O)c(cc(c4)OC(=O)c(c3)cc(c(O)c3O)O)O2)=O)O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-1.1719 0.3291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1719 -0.2928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6334 -0.6037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0949 -0.2928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0949 0.3291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6334 0.6400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4437 -0.6037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9822 -0.2928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9822 0.3291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4437 0.6400 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7101 0.6398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4932 -0.5878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4617 0.6059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0064 0.2914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5512 0.6059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5512 1.2349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0064 1.5494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4617 1.2349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1494 1.5943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6334 -1.2252 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0064 2.1778 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0043 -1.9204 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0043 -1.2591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6867 -1.2441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9899 -1.7694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5964 -1.7694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8996 -1.2441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5964 -0.7189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9899 -0.7189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8996 -0.1938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5060 -1.2441 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8996 -2.2945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2484 0.3291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7854 0.6391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2484 -0.2911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7854 1.3018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3593 1.6331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9332 1.3018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9332 0.6391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3593 0.3078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3593 2.2945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5060 1.6325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5060 0.3084 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
1 11 1 0 0 0 0
8 12 1 1 0 0 0
9 13 1 6 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
19 16 1 0 0 0 0
3 20 1 0 0 0 0
17 21 1 0 0 0 0
23 24 1 0 0 0 0
24 25 2 0 0 0 0
25 26 1 0 0 0 0
26 27 2 0 0 0 0
27 28 1 0 0 0 0
28 29 2 0 0 0 0
29 24 1 0 0 0 0
28 30 1 0 0 0 0
27 31 1 0 0 0 0
26 32 1 0 0 0 0
23 22 2 0 0 0 0
23 12 1 0 0 0 0
11 33 1 0 0 0 0
33 34 1 0 0 0 0
33 35 2 0 0 0 0
34 36 2 0 0 0 0
36 37 1 0 0 0 0
37 38 2 0 0 0 0
38 39 1 0 0 0 0
39 40 2 0 0 0 0
40 34 1 0 0 0 0
37 41 1 0 0 0 0
38 42 1 0 0 0 0
39 43 1 0 0 0 0
S SKP 8
ID FL63ACNS0016
KNApSAcK_ID C00008876
NAME Catechin 3,7,-di-O-galate
CAS_RN 128397-04-6
FORMULA C29H22O14
EXACTMASS 594.100955412
AVERAGEMASS 594.47658
SMILES c(c1)(c(c(cc1C(OC(C(c(c5)ccc(O)c5O)2)Cc(c4O)c(cc(c4)OC(=O)c(c3)cc(c(O)c3O)O)O2)=O)O)O)O
M END