Mol:FL63ACGS0022
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.7526 0.3732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7526 0.3732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7526 -0.2249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7526 -0.2249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2346 -0.5240 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2346 -0.5240 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7166 -0.2249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7166 -0.2249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7166 0.3732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7166 0.3732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2346 0.6722 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2346 0.6722 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1986 -0.5240 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1986 -0.5240 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6806 -0.2249 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.6806 -0.2249 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.6806 0.3732 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -0.6806 0.3732 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -1.1986 0.6722 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1986 0.6722 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1626 0.6722 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1626 0.6722 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3597 0.3707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3597 0.3707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8820 0.6722 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8820 0.6722 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8820 1.2753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8820 1.2753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3597 1.5769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3597 1.5769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1626 1.2753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1626 1.2753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3819 1.5640 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3819 1.5640 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2706 0.6722 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2706 0.6722 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1626 -0.5240 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1626 -0.5240 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3597 2.1791 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3597 2.1791 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2346 -1.1208 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2346 -1.1208 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2107 -1.3829 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -4.2107 -1.3829 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -3.8395 -1.8729 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.8395 -1.8729 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.3050 -1.6650 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.3050 -1.6650 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.7892 -1.6595 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.7892 -1.6595 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.1640 -1.2846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1640 -1.2846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6316 -1.5314 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -3.6316 -1.5314 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -4.8707 -1.5598 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8707 -1.5598 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4181 -1.9011 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4181 -1.9011 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9987 -2.1791 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9987 -2.1791 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9437 1.9089 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.9437 1.9089 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.6429 1.3879 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.6429 1.3879 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.2213 1.5532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2213 1.5532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8033 1.3879 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.8033 1.3879 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 4.1042 1.9089 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 4.1042 1.9089 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.5257 1.7437 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 3.5257 1.7437 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.3850 1.7592 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3850 1.7592 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5673 2.2182 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5673 2.2182 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5382 1.6583 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5382 1.6583 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8377 -0.6115 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8377 -0.6115 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7921 -0.3129 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7921 -0.3129 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0029 1.0955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0029 1.0955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0029 0.2706 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0029 0.2706 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 6 0 0 0 | + | 9 11 1 6 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 17 14 1 0 0 0 0 | + | 17 14 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 8 19 1 1 0 0 0 | + | 8 19 1 1 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 3 21 1 0 0 0 0 | + | 3 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 25 21 1 0 0 0 0 | + | 25 21 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 32 17 1 0 0 0 0 | + | 32 17 1 0 0 0 0 |
| − | 27 40 1 0 0 0 0 | + | 27 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 42 43 | + | M SAL 2 2 42 43 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 46 4.1563 1.4146 | + | M SVB 2 46 4.1563 1.4146 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 44 -3.8377 -0.6115 | + | M SVB 1 44 -3.8377 -0.6115 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL63ACGS0022 | + | ID FL63ACGS0022 |
| − | KNApSAcK_ID C00008858 | + | KNApSAcK_ID C00008858 |
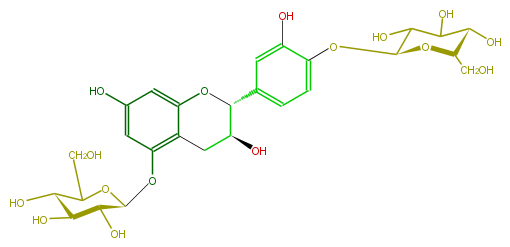
| − | NAME Catechin 5,4'-di-O-beta-D-glucopyranoside | + | NAME Catechin 5,4'-di-O-beta-D-glucopyranoside |
| − | CAS_RN 105330-56-1 | + | CAS_RN 105330-56-1 |
| − | FORMULA C27H34O16 | + | FORMULA C27H34O16 |
| − | EXACTMASS 614.18468504 | + | EXACTMASS 614.18468504 |
| − | AVERAGEMASS 614.54926 | + | AVERAGEMASS 614.54926 |
| − | SMILES O([C@@H](Oc(c2)c(C5)c(O[C@@H]([C@H]5O)c(c4)ccc(c4O)O[C@H](O3)C(C([C@@H](O)[C@@H](CO)3)O)O)cc2O)1)C(CO)[C@H](O)[C@@H]([C@@H]1O)O | + | SMILES O([C@@H](Oc(c2)c(C5)c(O[C@@H]([C@H]5O)c(c4)ccc(c4O)O[C@H](O3)C(C([C@@H](O)[C@@H](CO)3)O)O)cc2O)1)C(CO)[C@H](O)[C@@H]([C@@H]1O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-2.7526 0.3732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7526 -0.2249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2346 -0.5240 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7166 -0.2249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7166 0.3732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2346 0.6722 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1986 -0.5240 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6806 -0.2249 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.6806 0.3732 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-1.1986 0.6722 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1626 0.6722 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3597 0.3707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8820 0.6722 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8820 1.2753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3597 1.5769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1626 1.2753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3819 1.5640 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2706 0.6722 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1626 -0.5240 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3597 2.1791 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2346 -1.1208 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2107 -1.3829 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-3.8395 -1.8729 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.3050 -1.6650 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.7892 -1.6595 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.1640 -1.2846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6316 -1.5314 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-4.8707 -1.5598 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4181 -1.9011 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9987 -2.1791 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9437 1.9089 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.6429 1.3879 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.2213 1.5532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8033 1.3879 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
4.1042 1.9089 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.5257 1.7437 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.3850 1.7592 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5673 2.2182 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5382 1.6583 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8377 -0.6115 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7921 -0.3129 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0029 1.0955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0029 0.2706 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
9 11 1 6 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
17 14 1 0 0 0 0
1 18 1 0 0 0 0
8 19 1 1 0 0 0
15 20 1 0 0 0 0
3 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
25 21 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
36 38 1 0 0 0 0
35 39 1 0 0 0 0
32 17 1 0 0 0 0
27 40 1 0 0 0 0
40 41 1 0 0 0 0
34 42 1 0 0 0 0
42 43 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 42 43
M SBL 2 1 46
M SMT 2 CH2OH
M SVB 2 46 4.1563 1.4146
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 44
M SMT 1 CH2OH
M SVB 1 44 -3.8377 -0.6115
S SKP 8
ID FL63ACGS0022
KNApSAcK_ID C00008858
NAME Catechin 5,4'-di-O-beta-D-glucopyranoside
CAS_RN 105330-56-1
FORMULA C27H34O16
EXACTMASS 614.18468504
AVERAGEMASS 614.54926
SMILES O([C@@H](Oc(c2)c(C5)c(O[C@@H]([C@H]5O)c(c4)ccc(c4O)O[C@H](O3)C(C([C@@H](O)[C@@H](CO)3)O)O)cc2O)1)C(CO)[C@H](O)[C@@H]([C@@H]1O)O
M END