Mol:FL5FGANS0003
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 26 28 0 0 0 0 0 0 0 0999 V2000 | + | 26 28 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.7182 0.1271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7182 0.1271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8298 -0.5056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8298 -0.5056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3377 -0.9185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3377 -0.9185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7341 -0.6988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7341 -0.6988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6225 -0.0661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6225 -0.0661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1146 0.3468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1146 0.3468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2420 -1.1117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2420 -1.1117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3616 -0.8920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3616 -0.8920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4732 -0.2593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4732 -0.2593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0189 0.1536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0189 0.1536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3290 -1.6049 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3290 -1.6049 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0766 -0.0398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0766 -0.0398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5781 -0.4606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5781 -0.4606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1933 -0.2367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1933 -0.2367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3070 0.4081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3070 0.4081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8055 0.8290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8055 0.8290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1903 0.6050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1903 0.6050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2101 0.5398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2101 0.5398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4492 -1.5508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4492 -1.5508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9221 0.6320 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9221 0.6320 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1277 -1.5348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1277 -1.5348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8937 -2.1776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8937 -2.1776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7412 0.8628 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7412 0.8628 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1550 1.6730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1550 1.6730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4699 -0.1752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4699 -0.1752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9394 -0.8071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9394 -0.8071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 8 21 1 0 0 0 0 | + | 8 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 6 23 1 0 0 0 0 | + | 6 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 2 25 1 0 0 0 0 | + | 2 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 25 26 | + | M SAL 3 2 25 26 |
| − | M SBL 3 1 27 | + | M SBL 3 1 27 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 27 -2.5983 -0.3996 | + | M SVB 3 27 -2.5983 -0.3996 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 23 24 | + | M SAL 2 2 23 24 |
| − | M SBL 2 1 25 | + | M SBL 2 1 25 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 25 -1.0494 1.0455 | + | M SVB 2 25 -1.0494 1.0455 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 21 22 | + | M SAL 1 2 21 22 |
| − | M SBL 1 1 23 | + | M SBL 1 1 23 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 23 0.5403 -0.6973 | + | M SVB 1 23 0.5403 -0.6973 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FGANS0003 | + | ID FL5FGANS0003 |
| − | KNApSAcK_ID C00004668 | + | KNApSAcK_ID C00004668 |
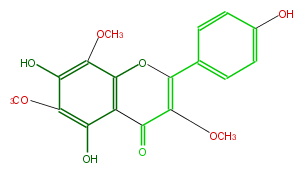
| − | NAME Sarothrin | + | NAME Sarothrin |
| − | CAS_RN 57393-71-2 | + | CAS_RN 57393-71-2 |
| − | FORMULA C18H16O8 | + | FORMULA C18H16O8 |
| − | EXACTMASS 360.08451748799996 | + | EXACTMASS 360.08451748799996 |
| − | AVERAGEMASS 360.31484 | + | AVERAGEMASS 360.31484 |
| − | SMILES c(C(O2)=C(C(c(c3O)c2c(c(c3OC)O)OC)=O)OC)(c1)ccc(O)c1 | + | SMILES c(C(O2)=C(C(c(c3O)c2c(c(c3OC)O)OC)=O)OC)(c1)ccc(O)c1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
26 28 0 0 0 0 0 0 0 0999 V2000
-1.7182 0.1271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8298 -0.5056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3377 -0.9185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7341 -0.6988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6225 -0.0661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1146 0.3468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2420 -1.1117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3616 -0.8920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4732 -0.2593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0189 0.1536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3290 -1.6049 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0766 -0.0398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5781 -0.4606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1933 -0.2367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3070 0.4081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8055 0.8290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1903 0.6050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2101 0.5398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4492 -1.5508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9221 0.6320 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1277 -1.5348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8937 -2.1776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7412 0.8628 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1550 1.6730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4699 -0.1752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9394 -0.8071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
3 19 1 0 0 0 0
15 20 1 0 0 0 0
8 21 1 0 0 0 0
21 22 1 0 0 0 0
6 23 1 0 0 0 0
23 24 1 0 0 0 0
2 25 1 0 0 0 0
25 26 1 0 0 0 0
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 25 26
M SBL 3 1 27
M SMT 3 OCH3
M SVB 3 27 -2.5983 -0.3996
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 23 24
M SBL 2 1 25
M SMT 2 OCH3
M SVB 2 25 -1.0494 1.0455
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 21 22
M SBL 1 1 23
M SMT 1 OCH3
M SVB 1 23 0.5403 -0.6973
S SKP 8
ID FL5FGANS0003
KNApSAcK_ID C00004668
NAME Sarothrin
CAS_RN 57393-71-2
FORMULA C18H16O8
EXACTMASS 360.08451748799996
AVERAGEMASS 360.31484
SMILES c(C(O2)=C(C(c(c3O)c2c(c(c3OC)O)OC)=O)OC)(c1)ccc(O)c1
M END