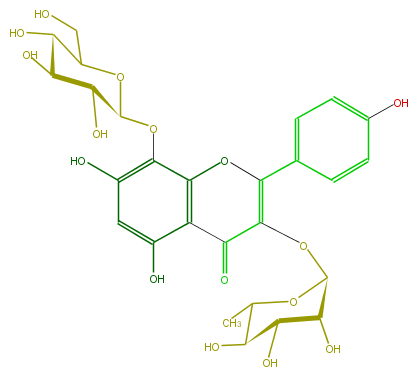
Mol:FL5FFAGS0026
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.7420 1.3737 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7420 1.3737 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7420 0.5487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7420 0.5487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4565 0.1362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4565 0.1362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1709 0.5487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1709 0.5487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1709 1.3737 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1709 1.3737 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4565 1.7862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4565 1.7862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0275 0.1362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0275 0.1362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3131 0.5487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3131 0.5487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4014 0.1362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4014 0.1362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4014 -0.6888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4014 -0.6888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3131 -1.1013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3131 -1.1013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0275 -0.6888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0275 -0.6888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1159 0.5487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1159 0.5487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8304 0.1362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8304 0.1362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8304 -0.6888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8304 -0.6888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1159 -1.1013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1159 -1.1013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3131 -1.8196 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3131 -1.8196 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5448 0.5487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5448 0.5487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8918 -1.1464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8918 -1.1464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7717 1.7205 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7717 1.7205 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1159 -1.8009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1159 -1.8009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1159 1.2027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1159 1.2027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8645 -2.3268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8645 -2.3268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7084 -3.1372 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7084 -3.1372 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3841 -2.6632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3841 -2.6632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2069 -2.5998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2069 -2.5998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3631 -1.7894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3631 -1.7894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6874 -2.2633 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6874 -2.2633 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4083 -3.2131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4083 -3.2131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1427 -3.4931 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1427 -3.4931 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1361 -3.1637 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1361 -3.1637 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3397 -2.6820 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3397 -2.6820 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1309 3.0717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1309 3.0717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1439 2.2465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1439 2.2465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3515 2.0160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3515 2.0160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7871 1.4138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7871 1.4138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7740 2.2390 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7740 2.2390 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5665 2.4697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5665 2.4697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6670 3.1340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6670 3.1340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2652 3.4931 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2652 3.4931 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7717 3.1102 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7717 3.1102 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6419 2.6772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6419 2.6772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2484 1.0999 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2484 1.0999 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 5 20 1 0 0 0 0 | + | 5 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 13 22 1 0 0 0 0 | + | 13 22 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 26 29 1 0 0 0 0 | + | 26 29 1 0 0 0 0 |
| − | 25 30 1 0 0 0 0 | + | 25 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 23 32 1 0 0 0 0 | + | 23 32 1 0 0 0 0 |
| − | 27 19 1 0 0 0 0 | + | 27 19 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 33 41 1 0 0 0 0 | + | 33 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 35 43 1 0 0 0 0 | + | 35 43 1 0 0 0 0 |
| − | 22 36 1 0 0 0 0 | + | 22 36 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FFAGS0026 | + | ID FL5FFAGS0026 |
| − | FORMULA C27H30O16 | + | FORMULA C27H30O16 |
| − | EXACTMASS 610.153384912 | + | EXACTMASS 610.153384912 |
| − | AVERAGEMASS 610.5175 | + | AVERAGEMASS 610.5175 |
| − | SMILES C(Oc(c52)c(cc(O)c2C(=O)C(=C(O5)c(c4)ccc(O)c4)OC(O3)C(O)C(O)C(C3C)O)O)(C1O)OC(C(C(O)1)O)CO | + | SMILES C(Oc(c52)c(cc(O)c2C(=O)C(=C(O5)c(c4)ccc(O)c4)OC(O3)C(O)C(O)C(C3C)O)O)(C1O)OC(C(C(O)1)O)CO |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
1.7420 1.3737 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7420 0.5487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4565 0.1362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1709 0.5487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1709 1.3737 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4565 1.7862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0275 0.1362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3131 0.5487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4014 0.1362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4014 -0.6888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3131 -1.1013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0275 -0.6888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1159 0.5487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8304 0.1362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8304 -0.6888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1159 -1.1013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3131 -1.8196 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5448 0.5487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8918 -1.1464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7717 1.7205 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1159 -1.8009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1159 1.2027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8645 -2.3268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7084 -3.1372 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3841 -2.6632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2069 -2.5998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3631 -1.7894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6874 -2.2633 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4083 -3.2131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1427 -3.4931 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1361 -3.1637 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3397 -2.6820 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1309 3.0717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1439 2.2465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3515 2.0160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7871 1.4138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7740 2.2390 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5665 2.4697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6670 3.1340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2652 3.4931 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7717 3.1102 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6419 2.6772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2484 1.0999 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
5 20 1 0 0 0 0
16 21 1 0 0 0 0
13 22 1 0 0 0 0
23 24 1 0 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
27 26 1 1 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
26 29 1 0 0 0 0
25 30 1 0 0 0 0
24 31 1 0 0 0 0
23 32 1 0 0 0 0
27 19 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
38 39 1 0 0 0 0
39 40 1 0 0 0 0
33 41 1 0 0 0 0
34 42 1 0 0 0 0
35 43 1 0 0 0 0
22 36 1 0 0 0 0
S SKP 5
ID FL5FFAGS0026
FORMULA C27H30O16
EXACTMASS 610.153384912
AVERAGEMASS 610.5175
SMILES C(Oc(c52)c(cc(O)c2C(=O)C(=C(O5)c(c4)ccc(O)c4)OC(O3)C(O)C(O)C(C3C)O)O)(C1O)OC(C(C(O)1)O)CO
M END