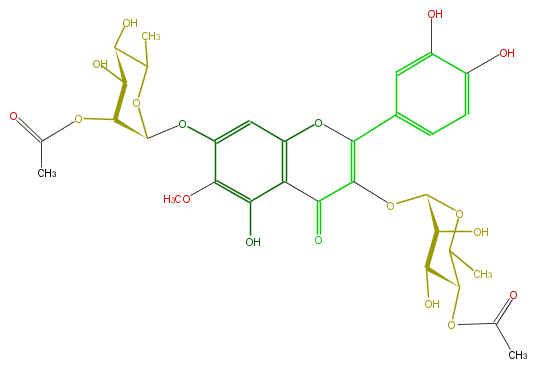
Mol:FL5FECGS0046
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 50 54 0 0 0 0 0 0 0 0999 V2000 | + | 50 54 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.0021 0.8726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0021 0.8726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0021 0.0477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0021 0.0477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2876 -0.3649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2876 -0.3649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4268 0.0477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4268 0.0477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4268 0.8726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4268 0.8726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2876 1.2851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2876 1.2851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1413 -0.3649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1413 -0.3649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8558 0.0477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8558 0.0477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8558 0.8726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8558 0.8726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1413 1.2851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1413 1.2851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1441 -1.1522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1441 -1.1522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7554 1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7554 1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4836 1.0235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4836 1.0235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2117 1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2117 1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2117 2.2847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2117 2.2847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4836 2.7051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4836 2.7051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7554 2.2847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7554 2.2847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2876 -1.1895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2876 -1.1895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9910 2.7347 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9910 2.7347 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6845 1.2470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6845 1.2470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6467 -0.4443 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6467 -0.4443 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3770 -1.7517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3770 -1.7517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0646 -2.1487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0646 -2.1487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8464 -1.3853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8464 -1.3853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0646 -0.6173 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0646 -0.6173 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3770 -0.2201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3770 -0.2201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5951 -0.9837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5951 -0.9837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4383 -0.9837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4383 -0.9837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4312 -2.4742 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4312 -2.4742 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9017 -2.9164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9017 -2.9164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4442 -1.8611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4442 -1.8611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0952 1.3258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0952 1.3258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4205 0.9364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4205 0.9364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6346 1.6853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6346 1.6853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4205 2.4388 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4205 2.4388 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0952 2.8283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0952 2.8283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8812 2.0793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8812 2.0793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8491 3.3603 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8491 3.3603 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4669 3.0812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4669 3.0812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4659 2.5087 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4659 2.5087 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8505 1.3588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8505 1.3588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4836 3.5457 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4836 3.5457 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8006 -2.9000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8006 -2.9000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1920 -2.3053 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1920 -2.3053 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1609 -3.5457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1609 -3.5457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6164 0.8748 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6164 0.8748 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1920 1.4155 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1920 1.4155 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6302 0.2381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6302 0.2381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6095 -0.3031 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6095 -0.3031 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6919 -0.8328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6919 -0.8328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 27 22 1 1 0 0 0 | + | 27 22 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 37 32 1 1 0 0 0 | + | 37 32 1 1 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 37 40 1 0 0 0 0 | + | 37 40 1 0 0 0 0 |
| − | 32 41 1 0 0 0 0 | + | 32 41 1 0 0 0 0 |
| − | 20 33 1 0 0 0 0 | + | 20 33 1 0 0 0 0 |
| − | 16 42 1 0 0 0 0 | + | 16 42 1 0 0 0 0 |
| − | 43 30 1 0 0 0 0 | + | 43 30 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 43 45 1 0 0 0 0 | + | 43 45 1 0 0 0 0 |
| − | 46 47 2 0 0 0 0 | + | 46 47 2 0 0 0 0 |
| − | 46 48 1 0 0 0 0 | + | 46 48 1 0 0 0 0 |
| − | 46 41 1 0 0 0 0 | + | 46 41 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 2 49 1 0 0 0 0 | + | 2 49 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 49 50 | + | M SAL 1 2 49 50 |
| − | M SBL 1 1 54 | + | M SBL 1 1 54 |
| − | M SMT 1 ^OCH3 | + | M SMT 1 ^OCH3 |
| − | M SBV 1 54 0.6075 0.3507 | + | M SBV 1 54 0.6075 0.3507 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FECGS0046 | + | ID FL5FECGS0046 |
| − | FORMULA C32H36O18 | + | FORMULA C32H36O18 |
| − | EXACTMASS 708.190164348 | + | EXACTMASS 708.190164348 |
| − | AVERAGEMASS 708.61744 | + | AVERAGEMASS 708.61744 |
| − | SMILES O(c43)C(=C(C(c(c(c(c(OC(O5)C(C(O)C(C(C)5)O)OC(C)=O)c4)OC)O)3)=O)OC(O2)C(C(O)C(OC(C)=O)C2C)O)c(c1)ccc(O)c1O | + | SMILES O(c43)C(=C(C(c(c(c(c(OC(O5)C(C(O)C(C(C)5)O)OC(C)=O)c4)OC)O)3)=O)OC(O2)C(C(O)C(OC(C)=O)C2C)O)c(c1)ccc(O)c1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
50 54 0 0 0 0 0 0 0 0999 V2000
-1.0021 0.8726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0021 0.0477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2876 -0.3649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4268 0.0477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4268 0.8726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2876 1.2851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1413 -0.3649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8558 0.0477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8558 0.8726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1413 1.2851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1441 -1.1522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7554 1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4836 1.0235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2117 1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2117 2.2847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4836 2.7051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7554 2.2847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2876 -1.1895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9910 2.7347 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6845 1.2470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6467 -0.4443 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3770 -1.7517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0646 -2.1487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8464 -1.3853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0646 -0.6173 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3770 -0.2201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5951 -0.9837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4383 -0.9837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4312 -2.4742 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9017 -2.9164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4442 -1.8611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0952 1.3258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4205 0.9364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6346 1.6853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4205 2.4388 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0952 2.8283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8812 2.0793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8491 3.3603 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4669 3.0812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4659 2.5087 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8505 1.3588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4836 3.5457 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8006 -2.9000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1920 -2.3053 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.1609 -3.5457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6164 0.8748 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1920 1.4155 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6302 0.2381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6095 -0.3031 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6919 -0.8328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
4 3 1 0 0 0 0
1 20 1 0 0 0 0
21 8 1 0 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 1 0 0 0
27 22 1 1 0 0 0
27 28 1 0 0 0 0
22 29 1 0 0 0 0
23 30 1 0 0 0 0
24 31 1 0 0 0 0
26 21 1 0 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 37 1 1 0 0 0
37 32 1 1 0 0 0
36 38 1 0 0 0 0
35 39 1 0 0 0 0
37 40 1 0 0 0 0
32 41 1 0 0 0 0
20 33 1 0 0 0 0
16 42 1 0 0 0 0
43 30 1 0 0 0 0
43 44 2 0 0 0 0
43 45 1 0 0 0 0
46 47 2 0 0 0 0
46 48 1 0 0 0 0
46 41 1 0 0 0 0
49 50 1 0 0 0 0
2 49 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 49 50
M SBL 1 1 54
M SMT 1 ^OCH3
M SBV 1 54 0.6075 0.3507
S SKP 5
ID FL5FECGS0046
FORMULA C32H36O18
EXACTMASS 708.190164348
AVERAGEMASS 708.61744
SMILES O(c43)C(=C(C(c(c(c(c(OC(O5)C(C(O)C(C(C)5)O)OC(C)=O)c4)OC)O)3)=O)OC(O2)C(C(O)C(OC(C)=O)C2C)O)c(c1)ccc(O)c1O
M END