Mol:FL5FECGS0039
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 38 42 0 0 0 0 0 0 0 0999 V2000 | + | 38 42 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.9389 -0.7752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9389 -0.7752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9389 -1.6002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9389 -1.6002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2244 -2.0127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2244 -2.0127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5099 -1.6002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5099 -1.6002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5099 -0.7752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5099 -0.7752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2244 -0.3626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2244 -0.3626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7955 -2.0127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7955 -2.0127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0810 -1.6002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0810 -1.6002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0810 -0.7752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0810 -0.7752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7955 -0.3626 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7955 -0.3626 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7955 -2.7772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7955 -2.7772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3668 -0.3629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3668 -0.3629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3614 -0.7832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3614 -0.7832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0896 -0.3629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0896 -0.3629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0896 0.4779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0896 0.4779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3614 0.8984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3614 0.8984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3668 0.4779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3668 0.4779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2244 -2.7360 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2244 -2.7360 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8946 0.9111 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8946 0.9111 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1685 0.6582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1685 0.6582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4682 0.3316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4682 0.3316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1984 0.0795 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1984 0.0795 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6890 -0.5232 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6890 -0.5232 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3894 -0.1967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3894 -0.1967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6590 0.0555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6590 0.0555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6574 0.8903 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6574 0.8903 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3011 0.2548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3011 0.2548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6926 -0.7171 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6926 -0.7171 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7234 -0.5202 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7234 -0.5202 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2084 -1.1877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2084 -1.1877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7234 -1.8551 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7234 -1.8551 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3409 -2.0275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3409 -2.0275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7714 -2.6697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7714 -2.6697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3614 1.7217 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3614 1.7217 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2691 2.7772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2691 2.7772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3405 -1.1079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3405 -1.1079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2084 -1.7156 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2084 -1.7156 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3802 -1.5556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3802 -1.5556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 25 27 1 0 0 0 0 | + | 25 27 1 0 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 21 19 1 0 0 0 0 | + | 21 19 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 1 29 1 0 0 0 0 | + | 1 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 2 1 0 0 0 0 | + | 31 2 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 8 32 1 0 0 0 0 | + | 8 32 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 16 34 1 0 0 0 0 | + | 16 34 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 23 36 1 0 0 0 0 | + | 23 36 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 32 33 | + | M SAL 1 2 32 33 |
| − | M SBL 1 1 37 | + | M SBL 1 1 37 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 37 -0.7402 0.4273 | + | M SBV 1 37 -0.7402 0.4273 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 34 35 | + | M SAL 2 2 34 35 |
| − | M SBL 2 1 39 | + | M SBL 2 1 39 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SBV 2 39 0.0000 -0.8232 | + | M SBV 2 39 0.0000 -0.8232 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 3 36 37 38 | + | M SAL 3 3 36 37 38 |
| − | M SBL 3 1 42 | + | M SBL 3 1 42 |
| − | M SMT 3 COOH | + | M SMT 3 COOH |
| − | M SBV 3 42 -0.6514 0.5847 | + | M SBV 3 42 -0.6514 0.5847 |
| − | S SKP 5 | + | S SKP 5 |
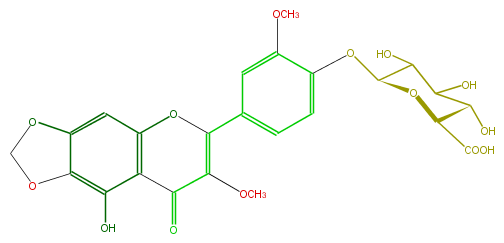
| − | ID FL5FECGS0039 | + | ID FL5FECGS0039 |
| − | FORMULA C24H22O14 | + | FORMULA C24H22O14 |
| − | EXACTMASS 534.100955412 | + | EXACTMASS 534.100955412 |
| − | AVERAGEMASS 534.42308 | + | AVERAGEMASS 534.42308 |
| − | SMILES c(OC)(c1)c(OC(C(O)5)OC(C(O)=O)C(C5O)O)ccc1C(=C(OC)4)Oc(c3C4=O)cc(O2)c(c3O)OC2 | + | SMILES c(OC)(c1)c(OC(C(O)5)OC(C(O)=O)C(C5O)O)ccc1C(=C(OC)4)Oc(c3C4=O)cc(O2)c(c3O)OC2 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
38 42 0 0 0 0 0 0 0 0999 V2000
-3.9389 -0.7752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9389 -1.6002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2244 -2.0127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5099 -1.6002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5099 -0.7752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2244 -0.3626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7955 -2.0127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0810 -1.6002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0810 -0.7752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7955 -0.3626 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7955 -2.7772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3668 -0.3629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3614 -0.7832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0896 -0.3629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0896 0.4779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3614 0.8984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3668 0.4779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2244 -2.7360 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8946 0.9111 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1685 0.6582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4682 0.3316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1984 0.0795 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6890 -0.5232 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3894 -0.1967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6590 0.0555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6574 0.8903 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3011 0.2548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6926 -0.7171 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7234 -0.5202 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.2084 -1.1877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7234 -1.8551 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3409 -2.0275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7714 -2.6697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3614 1.7217 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2691 2.7772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3405 -1.1079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2084 -1.7156 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3802 -1.5556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
20 21 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
25 27 1 0 0 0 0
24 28 1 0 0 0 0
21 19 1 0 0 0 0
19 15 1 0 0 0 0
1 29 1 0 0 0 0
29 30 1 0 0 0 0
30 31 1 0 0 0 0
31 2 1 0 0 0 0
32 33 1 0 0 0 0
8 32 1 0 0 0 0
34 35 1 0 0 0 0
16 34 1 0 0 0 0
36 37 2 0 0 0 0
36 38 1 0 0 0 0
23 36 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 32 33
M SBL 1 1 37
M SMT 1 OCH3
M SBV 1 37 -0.7402 0.4273
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 34 35
M SBL 2 1 39
M SMT 2 OCH3
M SBV 2 39 0.0000 -0.8232
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 3 36 37 38
M SBL 3 1 42
M SMT 3 COOH
M SBV 3 42 -0.6514 0.5847
S SKP 5
ID FL5FECGS0039
FORMULA C24H22O14
EXACTMASS 534.100955412
AVERAGEMASS 534.42308
SMILES c(OC)(c1)c(OC(C(O)5)OC(C(O)=O)C(C5O)O)ccc1C(=C(OC)4)Oc(c3C4=O)cc(O2)c(c3O)OC2
M END