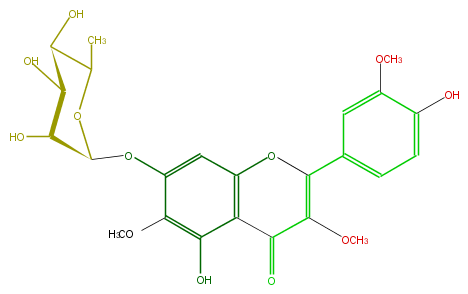
Mol:FL5FECGS0026
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 36 39 0 0 0 0 0 0 0 0999 V2000 | + | 36 39 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.3978 -0.7004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3978 -0.7004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3978 -1.5254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3978 -1.5254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6833 -1.9379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6833 -1.9379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0311 -1.5254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0311 -1.5254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0311 -0.7004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0311 -0.7004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6833 -0.2880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6833 -0.2880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7455 -1.9379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7455 -1.9379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4599 -1.5254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4599 -1.5254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4599 -0.7004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4599 -0.7004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7455 -0.2880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7455 -0.2880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7455 -2.7771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7455 -2.7771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1741 -0.2881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1741 -0.2881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9023 -0.7085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9023 -0.7085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6303 -0.2881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6303 -0.2881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6303 0.5526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6303 0.5526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9023 0.9731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9023 0.9731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1741 0.5526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1741 0.5526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1119 -0.2881 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1119 -0.2881 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6833 -2.7625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6833 -2.7625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2811 0.9284 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2811 0.9284 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6835 0.0876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6835 0.0876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8767 -0.3782 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8767 -0.3782 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1327 0.5176 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1327 0.5176 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8767 1.4189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8767 1.4189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6835 1.8848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6835 1.8848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4275 0.9889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4275 0.9889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2415 2.5550 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2415 2.5550 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8795 2.0316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8795 2.0316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1312 1.6151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1312 1.6151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2811 0.0893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2811 0.0893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9023 1.6623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9023 1.6623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4691 2.8147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4691 2.8147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2261 -1.9677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2261 -1.9677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3383 -2.6099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3383 -2.6099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9677 -1.8545 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9677 -1.8545 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4155 -2.8147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4155 -2.8147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 15 1 0 0 0 0 | + | 20 15 1 0 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 26 21 1 1 0 0 0 | + | 26 21 1 1 0 0 0 |
| − | 25 27 1 0 0 0 0 | + | 25 27 1 0 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 26 29 1 0 0 0 0 | + | 26 29 1 0 0 0 0 |
| − | 21 30 1 0 0 0 0 | + | 21 30 1 0 0 0 0 |
| − | 22 18 1 0 0 0 0 | + | 22 18 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 16 31 1 0 0 0 0 | + | 16 31 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 8 33 1 0 0 0 0 | + | 8 33 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 2 35 1 0 0 0 0 | + | 2 35 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 31 32 | + | M SAL 1 2 31 32 |
| − | M SBL 1 1 35 | + | M SBL 1 1 35 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 35 0.0000 -0.6892 | + | M SBV 1 35 0.0000 -0.6892 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 33 34 | + | M SAL 2 2 33 34 |
| − | M SBL 2 1 37 | + | M SBL 2 1 37 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SBV 2 37 -0.7662 0.4424 | + | M SBV 2 37 -0.7662 0.4424 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 35 36 | + | M SAL 3 2 35 36 |
| − | M SBL 3 1 39 | + | M SBL 3 1 39 |
| − | M SMT 3 ^ OCH3 | + | M SMT 3 ^ OCH3 |
| − | M SBV 3 39 0.5700 0.3291 | + | M SBV 3 39 0.5700 0.3291 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FECGS0026 | + | ID FL5FECGS0026 |
| − | FORMULA C24H26O12 | + | FORMULA C24H26O12 |
| − | EXACTMASS 506.142426296 | + | EXACTMASS 506.142426296 |
| − | AVERAGEMASS 506.45604 | + | AVERAGEMASS 506.45604 |
| − | SMILES O(C1Oc(c4OC)cc(c(c4O)3)OC(=C(OC)C3=O)c(c2)cc(c(c2)O)OC)C(C(O)C(O)C(O)1)C | + | SMILES O(C1Oc(c4OC)cc(c(c4O)3)OC(=C(OC)C3=O)c(c2)cc(c(c2)O)OC)C(C(O)C(O)C(O)1)C |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
36 39 0 0 0 0 0 0 0 0999 V2000
-1.3978 -0.7004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3978 -1.5254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6833 -1.9379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0311 -1.5254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0311 -0.7004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6833 -0.2880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7455 -1.9379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4599 -1.5254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4599 -0.7004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7455 -0.2880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7455 -2.7771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1741 -0.2881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9023 -0.7085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6303 -0.2881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6303 0.5526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9023 0.9731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1741 0.5526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1119 -0.2881 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6833 -2.7625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2811 0.9284 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6835 0.0876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8767 -0.3782 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1327 0.5176 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8767 1.4189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6835 1.8848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4275 0.9889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2415 2.5550 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8795 2.0316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1312 1.6151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2811 0.0893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9023 1.6623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4691 2.8147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2261 -1.9677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3383 -2.6099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9677 -1.8545 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4155 -2.8147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
3 19 1 0 0 0 0
20 15 1 0 0 0 0
22 21 1 1 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 1 0 0 0
26 21 1 1 0 0 0
25 27 1 0 0 0 0
24 28 1 0 0 0 0
26 29 1 0 0 0 0
21 30 1 0 0 0 0
22 18 1 0 0 0 0
31 32 1 0 0 0 0
16 31 1 0 0 0 0
33 34 1 0 0 0 0
8 33 1 0 0 0 0
35 36 1 0 0 0 0
2 35 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 31 32
M SBL 1 1 35
M SMT 1 OCH3
M SBV 1 35 0.0000 -0.6892
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 33 34
M SBL 2 1 37
M SMT 2 OCH3
M SBV 2 37 -0.7662 0.4424
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 35 36
M SBL 3 1 39
M SMT 3 ^ OCH3
M SBV 3 39 0.5700 0.3291
S SKP 5
ID FL5FECGS0026
FORMULA C24H26O12
EXACTMASS 506.142426296
AVERAGEMASS 506.45604
SMILES O(C1Oc(c4OC)cc(c(c4O)3)OC(=C(OC)C3=O)c(c2)cc(c(c2)O)OC)C(C(O)C(O)C(O)1)C
M END