Mol:FL5FEAGL0003
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 54 59 0 0 0 0 0 0 0 0999 V2000 | + | 54 59 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.1825 1.0747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1825 1.0747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1825 0.2653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1825 0.2653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4815 -0.1394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4815 -0.1394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2195 0.2653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2195 0.2653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2195 1.0747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2195 1.0747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4815 1.4795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4815 1.4795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9205 -0.1394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9205 -0.1394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6216 0.2653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6216 0.2653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6216 1.0747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6216 1.0747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9205 1.4795 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9205 1.4795 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9205 -0.9280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9205 -0.9280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3223 1.4793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3223 1.4793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0368 1.0669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0368 1.0669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7513 1.4793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7513 1.4793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7513 2.3044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7513 2.3044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0368 2.7168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0368 2.7168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3223 2.3044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3223 2.3044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8833 1.4793 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8833 1.4793 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3672 -0.2055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3672 -0.2055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4815 -0.9486 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4815 -0.9486 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2559 -1.3189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2559 -1.3189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8329 -2.0517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8329 -2.0517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6463 -1.8192 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6463 -1.8192 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4648 -2.0517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4648 -2.0517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8879 -1.3189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8879 -1.3189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0744 -1.5514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0744 -1.5514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5344 -1.4546 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5344 -1.4546 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4147 0.4054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4147 0.4054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9917 -0.3274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9917 -0.3274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8051 -0.0949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8051 -0.0949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6236 -0.3274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6236 -0.3274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0468 0.4054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0468 0.4054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2332 0.1729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2332 0.1729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6717 0.2760 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6717 0.2760 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7139 0.6898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7139 0.6898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7694 0.2184 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7694 0.2184 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3177 -0.2475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3177 -0.2475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7550 -0.6912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7550 -0.6912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2644 -1.9552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2644 -1.9552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1559 -2.4320 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1559 -2.4320 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4648 -2.7656 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4648 -2.7656 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5501 2.7656 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5501 2.7656 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7767 0.0935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7767 0.0935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2387 -0.6167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2387 -0.6167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4640 -0.3154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4640 -0.3154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7358 -0.4889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7358 -0.4889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2596 0.2361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2596 0.2361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0510 -0.0481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0510 -0.0481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4671 -0.1979 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4671 -0.1979 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0567 -0.6394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0567 -0.6394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9568 -0.9589 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9568 -0.9589 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0269 -0.1054 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0269 -0.1054 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6555 0.5351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6555 0.5351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8879 0.7980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8879 0.7980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 8 19 1 0 0 0 0 | + | 8 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 1 0 0 0 | + | 29 30 1 1 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 32 31 1 1 0 0 0 | + | 32 31 1 1 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 28 1 0 0 0 0 | + | 33 28 1 0 0 0 0 |
| − | 28 34 1 0 0 0 0 | + | 28 34 1 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 32 36 1 0 0 0 0 | + | 32 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 25 38 1 0 0 0 0 | + | 25 38 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 22 39 1 0 0 0 0 | + | 22 39 1 0 0 0 0 |
| − | 23 40 1 0 0 0 0 | + | 23 40 1 0 0 0 0 |
| − | 24 41 1 0 0 0 0 | + | 24 41 1 0 0 0 0 |
| − | 29 19 1 0 0 0 0 | + | 29 19 1 0 0 0 0 |
| − | 42 15 1 0 0 0 0 | + | 42 15 1 0 0 0 0 |
| − | 43 44 1 1 0 0 0 | + | 43 44 1 1 0 0 0 |
| − | 44 45 1 1 0 0 0 | + | 44 45 1 1 0 0 0 |
| − | 46 45 1 1 0 0 0 | + | 46 45 1 1 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 43 1 0 0 0 0 | + | 48 43 1 0 0 0 0 |
| − | 43 49 1 0 0 0 0 | + | 43 49 1 0 0 0 0 |
| − | 44 50 1 0 0 0 0 | + | 44 50 1 0 0 0 0 |
| − | 45 51 1 0 0 0 0 | + | 45 51 1 0 0 0 0 |
| − | 46 52 1 0 0 0 0 | + | 46 52 1 0 0 0 0 |
| − | 52 2 1 0 0 0 0 | + | 52 2 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 48 53 1 0 0 0 0 | + | 48 53 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 53 54 | + | M SAL 1 2 53 54 |
| − | M SBL 1 1 59 | + | M SBL 1 1 59 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 59 0.6045 -0.5832 | + | M SBV 1 59 0.6045 -0.5832 |
| − | S SKP 5 | + | S SKP 5 |
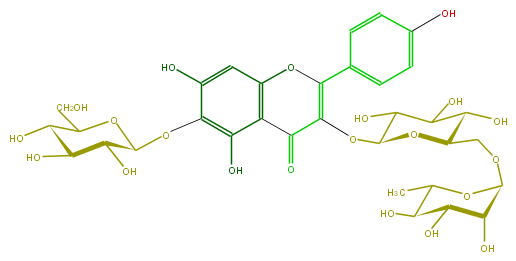
| − | ID FL5FEAGL0003 | + | ID FL5FEAGL0003 |
| − | FORMULA C33H40O21 | + | FORMULA C33H40O21 |
| − | EXACTMASS 772.206208342 | + | EXACTMASS 772.206208342 |
| − | AVERAGEMASS 772.6581 | + | AVERAGEMASS 772.6581 |
| − | SMILES C(C6O)(O)C(OC(C6O)C)OCC(O1)C(C(O)C(C1OC(C(=O)4)=C(c(c5)ccc(c5)O)Oc(c43)cc(O)c(c(O)3)OC(O2)C(O)C(O)C(O)C2CO)O)O | + | SMILES C(C6O)(O)C(OC(C6O)C)OCC(O1)C(C(O)C(C1OC(C(=O)4)=C(c(c5)ccc(c5)O)Oc(c43)cc(O)c(c(O)3)OC(O2)C(O)C(O)C(O)C2CO)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
54 59 0 0 0 0 0 0 0 0999 V2000
-1.1825 1.0747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1825 0.2653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4815 -0.1394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2195 0.2653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2195 1.0747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4815 1.4795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9205 -0.1394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6216 0.2653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6216 1.0747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9205 1.4795 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9205 -0.9280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3223 1.4793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0368 1.0669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7513 1.4793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7513 2.3044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0368 2.7168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3223 2.3044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8833 1.4793 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3672 -0.2055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4815 -0.9486 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2559 -1.3189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8329 -2.0517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6463 -1.8192 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4648 -2.0517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.8879 -1.3189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0744 -1.5514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5344 -1.4546 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4147 0.4054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9917 -0.3274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8051 -0.0949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6236 -0.3274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0468 0.4054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2332 0.1729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6717 0.2760 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7139 0.6898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.7694 0.2184 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.3177 -0.2475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.7550 -0.6912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2644 -1.9552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1559 -2.4320 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.4648 -2.7656 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5501 2.7656 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7767 0.0935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2387 -0.6167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4640 -0.3154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7358 -0.4889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2596 0.2361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0510 -0.0481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.4671 -0.1979 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0567 -0.6394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9568 -0.9589 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0269 -0.1054 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6555 0.5351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.8879 0.7980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
8 19 1 0 0 0 0
3 20 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
28 29 1 0 0 0 0
29 30 1 1 0 0 0
30 31 1 1 0 0 0
32 31 1 1 0 0 0
32 33 1 0 0 0 0
33 28 1 0 0 0 0
28 34 1 0 0 0 0
33 35 1 0 0 0 0
32 36 1 0 0 0 0
31 37 1 0 0 0 0
25 38 1 0 0 0 0
37 38 1 0 0 0 0
22 39 1 0 0 0 0
23 40 1 0 0 0 0
24 41 1 0 0 0 0
29 19 1 0 0 0 0
42 15 1 0 0 0 0
43 44 1 1 0 0 0
44 45 1 1 0 0 0
46 45 1 1 0 0 0
46 47 1 0 0 0 0
47 48 1 0 0 0 0
48 43 1 0 0 0 0
43 49 1 0 0 0 0
44 50 1 0 0 0 0
45 51 1 0 0 0 0
46 52 1 0 0 0 0
52 2 1 0 0 0 0
53 54 1 0 0 0 0
48 53 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 53 54
M SBL 1 1 59
M SMT 1 ^ CH2OH
M SBV 1 59 0.6045 -0.5832
S SKP 5
ID FL5FEAGL0003
FORMULA C33H40O21
EXACTMASS 772.206208342
AVERAGEMASS 772.6581
SMILES C(C6O)(O)C(OC(C6O)C)OCC(O1)C(C(O)C(C1OC(C(=O)4)=C(c(c5)ccc(c5)O)Oc(c43)cc(O)c(c(O)3)OC(O2)C(O)C(O)C(O)C2CO)O)O
M END