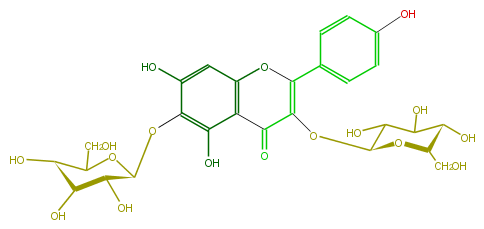
Mol:FL5FEAGL0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.4492 0.5646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4492 0.5646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4492 -0.0778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4492 -0.0778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8928 -0.3989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8928 -0.3989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3365 -0.0778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3365 -0.0778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3365 0.5646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3365 0.5646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8928 0.8858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8928 0.8858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2198 -0.3989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2198 -0.3989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7761 -0.0778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7761 -0.0778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7761 0.5646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7761 0.5646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2198 0.8858 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2198 0.8858 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2198 -0.8998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2198 -0.8998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3322 0.8857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3322 0.8857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8991 0.5583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8991 0.5583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4661 0.8857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4661 0.8857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4661 1.5403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4661 1.5403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8991 1.8677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8991 1.8677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3322 1.5403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3322 1.5403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0052 0.8857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0052 0.8857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2095 -0.5112 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2095 -0.5112 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0329 1.9366 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0329 1.9366 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8928 -1.0411 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8928 -1.0411 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0052 -0.3988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0052 -0.3988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9879 -1.0418 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -3.9879 -1.0418 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -3.5708 -1.5925 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.5708 -1.5925 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.9701 -1.3589 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.9701 -1.3589 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.3905 -1.3526 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.3905 -1.3526 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.8117 -0.9313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8117 -0.9313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3372 -1.2087 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -3.3372 -1.2087 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -4.6521 -0.9836 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6521 -0.9836 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9646 -2.1108 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9646 -2.1108 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6259 -1.9366 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6259 -1.9366 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6383 -0.2921 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.6383 -0.2921 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.3375 -0.8132 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.3375 -0.8132 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.9160 -0.6478 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9160 -0.6478 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4980 -0.8132 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.4980 -0.8132 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.7988 -0.2921 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.7988 -0.2921 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.2203 -0.4574 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 3.2203 -0.4574 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.0796 -0.4418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0796 -0.4418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2619 0.0171 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2619 0.0171 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2329 -0.5428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2329 -0.5428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3009 -0.7040 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3009 -0.7040 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1288 -0.1431 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1288 -0.1431 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6976 -1.1055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6976 -1.1055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6976 -1.9305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6976 -1.9305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 8 19 1 0 0 0 0 | + | 8 19 1 0 0 0 0 |
| − | 20 15 1 0 0 0 0 | + | 20 15 1 0 0 0 0 |
| − | 3 21 1 0 0 0 0 | + | 3 21 1 0 0 0 0 |
| − | 2 22 1 0 0 0 0 | + | 2 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 25 31 1 0 0 0 0 | + | 25 31 1 0 0 0 0 |
| − | 26 22 1 0 0 0 0 | + | 26 22 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 37 39 1 0 0 0 0 | + | 37 39 1 0 0 0 0 |
| − | 36 40 1 0 0 0 0 | + | 36 40 1 0 0 0 0 |
| − | 33 19 1 0 0 0 0 | + | 33 19 1 0 0 0 0 |
| − | 28 41 1 0 0 0 0 | + | 28 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 35 43 1 0 0 0 0 | + | 35 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 43 44 | + | M SAL 2 2 43 44 |
| − | M SBL 2 1 47 | + | M SBL 2 1 47 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 47 3.8509 -0.7865 | + | M SVB 2 47 3.8509 -0.7865 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 45 -3.091 -0.7666 | + | M SVB 1 45 -3.091 -0.7666 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FEAGL0002 | + | ID FL5FEAGL0002 |
| − | KNApSAcK_ID C00005308 | + | KNApSAcK_ID C00005308 |
| − | NAME 6-Hydroxykaempferol 3,6-diglucoside | + | NAME 6-Hydroxykaempferol 3,6-diglucoside |
| − | CAS_RN 142674-16-6 | + | CAS_RN 142674-16-6 |
| − | FORMULA C27H30O17 | + | FORMULA C27H30O17 |
| − | EXACTMASS 626.148299534 | + | EXACTMASS 626.148299534 |
| − | AVERAGEMASS 626.5169000000001 | + | AVERAGEMASS 626.5169000000001 |
| − | SMILES c(C(=C4O[C@@H](C(O)5)O[C@@H]([C@@H](C(O)5)O)CO)Oc(c3C4=O)cc(O)c(c3O)O[C@H](O2)[C@@H](O)[C@@H](O)[C@@H](O)C2CO)(c1)ccc(c1)O | + | SMILES c(C(=C4O[C@@H](C(O)5)O[C@@H]([C@@H](C(O)5)O)CO)Oc(c3C4=O)cc(O)c(c3O)O[C@H](O2)[C@@H](O)[C@@H](O)[C@@H](O)C2CO)(c1)ccc(c1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
-1.4492 0.5646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4492 -0.0778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8928 -0.3989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3365 -0.0778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3365 0.5646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8928 0.8858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2198 -0.3989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7761 -0.0778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7761 0.5646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2198 0.8858 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2198 -0.8998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3322 0.8857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8991 0.5583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4661 0.8857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4661 1.5403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8991 1.8677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3322 1.5403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0052 0.8857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2095 -0.5112 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0329 1.9366 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8928 -1.0411 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0052 -0.3988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9879 -1.0418 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-3.5708 -1.5925 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.9701 -1.3589 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.3905 -1.3526 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.8117 -0.9313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3372 -1.2087 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-4.6521 -0.9836 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9646 -2.1108 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6259 -1.9366 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6383 -0.2921 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.3375 -0.8132 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.9160 -0.6478 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4980 -0.8132 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.7988 -0.2921 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.2203 -0.4574 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.0796 -0.4418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2619 0.0171 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2329 -0.5428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3009 -0.7040 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1288 -0.1431 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6976 -1.1055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6976 -1.9305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
8 19 1 0 0 0 0
20 15 1 0 0 0 0
3 21 1 0 0 0 0
2 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
25 31 1 0 0 0 0
26 22 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
32 38 1 0 0 0 0
37 39 1 0 0 0 0
36 40 1 0 0 0 0
33 19 1 0 0 0 0
28 41 1 0 0 0 0
41 42 1 0 0 0 0
35 43 1 0 0 0 0
43 44 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 43 44
M SBL 2 1 47
M SMT 2 CH2OH
M SVB 2 47 3.8509 -0.7865
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 45
M SMT 1 CH2OH
M SVB 1 45 -3.091 -0.7666
S SKP 8
ID FL5FEAGL0002
KNApSAcK_ID C00005308
NAME 6-Hydroxykaempferol 3,6-diglucoside
CAS_RN 142674-16-6
FORMULA C27H30O17
EXACTMASS 626.148299534
AVERAGEMASS 626.5169000000001
SMILES c(C(=C4O[C@@H](C(O)5)O[C@@H]([C@@H](C(O)5)O)CO)Oc(c3C4=O)cc(O)c(c3O)O[C@H](O2)[C@@H](O)[C@@H](O)[C@@H](O)C2CO)(c1)ccc(c1)O
M END