Mol:FL5FEAGA0005
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 36 39 0 0 0 0 0 0 0 0999 V2000 | + | 36 39 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.2231 -4.8884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2231 -4.8884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8557 -4.7769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8557 -4.7769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0754 -4.1732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0754 -4.1732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6625 -3.6812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6625 -3.6812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0299 -3.7927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0299 -3.7927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1898 -4.3963 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1898 -4.3963 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8822 -3.0775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8822 -3.0775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4693 -2.5855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4693 -2.5855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1633 -2.6970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1633 -2.6970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3830 -3.3006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3830 -3.3006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3754 -2.9905 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3754 -2.9905 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5761 -2.2051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5761 -2.2051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3521 -1.5899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3521 -1.5899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7730 -1.0883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7730 -1.0883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4177 -1.2020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4177 -1.2020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6416 -1.8173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6416 -1.8173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2208 -2.3188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2208 -2.3188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8987 -1.8517 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8987 -1.8517 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3493 -0.5012 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.3493 -0.5012 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.9617 -1.1726 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.9617 -1.1726 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.7072 -0.9596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7072 -0.9596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4571 -1.1726 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.4571 -1.1726 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.8449 -0.5012 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.8449 -0.5012 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.0993 -0.7142 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.0993 -0.7142 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.6223 -0.6960 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6223 -0.6960 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4014 -0.1911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4014 -0.1911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6023 0.4040 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6023 0.4040 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7076 -4.0617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7076 -4.0617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7643 -5.4914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7643 -5.4914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9780 -6.2883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9780 -6.2883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8384 -0.7007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8384 -0.7007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5562 0.0745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5562 0.0745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3242 -5.3476 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3242 -5.3476 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0903 -5.9904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0903 -5.9904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2839 -1.5031 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2839 -1.5031 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4267 -2.4928 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4267 -2.4928 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 24 26 1 0 0 0 0 | + | 24 26 1 0 0 0 0 |
| − | 23 27 1 0 0 0 0 | + | 23 27 1 0 0 0 0 |
| − | 20 18 1 0 0 0 0 | + | 20 18 1 0 0 0 0 |
| − | 18 8 1 0 0 0 0 | + | 18 8 1 0 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 3 28 1 0 0 0 0 | + | 3 28 1 0 0 0 0 |
| − | 2 29 1 0 0 0 0 | + | 2 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 15 31 1 0 0 0 0 | + | 15 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 1 33 1 0 0 0 0 | + | 1 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 22 35 1 0 0 0 0 | + | 22 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 35 36 | + | M SAL 4 2 35 36 |
| − | M SBL 4 1 38 | + | M SBL 4 1 38 |
| − | M SMT 4 CH2OH | + | M SMT 4 CH2OH |
| − | M SVB 4 38 3.2839 -1.5031 | + | M SVB 4 38 3.2839 -1.5031 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 33 34 | + | M SAL 3 2 33 34 |
| − | M SBL 3 1 36 | + | M SBL 3 1 36 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 36 -2.9266 0.8188 | + | M SVB 3 36 -2.9266 0.8188 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 31 32 | + | M SAL 2 2 31 32 |
| − | M SBL 2 1 34 | + | M SBL 2 1 34 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 34 1.9127 1.503 | + | M SVB 2 34 1.9127 1.503 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 29 30 | + | M SAL 1 2 29 30 |
| − | M SBL 1 1 32 | + | M SBL 1 1 32 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 32 -3.2839 -0.3509 | + | M SVB 1 32 -3.2839 -0.3509 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FEAGA0005 | + | ID FL5FEAGA0005 |
| − | KNApSAcK_ID C00005334 | + | KNApSAcK_ID C00005334 |
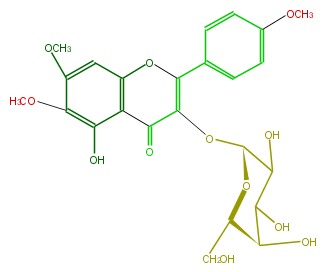
| − | NAME Mikanin 3-galactoside | + | NAME Mikanin 3-galactoside |
| − | CAS_RN 72945-44-9 | + | CAS_RN 72945-44-9 |
| − | FORMULA C24H26O12 | + | FORMULA C24H26O12 |
| − | EXACTMASS 506.142426296 | + | EXACTMASS 506.142426296 |
| − | AVERAGEMASS 506.45604 | + | AVERAGEMASS 506.45604 |
| − | SMILES OC([C@@H]1OC(=C(c(c4)ccc(c4)OC)2)C(=O)c(c(O)3)c(cc(c(OC)3)OC)O2)C([C@@H](O)[C@@H](CO)O1)O | + | SMILES OC([C@@H]1OC(=C(c(c4)ccc(c4)OC)2)C(=O)c(c(O)3)c(cc(c(OC)3)OC)O2)C([C@@H](O)[C@@H](CO)O1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
36 39 0 0 0 0 0 0 0 0999 V2000
0.2231 -4.8884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8557 -4.7769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0754 -4.1732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6625 -3.6812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0299 -3.7927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1898 -4.3963 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8822 -3.0775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4693 -2.5855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1633 -2.6970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3830 -3.3006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3754 -2.9905 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5761 -2.2051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3521 -1.5899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7730 -1.0883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4177 -1.2020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6416 -1.8173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2208 -2.3188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8987 -1.8517 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3493 -0.5012 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.9617 -1.1726 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.7072 -0.9596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4571 -1.1726 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.8449 -0.5012 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.0993 -0.7142 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.6223 -0.6960 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4014 -0.1911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6023 0.4040 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7076 -4.0617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7643 -5.4914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9780 -6.2883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8384 -0.7007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5562 0.0745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3242 -5.3476 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0903 -5.9904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2839 -1.5031 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4267 -2.4928 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
19 20 1 0 0 0 0
20 21 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
19 25 1 0 0 0 0
24 26 1 0 0 0 0
23 27 1 0 0 0 0
20 18 1 0 0 0 0
18 8 1 0 0 0 0
22 21 1 1 0 0 0
3 28 1 0 0 0 0
2 29 1 0 0 0 0
29 30 1 0 0 0 0
15 31 1 0 0 0 0
31 32 1 0 0 0 0
1 33 1 0 0 0 0
33 34 1 0 0 0 0
22 35 1 0 0 0 0
35 36 1 0 0 0 0
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 35 36
M SBL 4 1 38
M SMT 4 CH2OH
M SVB 4 38 3.2839 -1.5031
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 33 34
M SBL 3 1 36
M SMT 3 OCH3
M SVB 3 36 -2.9266 0.8188
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 31 32
M SBL 2 1 34
M SMT 2 OCH3
M SVB 2 34 1.9127 1.503
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 29 30
M SBL 1 1 32
M SMT 1 OCH3
M SVB 1 32 -3.2839 -0.3509
S SKP 8
ID FL5FEAGA0005
KNApSAcK_ID C00005334
NAME Mikanin 3-galactoside
CAS_RN 72945-44-9
FORMULA C24H26O12
EXACTMASS 506.142426296
AVERAGEMASS 506.45604
SMILES OC([C@@H]1OC(=C(c(c4)ccc(c4)OC)2)C(=O)c(c(O)3)c(cc(c(OC)3)OC)O2)C([C@@H](O)[C@@H](CO)O1)O
M END