Mol:FL5FCAGS0006
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 53 58 0 0 0 0 0 0 0 0999 V2000 | + | 53 58 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.8697 1.9166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8697 1.9166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8697 1.1070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8697 1.1070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1686 0.7022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1686 0.7022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4675 1.1070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4675 1.1070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4675 1.9166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4675 1.9166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1686 2.3214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1686 2.3214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7664 0.7022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7664 0.7022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0652 1.1070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0652 1.1070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0652 1.9166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0652 1.9166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7664 2.3214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7664 2.3214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7664 0.0710 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7664 0.0710 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6356 2.3213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6356 2.3213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3502 1.9087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3502 1.9087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0648 2.3213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0648 2.3213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0648 3.1463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0648 3.1463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3502 3.5589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3502 3.5589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6356 3.1463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6356 3.1463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7629 0.6361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7629 0.6361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1686 -0.1071 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1686 -0.1071 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6932 -0.7869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6932 -0.7869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2701 -1.5196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2701 -1.5196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0836 -1.2871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0836 -1.2871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9022 -1.5196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9022 -1.5196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3254 -0.7869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3254 -0.7869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5117 -1.0193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5117 -1.0193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9729 -0.9238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9729 -0.9238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7901 1.1852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7901 1.1852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3670 0.4525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3670 0.4525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1805 0.6850 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1805 0.6850 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9991 0.4525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9991 0.4525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4223 1.1852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4223 1.1852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6087 0.9528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6087 0.9528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1308 1.1164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1308 1.1164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9244 1.4079 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9244 1.4079 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3412 2.0207 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3412 2.0207 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7762 0.5542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7762 0.5542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2130 0.0473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2130 0.0473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6214 -1.4017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6214 -1.4017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5930 -1.9000 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5930 -1.9000 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9022 -2.2336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9022 -2.2336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8637 3.6076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8637 3.6076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1536 -2.1609 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1536 -2.1609 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2695 -2.8936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2695 -2.8936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5441 -2.6611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5441 -2.6611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3627 -2.8936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3627 -2.8936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7858 -2.1609 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7858 -2.1609 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9721 -2.3933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9721 -2.3933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4507 -2.2069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4507 -2.2069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7979 -2.7339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7979 -2.7339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0742 -3.2534 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0742 -3.2534 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3627 -3.6076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3627 -3.6076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6952 2.3933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6952 2.3933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3254 3.4847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3254 3.4847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 8 18 1 0 0 0 0 | + | 8 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 1 0 0 0 | + | 28 29 1 1 0 0 0 |
| − | 29 30 1 1 0 0 0 | + | 29 30 1 1 0 0 0 |
| − | 31 30 1 1 0 0 0 | + | 31 30 1 1 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 27 1 0 0 0 0 | + | 32 27 1 0 0 0 0 |
| − | 27 33 1 0 0 0 0 | + | 27 33 1 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 31 35 1 0 0 0 0 | + | 31 35 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 24 37 1 0 0 0 0 | + | 24 37 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 21 38 1 0 0 0 0 | + | 21 38 1 0 0 0 0 |
| − | 22 39 1 0 0 0 0 | + | 22 39 1 0 0 0 0 |
| − | 23 40 1 0 0 0 0 | + | 23 40 1 0 0 0 0 |
| − | 28 18 1 0 0 0 0 | + | 28 18 1 0 0 0 0 |
| − | 41 15 1 0 0 0 0 | + | 41 15 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 44 1 1 0 0 0 | + | 43 44 1 1 0 0 0 |
| − | 44 45 1 1 0 0 0 | + | 44 45 1 1 0 0 0 |
| − | 46 45 1 1 0 0 0 | + | 46 45 1 1 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 42 1 0 0 0 0 | + | 47 42 1 0 0 0 0 |
| − | 42 48 1 0 0 0 0 | + | 42 48 1 0 0 0 0 |
| − | 43 49 1 0 0 0 0 | + | 43 49 1 0 0 0 0 |
| − | 44 50 1 0 0 0 0 | + | 44 50 1 0 0 0 0 |
| − | 45 51 1 0 0 0 0 | + | 45 51 1 0 0 0 0 |
| − | 46 38 1 0 0 0 0 | + | 46 38 1 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 1 52 1 0 0 0 0 | + | 1 52 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 52 53 | + | M SAL 1 2 52 53 |
| − | M SBL 1 1 58 | + | M SBL 1 1 58 |
| − | M SMT 1 ^ OCH3 | + | M SMT 1 ^ OCH3 |
| − | M SBV 1 58 0.8255 -0.4766 | + | M SBV 1 58 0.8255 -0.4766 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FCAGS0006 | + | ID FL5FCAGS0006 |
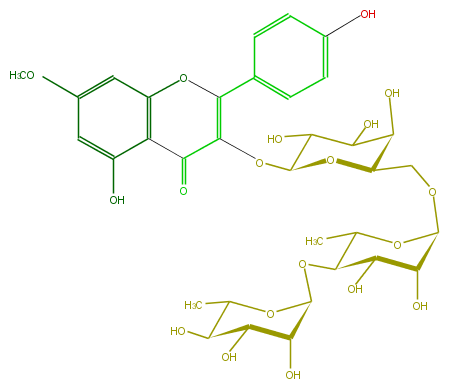
| − | FORMULA C34H42O19 | + | FORMULA C34H42O19 |
| − | EXACTMASS 754.2320291619999 | + | EXACTMASS 754.2320291619999 |
| − | AVERAGEMASS 754.68588 | + | AVERAGEMASS 754.68588 |
| − | SMILES C(O6)(C(O)C(O)C(C6C)OC(C5O)OC(C)C(C5O)O)OCC(C(O)4)OC(C(C4O)O)OC(=C2c(c3)ccc(c3)O)C(c(c(O2)1)c(cc(OC)c1)O)=O | + | SMILES C(O6)(C(O)C(O)C(C6C)OC(C5O)OC(C)C(C5O)O)OCC(C(O)4)OC(C(C4O)O)OC(=C2c(c3)ccc(c3)O)C(c(c(O2)1)c(cc(OC)c1)O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
53 58 0 0 0 0 0 0 0 0999 V2000
-2.8697 1.9166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8697 1.1070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1686 0.7022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4675 1.1070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4675 1.9166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1686 2.3214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7664 0.7022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0652 1.1070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0652 1.9166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7664 2.3214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7664 0.0710 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6356 2.3213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3502 1.9087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0648 2.3213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0648 3.1463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3502 3.5589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6356 3.1463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7629 0.6361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1686 -0.1071 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6932 -0.7869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2701 -1.5196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0836 -1.2871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9022 -1.5196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3254 -0.7869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5117 -1.0193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9729 -0.9238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7901 1.1852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3670 0.4525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1805 0.6850 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9991 0.4525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4223 1.1852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6087 0.9528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1308 1.1164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9244 1.4079 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3412 2.0207 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7762 0.5542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2130 0.0473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6214 -1.4017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5930 -1.9000 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9022 -2.2336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8637 3.6076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1536 -2.1609 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2695 -2.8936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5441 -2.6611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3627 -2.8936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7858 -2.1609 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9721 -2.3933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4507 -2.2069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7979 -2.7339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0742 -3.2534 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3627 -3.6076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6952 2.3933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3254 3.4847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
8 18 1 0 0 0 0
3 19 1 0 0 0 0
20 21 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
27 28 1 0 0 0 0
28 29 1 1 0 0 0
29 30 1 1 0 0 0
31 30 1 1 0 0 0
31 32 1 0 0 0 0
32 27 1 0 0 0 0
27 33 1 0 0 0 0
32 34 1 0 0 0 0
31 35 1 0 0 0 0
30 36 1 0 0 0 0
24 37 1 0 0 0 0
36 37 1 0 0 0 0
21 38 1 0 0 0 0
22 39 1 0 0 0 0
23 40 1 0 0 0 0
28 18 1 0 0 0 0
41 15 1 0 0 0 0
42 43 1 0 0 0 0
43 44 1 1 0 0 0
44 45 1 1 0 0 0
46 45 1 1 0 0 0
46 47 1 0 0 0 0
47 42 1 0 0 0 0
42 48 1 0 0 0 0
43 49 1 0 0 0 0
44 50 1 0 0 0 0
45 51 1 0 0 0 0
46 38 1 0 0 0 0
52 53 1 0 0 0 0
1 52 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 52 53
M SBL 1 1 58
M SMT 1 ^ OCH3
M SBV 1 58 0.8255 -0.4766
S SKP 5
ID FL5FCAGS0006
FORMULA C34H42O19
EXACTMASS 754.2320291619999
AVERAGEMASS 754.68588
SMILES C(O6)(C(O)C(O)C(C6C)OC(C5O)OC(C)C(C5O)O)OCC(C(O)4)OC(C(C4O)O)OC(=C2c(c3)ccc(c3)O)C(c(c(O2)1)c(cc(OC)c1)O)=O
M END