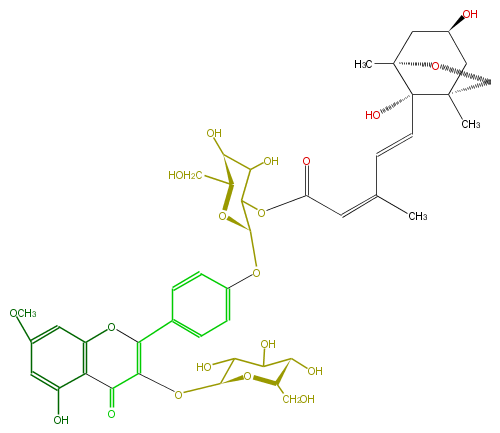
Mol:FL5FCAGL0010
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 63 69 0 0 0 0 0 0 0 0999 V2000 | + | 63 69 0 0 0 0 0 0 0 0999 V2000 |
| − | -4.5651 -2.6021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5651 -2.6021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5651 -3.2444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5651 -3.2444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0088 -3.5656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0088 -3.5656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4525 -3.2444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4525 -3.2444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4525 -2.6021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4525 -2.6021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0088 -2.2809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0088 -2.2809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8962 -3.5656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8962 -3.5656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3399 -3.2444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3399 -3.2444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3399 -2.6021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3399 -2.6021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8962 -2.2809 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8962 -2.2809 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8962 -4.0664 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8962 -4.0664 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6395 -2.1572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6395 -2.1572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0725 -2.4846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0725 -2.4846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5055 -2.1572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5055 -2.1572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5055 -1.5026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5055 -1.5026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0725 -1.1752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0725 -1.1752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6395 -1.5026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6395 -1.5026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0088 -4.2077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0088 -4.2077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1013 -1.1522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1013 -1.1522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5126 -3.6746 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5126 -3.6746 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3539 -2.9432 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -0.3539 -2.9432 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.6547 -3.4642 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.6547 -3.4642 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.0762 -3.2989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0762 -3.2989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5057 -3.4642 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.5057 -3.4642 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.8066 -2.9432 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.8066 -2.9432 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.2281 -3.1084 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.2281 -3.1084 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.9126 -3.0929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9126 -3.0929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2697 -2.6339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2697 -2.6339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2407 -3.1938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2407 -3.1938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5993 0.0245 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5993 0.0245 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0313 -0.3034 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.0313 -0.3034 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.2116 0.3271 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -0.2116 0.3271 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.0313 0.9615 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -0.0313 0.9615 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.5993 1.2894 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.5993 1.2894 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.4191 0.6588 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -0.4191 0.6588 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -0.8555 1.7331 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8555 1.7331 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3231 1.1661 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3231 1.1661 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2061 0.0860 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2061 0.0860 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1328 0.4310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1328 0.4310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1328 1.1522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1328 1.1522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8584 0.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8584 0.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5836 0.4307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5836 0.4307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3088 0.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3088 0.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5836 1.2681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5836 1.2681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3088 1.6868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3088 1.6868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3088 2.5242 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 3.3088 2.5242 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 4.0562 2.5242 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 4.0562 2.5242 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 4.4299 3.1714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4299 3.1714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0562 3.8186 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 4.0562 3.8186 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.3088 3.8186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3088 3.8186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9351 3.1714 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 2.9351 3.1714 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 2.5887 2.1084 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5887 2.1084 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9224 2.7811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9224 2.7811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8086 3.1317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8086 3.1317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4453 4.2077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4453 4.2077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4264 3.1714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4264 3.1714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4071 1.9164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4071 1.9164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7053 -3.7566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7053 -3.7566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7053 -4.5816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7053 -4.5816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0953 0.8771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0953 0.8771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8372 0.2065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8372 0.2065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9224 -1.9833 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9224 -1.9833 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4224 -1.1173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4224 -1.1173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 22 20 1 0 0 0 0 | + | 22 20 1 0 0 0 0 |
| − | 31 30 1 1 0 0 0 | + | 31 30 1 1 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 35 30 1 1 0 0 0 | + | 35 30 1 1 0 0 0 |
| − | 34 36 1 0 0 0 0 | + | 34 36 1 0 0 0 0 |
| − | 33 37 1 0 0 0 0 | + | 33 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 19 31 1 0 0 0 0 | + | 19 31 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 39 41 1 0 0 0 0 | + | 39 41 1 0 0 0 0 |
| − | 41 42 2 0 0 0 0 | + | 41 42 2 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 42 44 1 0 0 0 0 | + | 42 44 1 0 0 0 0 |
| − | 44 45 2 0 0 0 0 | + | 44 45 2 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 51 46 1 0 0 0 0 | + | 51 46 1 0 0 0 0 |
| − | 46 52 1 6 0 0 0 | + | 46 52 1 6 0 0 0 |
| − | 47 53 1 6 0 0 0 | + | 47 53 1 6 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 51 54 1 6 0 0 0 | + | 51 54 1 6 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 49 55 1 1 0 0 0 | + | 49 55 1 1 0 0 0 |
| − | 51 56 1 0 0 0 0 | + | 51 56 1 0 0 0 0 |
| − | 47 57 1 0 0 0 0 | + | 47 57 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 24 58 1 0 0 0 0 | + | 24 58 1 0 0 0 0 |
| − | 58 59 1 0 0 0 0 | + | 58 59 1 0 0 0 0 |
| − | 35 60 1 0 0 0 0 | + | 35 60 1 0 0 0 0 |
| − | 60 61 1 0 0 0 0 | + | 60 61 1 0 0 0 0 |
| − | 1 62 1 0 0 0 0 | + | 1 62 1 0 0 0 0 |
| − | 62 63 1 0 0 0 0 | + | 62 63 1 0 0 0 0 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 60 61 | + | M SAL 3 2 60 61 |
| − | M SBL 3 1 66 | + | M SBL 3 1 66 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SVB 3 66 -0.9397 1.1424 | + | M SVB 3 66 -0.9397 1.1424 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 58 59 | + | M SAL 2 2 58 59 |
| − | M SBL 2 1 64 | + | M SBL 2 1 64 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 64 0.8587 -3.4375 | + | M SVB 2 64 0.8587 -3.4375 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 62 63 | + | M SAL 1 2 62 63 |
| − | M SBL 1 1 68 | + | M SBL 1 1 68 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 68 -4.9224 -1.9833 | + | M SVB 1 68 -4.9224 -1.9833 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FCAGL0010 | + | ID FL5FCAGL0010 |
| − | KNApSAcK_ID C00005930 | + | KNApSAcK_ID C00005930 |
| − | NAME Rhamnocitrin 3-glucoside-4'-(2''-dihydrophaseoylglucoside) | + | NAME Rhamnocitrin 3-glucoside-4'-(2''-dihydrophaseoylglucoside) |
| − | CAS_RN 138505-45-0 | + | CAS_RN 138505-45-0 |
| − | FORMULA C43H52O20 | + | FORMULA C43H52O20 |
| − | EXACTMASS 888.305194104 | + | EXACTMASS 888.305194104 |
| − | AVERAGEMASS 888.8609799999999 | + | AVERAGEMASS 888.8609799999999 |
| − | SMILES O[C@]([C@@](C)67)([C@](C[C@@H](C7)O)(CO6)C)C=CC(=CC(OC([C@H]1Oc(c5)ccc(c5)C(=C2O[C@H](O4)C(C([C@H]([C@H]4CO)O)O)O)Oc(c3)c(c(O)cc3OC)C2=O)C([C@@H]([C@@H](O1)CO)O)O)=O)C | + | SMILES O[C@]([C@@](C)67)([C@](C[C@@H](C7)O)(CO6)C)C=CC(=CC(OC([C@H]1Oc(c5)ccc(c5)C(=C2O[C@H](O4)C(C([C@H]([C@H]4CO)O)O)O)Oc(c3)c(c(O)cc3OC)C2=O)C([C@@H]([C@@H](O1)CO)O)O)=O)C |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
63 69 0 0 0 0 0 0 0 0999 V2000
-4.5651 -2.6021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5651 -3.2444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0088 -3.5656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4525 -3.2444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4525 -2.6021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0088 -2.2809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8962 -3.5656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3399 -3.2444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3399 -2.6021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8962 -2.2809 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8962 -4.0664 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6395 -2.1572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0725 -2.4846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5055 -2.1572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5055 -1.5026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0725 -1.1752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6395 -1.5026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0088 -4.2077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1013 -1.1522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5126 -3.6746 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3539 -2.9432 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.6547 -3.4642 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.0762 -3.2989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5057 -3.4642 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.8066 -2.9432 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.2281 -3.1084 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.9126 -3.0929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2697 -2.6339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2407 -3.1938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5993 0.0245 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0313 -0.3034 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.2116 0.3271 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.0313 0.9615 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.5993 1.2894 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.4191 0.6588 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-0.8555 1.7331 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3231 1.1661 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2061 0.0860 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1328 0.4310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1328 1.1522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8584 0.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5836 0.4307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3088 0.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5836 1.2681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3088 1.6868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3088 2.5242 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
4.0562 2.5242 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
4.4299 3.1714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0562 3.8186 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.3088 3.8186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9351 3.1714 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
2.5887 2.1084 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9224 2.7811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8086 3.1317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4453 4.2077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4264 3.1714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4071 1.9164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7053 -3.7566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7053 -4.5816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0953 0.8771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8372 0.2065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9224 -1.9833 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.4224 -1.1173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
4 3 1 0 0 0 0
20 8 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
22 20 1 0 0 0 0
31 30 1 1 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 1 0 0 0
35 30 1 1 0 0 0
34 36 1 0 0 0 0
33 37 1 0 0 0 0
32 38 1 0 0 0 0
19 31 1 0 0 0 0
39 40 2 0 0 0 0
39 41 1 0 0 0 0
41 42 2 0 0 0 0
42 43 1 0 0 0 0
42 44 1 0 0 0 0
44 45 2 0 0 0 0
45 46 1 0 0 0 0
46 47 1 0 0 0 0
48 49 1 0 0 0 0
49 50 1 0 0 0 0
50 51 1 0 0 0 0
51 46 1 0 0 0 0
46 52 1 6 0 0 0
47 53 1 6 0 0 0
53 54 1 0 0 0 0
51 54 1 6 0 0 0
47 48 1 0 0 0 0
49 55 1 1 0 0 0
51 56 1 0 0 0 0
47 57 1 0 0 0 0
38 39 1 0 0 0 0
24 58 1 0 0 0 0
58 59 1 0 0 0 0
35 60 1 0 0 0 0
60 61 1 0 0 0 0
1 62 1 0 0 0 0
62 63 1 0 0 0 0
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 60 61
M SBL 3 1 66
M SMT 3 CH2OH
M SVB 3 66 -0.9397 1.1424
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 58 59
M SBL 2 1 64
M SMT 2 CH2OH
M SVB 2 64 0.8587 -3.4375
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 62 63
M SBL 1 1 68
M SMT 1 OCH3
M SVB 1 68 -4.9224 -1.9833
S SKP 8
ID FL5FCAGL0010
KNApSAcK_ID C00005930
NAME Rhamnocitrin 3-glucoside-4'-(2''-dihydrophaseoylglucoside)
CAS_RN 138505-45-0
FORMULA C43H52O20
EXACTMASS 888.305194104
AVERAGEMASS 888.8609799999999
SMILES O[C@]([C@@](C)67)([C@](C[C@@H](C7)O)(CO6)C)C=CC(=CC(OC([C@H]1Oc(c5)ccc(c5)C(=C2O[C@H](O4)C(C([C@H]([C@H]4CO)O)O)O)Oc(c3)c(c(O)cc3OC)C2=O)C([C@@H]([C@@H](O1)CO)O)O)=O)C
M END