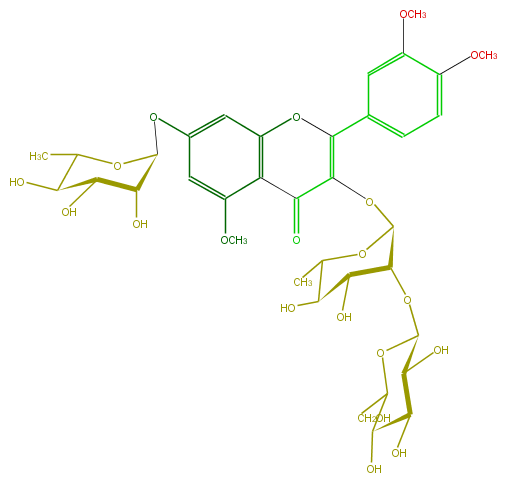
Mol:FL5FBFGS0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 56 61 0 0 0 0 0 0 0 0999 V2000 | + | 56 61 0 0 0 0 0 0 0 0999 V2000 |
| − | 2.7567 1.8729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7567 1.8729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0422 2.2854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0422 2.2854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0422 3.1104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0422 3.1104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7567 3.5229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7567 3.5229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4710 3.1104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4710 3.1104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4710 2.2854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4710 2.2854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3277 1.8729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3277 1.8729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6133 2.2854 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6133 2.2854 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1011 1.8729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1011 1.8729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1011 1.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1011 1.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6133 0.6355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6133 0.6355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3277 1.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3277 1.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8156 2.2854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8156 2.2854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5300 1.8729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5300 1.8729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5300 1.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5300 1.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8156 0.6355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8156 0.6355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6133 -0.1800 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6133 -0.1800 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2445 2.2854 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2445 2.2854 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0891 0.5828 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0891 0.5828 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7589 1.5018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7589 1.5018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1716 0.7871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1716 0.7871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3781 1.0139 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3781 1.0139 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5799 0.8046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5799 0.8046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1671 1.5193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1671 1.5193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9606 1.2926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9606 1.2926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5799 0.1497 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5799 0.1497 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0085 0.3835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0085 0.3835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8988 0.9819 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8988 0.9819 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4163 1.5018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4163 1.5018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1239 -0.6125 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1239 -0.6125 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0519 -1.4347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0519 -1.4347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6751 -0.8938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6751 -0.8938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4871 -0.7462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4871 -0.7462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5591 0.0760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5591 0.0760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9357 -0.4649 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9357 -0.4649 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8420 -1.3831 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8420 -1.3831 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4998 -1.7154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4998 -1.7154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5195 -1.5367 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5195 -1.5367 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6384 -1.0199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6384 -1.0199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1362 -4.0323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1362 -4.0323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8870 -3.6899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8870 -3.6899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7551 -2.8753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7551 -2.8753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0568 -2.1072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0568 -2.1072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3059 -2.4494 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3059 -2.4494 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4377 -3.2642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4377 -3.2642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1102 -4.7604 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1102 -4.7604 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5956 -4.4480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5956 -4.4480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4444 -2.3764 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4444 -2.3764 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1855 3.5229 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1855 3.5229 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8988 3.1111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8988 3.1111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8156 -0.1895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8156 -0.1895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5300 -0.6020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5300 -0.6020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8183 -3.7443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8183 -3.7443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4966 -4.1956 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4966 -4.1956 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7567 4.3479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7567 4.3479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0422 4.7604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0422 4.7604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 23 26 1 0 0 0 0 | + | 23 26 1 0 0 0 0 |
| − | 22 27 1 0 0 0 0 | + | 22 27 1 0 0 0 0 |
| − | 21 28 1 0 0 0 0 | + | 21 28 1 0 0 0 0 |
| − | 20 29 1 0 0 0 0 | + | 20 29 1 0 0 0 0 |
| − | 24 18 1 0 0 0 0 | + | 24 18 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 33 36 1 0 0 0 0 | + | 33 36 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 31 38 1 0 0 0 0 | + | 31 38 1 0 0 0 0 |
| − | 30 39 1 0 0 0 0 | + | 30 39 1 0 0 0 0 |
| − | 34 19 1 0 0 0 0 | + | 34 19 1 0 0 0 0 |
| − | 40 41 1 1 0 0 0 | + | 40 41 1 1 0 0 0 |
| − | 41 42 1 1 0 0 0 | + | 41 42 1 1 0 0 0 |
| − | 43 42 1 1 0 0 0 | + | 43 42 1 1 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 40 1 0 0 0 0 | + | 45 40 1 0 0 0 0 |
| − | 40 46 1 0 0 0 0 | + | 40 46 1 0 0 0 0 |
| − | 41 47 1 0 0 0 0 | + | 41 47 1 0 0 0 0 |
| − | 42 48 1 0 0 0 0 | + | 42 48 1 0 0 0 0 |
| − | 43 36 1 0 0 0 0 | + | 43 36 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 5 49 1 0 0 0 0 | + | 5 49 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 16 51 1 0 0 0 0 | + | 16 51 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 45 53 1 0 0 0 0 | + | 45 53 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 4 55 1 0 0 0 0 | + | 4 55 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 49 50 | + | M SAL 1 2 49 50 |
| − | M SBL 1 1 55 | + | M SBL 1 1 55 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 55 -0.7145 -0.4125 | + | M SBV 1 55 -0.7145 -0.4125 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 51 52 | + | M SAL 2 2 51 52 |
| − | M SBL 2 1 57 | + | M SBL 2 1 57 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SBV 2 57 0.0000 0.8250 | + | M SBV 2 57 0.0000 0.8250 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 53 54 | + | M SAL 3 2 53 54 |
| − | M SBL 3 1 59 | + | M SBL 3 1 59 |
| − | M SMT 3 ^ CH2OH | + | M SMT 3 ^ CH2OH |
| − | M SBV 3 59 0.6193 0.4801 | + | M SBV 3 59 0.6193 0.4801 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 55 56 | + | M SAL 4 2 55 56 |
| − | M SBL 4 1 61 | + | M SBL 4 1 61 |
| − | M SMT 4 OCH3 | + | M SMT 4 OCH3 |
| − | M SBV 4 61 0.0000 -0.8250 | + | M SBV 4 61 0.0000 -0.8250 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FBFGS0001 | + | ID FL5FBFGS0001 |
| − | FORMULA C36H46O20 | + | FORMULA C36H46O20 |
| − | EXACTMASS 798.2582439119999 | + | EXACTMASS 798.2582439119999 |
| − | AVERAGEMASS 798.7384400000001 | + | AVERAGEMASS 798.7384400000001 |
| − | SMILES c(c6OC)(ccc(c6)C(O1)=C(OC(O5)C(C(C(O)C(C)5)O)OC(O4)C(C(O)C(C4CO)O)O)C(c(c3OC)c1cc(c3)OC(C(O)2)OC(C)C(O)C2O)=O)OC | + | SMILES c(c6OC)(ccc(c6)C(O1)=C(OC(O5)C(C(C(O)C(C)5)O)OC(O4)C(C(O)C(C4CO)O)O)C(c(c3OC)c1cc(c3)OC(C(O)2)OC(C)C(O)C2O)=O)OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
56 61 0 0 0 0 0 0 0 0999 V2000
2.7567 1.8729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0422 2.2854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0422 3.1104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7567 3.5229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4710 3.1104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4710 2.2854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3277 1.8729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6133 2.2854 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1011 1.8729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1011 1.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6133 0.6355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3277 1.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8156 2.2854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5300 1.8729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5300 1.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8156 0.6355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6133 -0.1800 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2445 2.2854 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0891 0.5828 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7589 1.5018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1716 0.7871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3781 1.0139 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5799 0.8046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1671 1.5193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9606 1.2926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5799 0.1497 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0085 0.3835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8988 0.9819 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4163 1.5018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1239 -0.6125 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0519 -1.4347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6751 -0.8938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4871 -0.7462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5591 0.0760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9357 -0.4649 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8420 -1.3831 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4998 -1.7154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5195 -1.5367 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6384 -1.0199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1362 -4.0323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8870 -3.6899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7551 -2.8753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0568 -2.1072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3059 -2.4494 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4377 -3.2642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1102 -4.7604 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5956 -4.4480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4444 -2.3764 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1855 3.5229 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8988 3.1111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8156 -0.1895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5300 -0.6020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8183 -3.7443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4966 -4.1956 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7567 4.3479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0422 4.7604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
20 21 1 0 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
23 26 1 0 0 0 0
22 27 1 0 0 0 0
21 28 1 0 0 0 0
20 29 1 0 0 0 0
24 18 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
33 36 1 0 0 0 0
32 37 1 0 0 0 0
31 38 1 0 0 0 0
30 39 1 0 0 0 0
34 19 1 0 0 0 0
40 41 1 1 0 0 0
41 42 1 1 0 0 0
43 42 1 1 0 0 0
43 44 1 0 0 0 0
44 45 1 0 0 0 0
45 40 1 0 0 0 0
40 46 1 0 0 0 0
41 47 1 0 0 0 0
42 48 1 0 0 0 0
43 36 1 0 0 0 0
49 50 1 0 0 0 0
5 49 1 0 0 0 0
51 52 1 0 0 0 0
16 51 1 0 0 0 0
53 54 1 0 0 0 0
45 53 1 0 0 0 0
55 56 1 0 0 0 0
4 55 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 49 50
M SBL 1 1 55
M SMT 1 OCH3
M SBV 1 55 -0.7145 -0.4125
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 51 52
M SBL 2 1 57
M SMT 2 OCH3
M SBV 2 57 0.0000 0.8250
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 53 54
M SBL 3 1 59
M SMT 3 ^ CH2OH
M SBV 3 59 0.6193 0.4801
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 55 56
M SBL 4 1 61
M SMT 4 OCH3
M SBV 4 61 0.0000 -0.8250
S SKP 5
ID FL5FBFGS0001
FORMULA C36H46O20
EXACTMASS 798.2582439119999
AVERAGEMASS 798.7384400000001
SMILES c(c6OC)(ccc(c6)C(O1)=C(OC(O5)C(C(C(O)C(C)5)O)OC(O4)C(C(O)C(C4CO)O)O)C(c(c3OC)c1cc(c3)OC(C(O)2)OC(C)C(O)C2O)=O)OC
M END