Mol:FL5FAJGA0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 46 50 0 0 0 0 0 0 0 0999 V2000 | + | 46 50 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.7614 1.3864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7614 1.3864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7614 0.5614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7614 0.5614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0468 0.1488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0468 0.1488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3323 0.5614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3323 0.5614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3323 1.3864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3323 1.3864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0468 1.7990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0468 1.7990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6177 0.1488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6177 0.1488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9032 0.5614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9032 0.5614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9032 1.3864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9032 1.3864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6177 1.7990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6177 1.7990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6177 -0.4944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6177 -0.4944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1889 1.7988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1889 1.7988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5394 1.3783 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5394 1.3783 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2675 1.7988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2675 1.7988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2675 2.6398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2675 2.6398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5394 3.0602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5394 3.0602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1889 2.6398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1889 2.6398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4756 1.7988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4756 1.7988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3970 0.0619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3970 0.0619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5407 3.6932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5407 3.6932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0468 -0.6759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0468 -0.6759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9955 1.3784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9955 1.3784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0749 -1.2016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0749 -1.2016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5763 -0.7038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5763 -0.7038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4078 -1.5785 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4078 -1.5785 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6322 -2.3684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6322 -2.3684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0749 -2.7769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0749 -2.7769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1494 -1.9916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1494 -1.9916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0628 -0.4045 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0628 -0.4045 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2883 -2.3368 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2883 -2.3368 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7102 -2.6065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7102 -2.6065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5343 -1.9874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5343 -1.9874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2521 -2.4510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2521 -2.4510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8513 -1.9254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8513 -1.9254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5225 -1.6729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5225 -1.6729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8547 -1.3614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8547 -1.3614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3612 -1.9061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3612 -1.9061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4492 -2.6200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4492 -2.6200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3543 -2.1447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3543 -2.1447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9032 -2.3811 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9032 -2.3811 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5914 -3.1634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5914 -3.1634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3263 -3.6932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3263 -3.6932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4322 -1.0528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4322 -1.0528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4756 -1.2368 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4756 -1.2368 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9955 3.0600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9955 3.0600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9133 2.5302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9133 2.5302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 8 19 1 0 0 0 0 | + | 8 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 3 21 1 0 0 0 0 | + | 3 21 1 0 0 0 0 |
| − | 14 22 1 0 0 0 0 | + | 14 22 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 28 30 1 0 0 0 0 | + | 28 30 1 0 0 0 0 |
| − | 27 31 1 0 0 0 0 | + | 27 31 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 35 40 1 0 0 0 0 | + | 35 40 1 0 0 0 0 |
| − | 32 31 1 0 0 0 0 | + | 32 31 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 26 41 1 0 0 0 0 | + | 26 41 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 36 43 1 0 0 0 0 | + | 36 43 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 15 45 1 0 0 0 0 | + | 15 45 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 46 | + | M SBL 1 1 46 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 46 -0.0408 0.7950 | + | M SBV 1 46 -0.0408 0.7950 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 43 44 | + | M SAL 2 2 43 44 |
| − | M SBL 2 1 48 | + | M SBL 2 1 48 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 48 -0.5775 -0.3086 | + | M SBV 2 48 -0.5775 -0.3086 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 45 46 | + | M SAL 3 2 45 46 |
| − | M SBL 3 1 50 | + | M SBL 3 1 50 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SBV 3 50 -0.7280 -0.4203 | + | M SBV 3 50 -0.7280 -0.4203 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAJGA0001 | + | ID FL5FAJGA0001 |
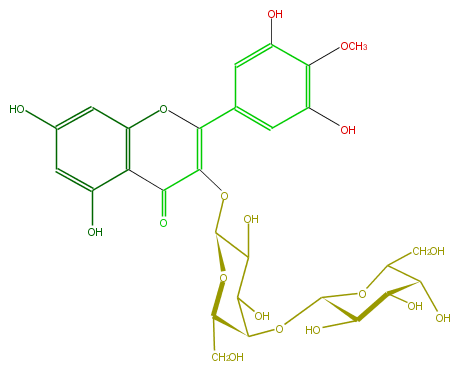
| − | FORMULA C28H32O18 | + | FORMULA C28H32O18 |
| − | EXACTMASS 656.1588642199999 | + | EXACTMASS 656.1588642199999 |
| − | AVERAGEMASS 656.54288 | + | AVERAGEMASS 656.54288 |
| − | SMILES OC(C1OC(C5O)C(CO)OC(C5O)OC(C4=O)=C(Oc(c34)cc(cc(O)3)O)c(c2)cc(c(c(O)2)OC)O)C(C(O)C(O1)CO)O | + | SMILES OC(C1OC(C5O)C(CO)OC(C5O)OC(C4=O)=C(Oc(c34)cc(cc(O)3)O)c(c2)cc(c(c(O)2)OC)O)C(C(O)C(O1)CO)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
46 50 0 0 0 0 0 0 0 0999 V2000
-3.7614 1.3864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7614 0.5614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0468 0.1488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3323 0.5614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3323 1.3864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0468 1.7990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6177 0.1488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9032 0.5614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9032 1.3864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6177 1.7990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6177 -0.4944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1889 1.7988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5394 1.3783 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2675 1.7988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2675 2.6398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5394 3.0602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1889 2.6398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4756 1.7988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3970 0.0619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5407 3.6932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0468 -0.6759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9955 1.3784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0749 -1.2016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5763 -0.7038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4078 -1.5785 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6322 -2.3684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0749 -2.7769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1494 -1.9916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0628 -0.4045 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2883 -2.3368 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7102 -2.6065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5343 -1.9874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2521 -2.4510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8513 -1.9254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5225 -1.6729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8547 -1.3614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3612 -1.9061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4492 -2.6200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3543 -2.1447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9032 -2.3811 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5914 -3.1634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3263 -3.6932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4322 -1.0528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4756 -1.2368 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9955 3.0600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9133 2.5302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
8 19 1 0 0 0 0
16 20 1 0 0 0 0
3 21 1 0 0 0 0
14 22 1 0 0 0 0
23 24 1 0 0 0 0
24 25 1 1 0 0 0
25 26 1 1 0 0 0
27 26 1 1 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
23 29 1 0 0 0 0
28 30 1 0 0 0 0
27 31 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
33 38 1 0 0 0 0
34 39 1 0 0 0 0
35 40 1 0 0 0 0
32 31 1 0 0 0 0
24 19 1 0 0 0 0
41 42 1 0 0 0 0
26 41 1 0 0 0 0
43 44 1 0 0 0 0
36 43 1 0 0 0 0
45 46 1 0 0 0 0
15 45 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 46
M SMT 1 CH2OH
M SBV 1 46 -0.0408 0.7950
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 43 44
M SBL 2 1 48
M SMT 2 CH2OH
M SBV 2 48 -0.5775 -0.3086
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 45 46
M SBL 3 1 50
M SMT 3 OCH3
M SBV 3 50 -0.7280 -0.4203
S SKP 5
ID FL5FAJGA0001
FORMULA C28H32O18
EXACTMASS 656.1588642199999
AVERAGEMASS 656.54288
SMILES OC(C1OC(C5O)C(CO)OC(C5O)OC(C4=O)=C(Oc(c34)cc(cc(O)3)O)c(c2)cc(c(c(O)2)OC)O)C(C(O)C(O1)CO)O
M END