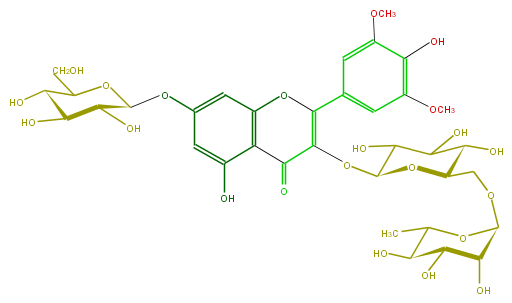
Mol:FL5FAIGL0003
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 57 62 0 0 0 0 0 0 0 0999 V2000 | + | 57 62 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.2813 0.3633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2813 0.3633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2813 -0.4617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2813 -0.4617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5669 -0.8741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5669 -0.8741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1476 -0.4617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1476 -0.4617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1476 0.3633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1476 0.3633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5669 0.7758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5669 0.7758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8620 -0.8741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8620 -0.8741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5764 -0.4617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5764 -0.4617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5764 0.3633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5764 0.3633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8620 0.7758 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8620 0.7758 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8620 -1.5174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8620 -1.5174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2906 0.7757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2906 0.7757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0187 0.3552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0187 0.3552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7468 0.7757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7468 0.7757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7468 1.6163 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7468 1.6163 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0187 2.0368 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0187 2.0368 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2906 1.6163 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2906 1.6163 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9955 0.7757 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9955 0.7757 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3728 -0.9007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3728 -0.9007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5669 -1.6988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5669 -1.6988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3170 -2.4122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3170 -2.4122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8858 -3.1588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8858 -3.1588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7149 -2.9219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7149 -2.9219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5491 -3.1588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5491 -3.1588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9803 -2.4122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9803 -2.4122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1511 -2.6490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1511 -2.6490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5396 -2.5424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5396 -2.5424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5322 -0.4443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5322 -0.4443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1011 -1.1909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1011 -1.1909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9302 -0.9540 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9302 -0.9540 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7643 -1.1909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7643 -1.1909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1956 -0.4443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1956 -0.4443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3664 -0.6811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3664 -0.6811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7535 -0.4951 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7535 -0.4951 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0025 -0.1248 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0025 -0.1248 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8691 -0.5638 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8691 -0.5638 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3912 -1.0892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3912 -1.0892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8047 -1.6287 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8047 -1.6287 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2457 -3.0050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2457 -3.0050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2376 -3.5476 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2376 -3.5476 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5491 -3.8864 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5491 -3.8864 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4478 2.0714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4478 2.0714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8590 0.8700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8590 0.8700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3382 0.1827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3382 0.1827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5884 0.4742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5884 0.4742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8070 0.4656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8070 0.4656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3907 1.0080 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3907 1.0080 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1566 0.7329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1566 0.7329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4652 0.5974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4652 0.5974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1583 0.1117 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1583 0.1117 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8275 -0.0214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8275 -0.0214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4343 0.4237 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4343 0.4237 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7185 0.4237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7185 0.4237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0128 2.7340 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0128 2.7340 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5797 3.8864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5797 3.8864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7273 1.3702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7273 1.3702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.9803 1.6514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.9803 1.6514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 8 19 1 0 0 0 0 | + | 8 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 1 0 0 0 | + | 29 30 1 1 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 32 31 1 1 0 0 0 | + | 32 31 1 1 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 28 1 0 0 0 0 | + | 33 28 1 0 0 0 0 |
| − | 28 34 1 0 0 0 0 | + | 28 34 1 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 32 36 1 0 0 0 0 | + | 32 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 25 38 1 0 0 0 0 | + | 25 38 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 22 39 1 0 0 0 0 | + | 22 39 1 0 0 0 0 |
| − | 23 40 1 0 0 0 0 | + | 23 40 1 0 0 0 0 |
| − | 24 41 1 0 0 0 0 | + | 24 41 1 0 0 0 0 |
| − | 29 19 1 0 0 0 0 | + | 29 19 1 0 0 0 0 |
| − | 42 15 1 0 0 0 0 | + | 42 15 1 0 0 0 0 |
| − | 43 44 1 1 0 0 0 | + | 43 44 1 1 0 0 0 |
| − | 44 45 1 1 0 0 0 | + | 44 45 1 1 0 0 0 |
| − | 46 45 1 1 0 0 0 | + | 46 45 1 1 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 43 1 0 0 0 0 | + | 48 43 1 0 0 0 0 |
| − | 43 49 1 0 0 0 0 | + | 43 49 1 0 0 0 0 |
| − | 44 50 1 0 0 0 0 | + | 44 50 1 0 0 0 0 |
| − | 45 51 1 0 0 0 0 | + | 45 51 1 0 0 0 0 |
| − | 46 18 1 0 0 0 0 | + | 46 18 1 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 14 52 1 0 0 0 0 | + | 14 52 1 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 16 54 1 0 0 0 0 | + | 16 54 1 0 0 0 0 |
| − | 56 57 1 0 0 0 0 | + | 56 57 1 0 0 0 0 |
| − | 48 56 1 0 0 0 0 | + | 48 56 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 52 53 | + | M SAL 1 2 52 53 |
| − | M SBL 1 1 58 | + | M SBL 1 1 58 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 58 -0.6875 0.3520 | + | M SBV 1 58 -0.6875 0.3520 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 54 55 | + | M SAL 2 2 54 55 |
| − | M SBL 2 1 60 | + | M SBL 2 1 60 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SBV 2 60 0.0059 -0.6972 | + | M SBV 2 60 0.0059 -0.6972 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 56 57 | + | M SAL 3 2 56 57 |
| − | M SBL 3 1 62 | + | M SBL 3 1 62 |
| − | M SMT 3 ^ CH2OH | + | M SMT 3 ^ CH2OH |
| − | M SBV 3 62 0.5707 -0.6373 | + | M SBV 3 62 0.5707 -0.6373 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAIGL0003 | + | ID FL5FAIGL0003 |
| − | FORMULA C35H44O22 | + | FORMULA C35H44O22 |
| − | EXACTMASS 816.232423092 | + | EXACTMASS 816.232423092 |
| − | AVERAGEMASS 816.71066 | + | AVERAGEMASS 816.71066 |
| − | SMILES c(OC)(c1)c(c(OC)cc(C(=C4OC(C(O)6)OC(C(O)C(O)6)COC(C5O)OC(C)C(C5O)O)Oc(c(C(=O)4)3)cc(cc(O)3)OC(C(O)2)OC(C(O)C(O)2)CO)1)O | + | SMILES c(OC)(c1)c(c(OC)cc(C(=C4OC(C(O)6)OC(C(O)C(O)6)COC(C5O)OC(C)C(C5O)O)Oc(c(C(=O)4)3)cc(cc(O)3)OC(C(O)2)OC(C(O)C(O)2)CO)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
57 62 0 0 0 0 0 0 0 0999 V2000
-1.2813 0.3633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2813 -0.4617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5669 -0.8741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1476 -0.4617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1476 0.3633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5669 0.7758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8620 -0.8741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5764 -0.4617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5764 0.3633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8620 0.7758 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8620 -1.5174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2906 0.7757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0187 0.3552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7468 0.7757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7468 1.6163 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0187 2.0368 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2906 1.6163 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9955 0.7757 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3728 -0.9007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5669 -1.6988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3170 -2.4122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8858 -3.1588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7149 -2.9219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5491 -3.1588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9803 -2.4122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1511 -2.6490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5396 -2.5424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5322 -0.4443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1011 -1.1909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9302 -0.9540 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7643 -1.1909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1956 -0.4443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3664 -0.6811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7535 -0.4951 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0025 -0.1248 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.8691 -0.5638 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.3912 -1.0892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.8047 -1.6287 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2457 -3.0050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2376 -3.5476 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.5491 -3.8864 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4478 2.0714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8590 0.8700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3382 0.1827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5884 0.4742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8070 0.4656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3907 1.0080 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1566 0.7329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.4652 0.5974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.1583 0.1117 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8275 -0.0214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4343 0.4237 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.7185 0.4237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0128 2.7340 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5797 3.8864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7273 1.3702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.9803 1.6514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
8 19 1 0 0 0 0
3 20 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
28 29 1 0 0 0 0
29 30 1 1 0 0 0
30 31 1 1 0 0 0
32 31 1 1 0 0 0
32 33 1 0 0 0 0
33 28 1 0 0 0 0
28 34 1 0 0 0 0
33 35 1 0 0 0 0
32 36 1 0 0 0 0
31 37 1 0 0 0 0
25 38 1 0 0 0 0
37 38 1 0 0 0 0
22 39 1 0 0 0 0
23 40 1 0 0 0 0
24 41 1 0 0 0 0
29 19 1 0 0 0 0
42 15 1 0 0 0 0
43 44 1 1 0 0 0
44 45 1 1 0 0 0
46 45 1 1 0 0 0
46 47 1 0 0 0 0
47 48 1 0 0 0 0
48 43 1 0 0 0 0
43 49 1 0 0 0 0
44 50 1 0 0 0 0
45 51 1 0 0 0 0
46 18 1 0 0 0 0
52 53 1 0 0 0 0
14 52 1 0 0 0 0
54 55 1 0 0 0 0
16 54 1 0 0 0 0
56 57 1 0 0 0 0
48 56 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 52 53
M SBL 1 1 58
M SMT 1 OCH3
M SBV 1 58 -0.6875 0.3520
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 54 55
M SBL 2 1 60
M SMT 2 OCH3
M SBV 2 60 0.0059 -0.6972
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 56 57
M SBL 3 1 62
M SMT 3 ^ CH2OH
M SBV 3 62 0.5707 -0.6373
S SKP 5
ID FL5FAIGL0003
FORMULA C35H44O22
EXACTMASS 816.232423092
AVERAGEMASS 816.71066
SMILES c(OC)(c1)c(c(OC)cc(C(=C4OC(C(O)6)OC(C(O)C(O)6)COC(C5O)OC(C)C(C5O)O)Oc(c(C(=O)4)3)cc(cc(O)3)OC(C(O)2)OC(C(O)C(O)2)CO)1)O
M END