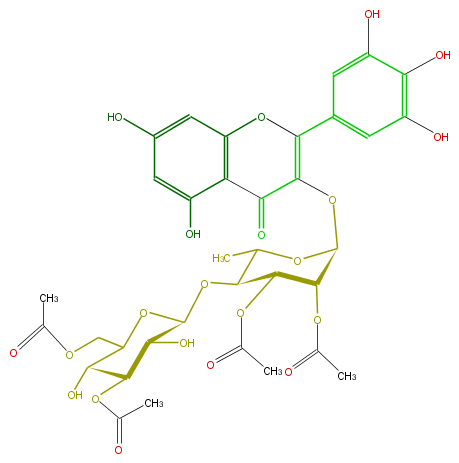
Mol:FL5FAGGS0036
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 56 60 0 0 0 0 0 0 0 0999 V2000 | + | 56 60 0 0 0 0 0 0 0 0999 V2000 |
| − | 2.8334 1.8887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8334 1.8887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1189 2.3012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1189 2.3012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1189 3.1262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1189 3.1262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8334 3.5387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8334 3.5387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5479 3.1262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5479 3.1262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5479 2.3012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5479 2.3012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4044 1.8887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4044 1.8887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6900 2.3012 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6900 2.3012 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0245 1.8887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0245 1.8887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0245 1.0637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0245 1.0637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6900 0.6512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6900 0.6512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4044 1.0637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4044 1.0637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7390 2.3012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7390 2.3012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4534 1.8887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4534 1.8887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4534 1.0637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4534 1.0637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7390 0.6512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7390 0.6512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6900 -0.0557 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6900 -0.0557 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1679 2.3012 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1679 2.3012 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1189 0.6512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1189 0.6512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7390 -0.0398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7390 -0.0398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2623 3.5387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2623 3.5387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8334 4.3637 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8334 4.3637 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2258 1.9098 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2258 1.9098 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6159 -0.3593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6159 -0.3593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2032 -1.0740 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2032 -1.0740 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9967 -0.8472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9967 -0.8472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7950 -1.0564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7950 -1.0564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2078 -0.3417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2078 -0.3417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4142 -0.5684 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4142 -0.5684 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8096 -1.7650 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8096 -1.7650 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2833 -1.6206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2833 -1.6206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4374 -1.0269 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4374 -1.0269 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0037 -0.5233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0037 -0.5233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8096 -2.3837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8096 -2.3837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2447 -2.7924 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2447 -2.7924 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2970 -2.8712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2970 -2.8712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2833 -2.3219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2833 -2.3219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3163 -2.6570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3163 -2.6570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8026 -2.7793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8026 -2.7793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7909 -2.7201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7909 -2.7201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9938 -2.9337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9938 -2.9337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5771 -2.2212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5771 -2.2212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8556 -1.8206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8556 -1.8206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6528 -1.6069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6528 -1.6069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0695 -2.3193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0695 -2.3193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5629 -2.1362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5629 -2.1362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1404 -2.4662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1404 -2.4662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1027 -3.2602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1027 -3.2602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6248 -3.3325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6248 -3.3325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8714 -2.2212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8714 -2.2212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1524 -3.7535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1524 -3.7535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1524 -4.3637 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1524 -4.3637 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5771 -3.4213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5771 -3.4213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6767 -1.8887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6767 -1.8887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2623 -2.4068 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2623 -2.4068 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6767 -1.3112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6767 -1.3112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 5 21 1 0 0 0 0 | + | 5 21 1 0 0 0 0 |
| − | 4 22 1 0 0 0 0 | + | 4 22 1 0 0 0 0 |
| − | 6 23 1 0 0 0 0 | + | 6 23 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 28 27 1 1 0 0 0 | + | 28 27 1 1 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 24 1 0 0 0 0 | + | 29 24 1 0 0 0 0 |
| − | 27 30 1 0 0 0 0 | + | 27 30 1 0 0 0 0 |
| − | 26 31 1 0 0 0 0 | + | 26 31 1 0 0 0 0 |
| − | 25 32 1 0 0 0 0 | + | 25 32 1 0 0 0 0 |
| − | 24 33 1 0 0 0 0 | + | 24 33 1 0 0 0 0 |
| − | 30 34 1 0 0 0 0 | + | 30 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 34 36 1 0 0 0 0 | + | 34 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 37 38 2 0 0 0 0 | + | 37 38 2 0 0 0 0 |
| − | 37 39 1 0 0 0 0 | + | 37 39 1 0 0 0 0 |
| − | 28 19 1 0 0 0 0 | + | 28 19 1 0 0 0 0 |
| − | 40 41 1 1 0 0 0 | + | 40 41 1 1 0 0 0 |
| − | 41 42 1 1 0 0 0 | + | 41 42 1 1 0 0 0 |
| − | 43 42 1 1 0 0 0 | + | 43 42 1 1 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 40 1 0 0 0 0 | + | 45 40 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 40 48 1 0 0 0 0 | + | 40 48 1 0 0 0 0 |
| − | 41 49 1 0 0 0 0 | + | 41 49 1 0 0 0 0 |
| − | 42 50 1 0 0 0 0 | + | 42 50 1 0 0 0 0 |
| − | 49 51 1 0 0 0 0 | + | 49 51 1 0 0 0 0 |
| − | 51 52 2 0 0 0 0 | + | 51 52 2 0 0 0 0 |
| − | 51 53 1 0 0 0 0 | + | 51 53 1 0 0 0 0 |
| − | 47 54 1 0 0 0 0 | + | 47 54 1 0 0 0 0 |
| − | 54 55 2 0 0 0 0 | + | 54 55 2 0 0 0 0 |
| − | 54 56 1 0 0 0 0 | + | 54 56 1 0 0 0 0 |
| − | 43 32 1 0 0 0 0 | + | 43 32 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAGGS0036 | + | ID FL5FAGGS0036 |
| − | FORMULA C35H38O21 | + | FORMULA C35H38O21 |
| − | EXACTMASS 794.190558278 | + | EXACTMASS 794.190558278 |
| − | AVERAGEMASS 794.66362 | + | AVERAGEMASS 794.66362 |
| − | SMILES C(OC(C)=O)(C(O)5)C(O)C(OC5COC(C)=O)OC(C(C)1)C(OC(C)=O)C(OC(C)=O)C(OC(C(=O)2)=C(c(c4)cc(c(c(O)4)O)O)Oc(c3)c2c(O)cc3O)O1 | + | SMILES C(OC(C)=O)(C(O)5)C(O)C(OC5COC(C)=O)OC(C(C)1)C(OC(C)=O)C(OC(C)=O)C(OC(C(=O)2)=C(c(c4)cc(c(c(O)4)O)O)Oc(c3)c2c(O)cc3O)O1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
56 60 0 0 0 0 0 0 0 0999 V2000
2.8334 1.8887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1189 2.3012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1189 3.1262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8334 3.5387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5479 3.1262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5479 2.3012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4044 1.8887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6900 2.3012 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0245 1.8887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0245 1.0637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6900 0.6512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4044 1.0637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7390 2.3012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4534 1.8887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4534 1.0637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7390 0.6512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6900 -0.0557 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1679 2.3012 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1189 0.6512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7390 -0.0398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2623 3.5387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8334 4.3637 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2258 1.9098 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6159 -0.3593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2032 -1.0740 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9967 -0.8472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7950 -1.0564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2078 -0.3417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4142 -0.5684 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8096 -1.7650 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2833 -1.6206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4374 -1.0269 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0037 -0.5233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8096 -2.3837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2447 -2.7924 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2970 -2.8712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2833 -2.3219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3163 -2.6570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8026 -2.7793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7909 -2.7201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9938 -2.9337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5771 -2.2212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8556 -1.8206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6528 -1.6069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0695 -2.3193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5629 -2.1362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1404 -2.4662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1027 -3.2602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6248 -3.3325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8714 -2.2212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1524 -3.7535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1524 -4.3637 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5771 -3.4213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6767 -1.8887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2623 -2.4068 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6767 -1.3112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
16 20 1 0 0 0 0
5 21 1 0 0 0 0
4 22 1 0 0 0 0
6 23 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 1 0 0 0
27 26 1 1 0 0 0
28 27 1 1 0 0 0
28 29 1 0 0 0 0
29 24 1 0 0 0 0
27 30 1 0 0 0 0
26 31 1 0 0 0 0
25 32 1 0 0 0 0
24 33 1 0 0 0 0
30 34 1 0 0 0 0
34 35 2 0 0 0 0
34 36 1 0 0 0 0
31 37 1 0 0 0 0
37 38 2 0 0 0 0
37 39 1 0 0 0 0
28 19 1 0 0 0 0
40 41 1 1 0 0 0
41 42 1 1 0 0 0
43 42 1 1 0 0 0
43 44 1 0 0 0 0
44 45 1 0 0 0 0
45 40 1 0 0 0 0
45 46 1 0 0 0 0
46 47 1 0 0 0 0
40 48 1 0 0 0 0
41 49 1 0 0 0 0
42 50 1 0 0 0 0
49 51 1 0 0 0 0
51 52 2 0 0 0 0
51 53 1 0 0 0 0
47 54 1 0 0 0 0
54 55 2 0 0 0 0
54 56 1 0 0 0 0
43 32 1 0 0 0 0
S SKP 5
ID FL5FAGGS0036
FORMULA C35H38O21
EXACTMASS 794.190558278
AVERAGEMASS 794.66362
SMILES C(OC(C)=O)(C(O)5)C(O)C(OC5COC(C)=O)OC(C(C)1)C(OC(C)=O)C(OC(C)=O)C(OC(C(=O)2)=C(c(c4)cc(c(c(O)4)O)O)Oc(c3)c2c(O)cc3O)O1
M END