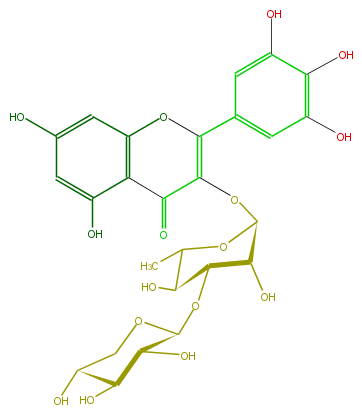
Mol:FL5FAGGS0021
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.7861 1.3917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7861 1.3917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0717 1.8042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0717 1.8042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0717 2.6291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0717 2.6291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7861 3.0416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7861 3.0416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5006 2.6291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5006 2.6291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5006 1.8042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5006 1.8042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3572 1.3917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3572 1.3917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3572 1.8042 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3572 1.8042 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0717 1.3917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0717 1.3917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0717 0.5667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0717 0.5667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3572 0.1542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3572 0.1542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3572 0.5667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3572 0.5667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7861 1.8042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7861 1.8042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5005 1.3917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5005 1.3917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5005 0.5667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5005 0.5667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7861 0.1542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7861 0.1542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3572 -0.5528 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3572 -0.5528 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2150 1.8042 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2150 1.8042 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0717 0.1542 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0717 0.1542 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7861 -0.5368 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7861 -0.5368 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2150 3.0416 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2150 3.0416 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7861 3.8666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7861 3.8666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1785 1.4128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1785 1.4128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0213 -0.8634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0213 -0.8634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1221 -1.6762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1221 -1.6762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5460 -1.1918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5460 -1.1918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3677 -1.1153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3677 -1.1153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5111 -0.3025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5111 -0.3025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8429 -0.7870 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8429 -0.7870 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6595 -1.7849 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6595 -1.7849 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2942 -1.9795 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2942 -1.9795 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5806 -1.6025 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5806 -1.6025 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5254 -1.1635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5254 -1.1635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0773 -3.2862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0773 -3.2862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3019 -3.5685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3019 -3.5685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8247 -2.8952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8247 -2.8952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0711 -2.5589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0711 -2.5589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8465 -2.2766 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8465 -2.2766 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3237 -2.9499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3237 -2.9499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0721 -2.9768 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0721 -2.9768 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8126 -3.8596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8126 -3.8596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4705 -3.8666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4705 -3.8666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 5 21 1 0 0 0 0 | + | 5 21 1 0 0 0 0 |
| − | 4 22 1 0 0 0 0 | + | 4 22 1 0 0 0 0 |
| − | 6 23 1 0 0 0 0 | + | 6 23 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 28 27 1 1 0 0 0 | + | 28 27 1 1 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 24 1 0 0 0 0 | + | 29 24 1 0 0 0 0 |
| − | 27 30 1 0 0 0 0 | + | 27 30 1 0 0 0 0 |
| − | 26 31 1 0 0 0 0 | + | 26 31 1 0 0 0 0 |
| − | 25 32 1 0 0 0 0 | + | 25 32 1 0 0 0 0 |
| − | 24 33 1 0 0 0 0 | + | 24 33 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 37 36 1 1 0 0 0 | + | 37 36 1 1 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 36 40 1 0 0 0 0 | + | 36 40 1 0 0 0 0 |
| − | 35 41 1 0 0 0 0 | + | 35 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 37 31 1 0 0 0 0 | + | 37 31 1 0 0 0 0 |
| − | 28 19 1 0 0 0 0 | + | 28 19 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAGGS0021 | + | ID FL5FAGGS0021 |
| − | FORMULA C26H28O16 | + | FORMULA C26H28O16 |
| − | EXACTMASS 596.137734848 | + | EXACTMASS 596.137734848 |
| − | AVERAGEMASS 596.49092 | + | AVERAGEMASS 596.49092 |
| − | SMILES c(c1O)c(c(C3=O)c(OC(=C3OC(O5)C(C(C(O)C5C)OC(C(O)4)OCC(O)C4O)O)c(c2)cc(c(c(O)2)O)O)c1)O | + | SMILES c(c1O)c(c(C3=O)c(OC(=C3OC(O5)C(C(C(O)C5C)OC(C(O)4)OCC(O)C4O)O)c(c2)cc(c(c(O)2)O)O)c1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
1.7861 1.3917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0717 1.8042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0717 2.6291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7861 3.0416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5006 2.6291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5006 1.8042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3572 1.3917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3572 1.8042 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0717 1.3917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0717 0.5667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3572 0.1542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3572 0.5667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7861 1.8042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5005 1.3917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5005 0.5667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7861 0.1542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3572 -0.5528 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2150 1.8042 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0717 0.1542 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7861 -0.5368 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2150 3.0416 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7861 3.8666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1785 1.4128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0213 -0.8634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1221 -1.6762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5460 -1.1918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3677 -1.1153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5111 -0.3025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8429 -0.7870 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6595 -1.7849 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2942 -1.9795 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5806 -1.6025 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5254 -1.1635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0773 -3.2862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3019 -3.5685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8247 -2.8952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0711 -2.5589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8465 -2.2766 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3237 -2.9499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0721 -2.9768 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8126 -3.8596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4705 -3.8666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
16 20 1 0 0 0 0
5 21 1 0 0 0 0
4 22 1 0 0 0 0
6 23 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 1 0 0 0
27 26 1 1 0 0 0
28 27 1 1 0 0 0
28 29 1 0 0 0 0
29 24 1 0 0 0 0
27 30 1 0 0 0 0
26 31 1 0 0 0 0
25 32 1 0 0 0 0
24 33 1 0 0 0 0
34 35 1 1 0 0 0
35 36 1 1 0 0 0
37 36 1 1 0 0 0
37 38 1 0 0 0 0
38 39 1 0 0 0 0
39 34 1 0 0 0 0
36 40 1 0 0 0 0
35 41 1 0 0 0 0
34 42 1 0 0 0 0
37 31 1 0 0 0 0
28 19 1 0 0 0 0
S SKP 5
ID FL5FAGGS0021
FORMULA C26H28O16
EXACTMASS 596.137734848
AVERAGEMASS 596.49092
SMILES c(c1O)c(c(C3=O)c(OC(=C3OC(O5)C(C(C(O)C5C)OC(C(O)4)OCC(O)C4O)O)c(c2)cc(c(c(O)2)O)O)c1)O
M END