Mol:FL5FADGSS001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 39 42 0 0 0 0 0 0 0 0999 V2000 | + | 39 42 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.8098 0.2566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8098 0.2566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8099 -0.5530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8099 -0.5530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1087 -0.9578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1087 -0.9578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4078 -0.5530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4078 -0.5530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4077 0.2566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4077 0.2566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1087 0.6613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1087 0.6613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7066 -0.9578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7066 -0.9578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0056 -0.5530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0056 -0.5530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0056 0.2566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0056 0.2566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7066 0.6613 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7066 0.6613 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7066 -1.6784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7066 -1.6784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6952 0.6612 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6952 0.6612 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4097 0.2487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4097 0.2487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1243 0.6611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1243 0.6611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1243 1.4861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1243 1.4861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4097 1.8988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4097 1.8988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6952 1.4862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6952 1.4862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7728 -1.0152 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7728 -1.0152 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0693 -1.1398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0693 -1.1398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2689 -1.7002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2689 -1.7002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2338 -1.8539 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2338 -1.8539 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9769 -2.4967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9769 -2.4967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7773 -1.9363 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7773 -1.9363 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8124 -1.7825 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8124 -1.7825 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4063 -0.7571 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4063 -0.7571 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3674 -1.3939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3674 -1.3939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1865 -2.3455 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1865 -2.3455 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8618 1.9119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8618 1.9119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1087 -1.6974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1087 -1.6974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9743 0.7100 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9743 0.7100 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2757 0.7099 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2757 0.7099 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4534 0.7099 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4534 0.7099 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2757 1.4422 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2757 1.4422 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2757 0.0160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2757 0.0160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9420 -2.7552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9420 -2.7552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9743 -2.0324 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9743 -2.0324 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0326 -3.7910 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0326 -3.7910 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4097 2.6602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4097 2.6602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9660 3.7910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9660 3.7910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 24 26 1 0 0 0 0 | + | 24 26 1 0 0 0 0 |
| − | 23 27 1 0 0 0 0 | + | 23 27 1 0 0 0 0 |
| − | 20 18 1 0 0 0 0 | + | 20 18 1 0 0 0 0 |
| − | 18 8 1 0 0 0 0 | + | 18 8 1 0 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 15 28 1 0 0 0 0 | + | 15 28 1 0 0 0 0 |
| − | 3 29 1 0 0 0 0 | + | 3 29 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 31 33 2 0 0 0 0 | + | 31 33 2 0 0 0 0 |
| − | 31 34 2 0 0 0 0 | + | 31 34 2 0 0 0 0 |
| − | 1 32 1 0 0 0 0 | + | 1 32 1 0 0 0 0 |
| − | 35 36 2 0 0 0 0 | + | 35 36 2 0 0 0 0 |
| − | 35 37 1 0 0 0 0 | + | 35 37 1 0 0 0 0 |
| − | 22 35 1 0 0 0 0 | + | 22 35 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 16 38 1 0 0 0 0 | + | 16 38 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 3 35 36 37 | + | M SAL 1 3 35 36 37 |
| − | M SBL 1 1 40 | + | M SBL 1 1 40 |
| − | M SMT 1 COOH | + | M SMT 1 COOH |
| − | M SBV 1 40 -0.9650 0.2586 | + | M SBV 1 40 -0.9650 0.2586 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 38 39 | + | M SAL 2 2 38 39 |
| − | M SBL 2 1 42 | + | M SBL 2 1 42 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SBV 2 42 0.0000 -0.7614 | + | M SBV 2 42 0.0000 -0.7614 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FADGSS001 | + | ID FL5FADGSS001 |
| − | FORMULA C22H20O16S | + | FORMULA C22H20O16S |
| − | EXACTMASS 572.047205282 | + | EXACTMASS 572.047205282 |
| − | AVERAGEMASS 572.4506 | + | AVERAGEMASS 572.4506 |
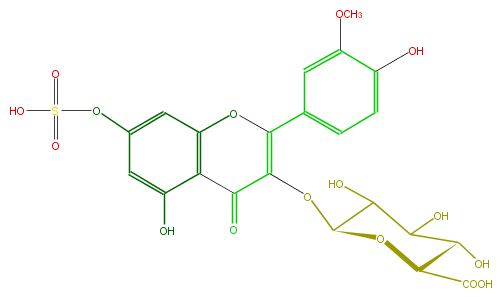
| − | SMILES O(c(c(O)4)cc(cc4)C(O1)=C(OC(C(O)3)OC(C(O)C3O)C(O)=O)C(=O)c(c2O)c(cc(c2)OS(O)(=O)=O)1)C | + | SMILES O(c(c(O)4)cc(cc4)C(O1)=C(OC(C(O)3)OC(C(O)C3O)C(O)=O)C(=O)c(c2O)c(cc(c2)OS(O)(=O)=O)1)C |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
39 42 0 0 0 0 0 0 0 0999 V2000
-2.8098 0.2566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8099 -0.5530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1087 -0.9578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4078 -0.5530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4077 0.2566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1087 0.6613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7066 -0.9578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0056 -0.5530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0056 0.2566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7066 0.6613 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7066 -1.6784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6952 0.6612 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4097 0.2487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1243 0.6611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1243 1.4861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4097 1.8988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6952 1.4862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7728 -1.0152 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0693 -1.1398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2689 -1.7002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2338 -1.8539 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9769 -2.4967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7773 -1.9363 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8124 -1.7825 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4063 -0.7571 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3674 -1.3939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1865 -2.3455 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8618 1.9119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1087 -1.6974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9743 0.7100 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2757 0.7099 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0
-3.4534 0.7099 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2757 1.4422 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2757 0.0160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9420 -2.7552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9743 -2.0324 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0326 -3.7910 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4097 2.6602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9660 3.7910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
19 20 1 0 0 0 0
20 21 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
19 25 1 0 0 0 0
24 26 1 0 0 0 0
23 27 1 0 0 0 0
20 18 1 0 0 0 0
18 8 1 0 0 0 0
22 21 1 1 0 0 0
15 28 1 0 0 0 0
3 29 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
31 33 2 0 0 0 0
31 34 2 0 0 0 0
1 32 1 0 0 0 0
35 36 2 0 0 0 0
35 37 1 0 0 0 0
22 35 1 0 0 0 0
38 39 1 0 0 0 0
16 38 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 3 35 36 37
M SBL 1 1 40
M SMT 1 COOH
M SBV 1 40 -0.9650 0.2586
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 38 39
M SBL 2 1 42
M SMT 2 OCH3
M SBV 2 42 0.0000 -0.7614
S SKP 5
ID FL5FADGSS001
FORMULA C22H20O16S
EXACTMASS 572.047205282
AVERAGEMASS 572.4506
SMILES O(c(c(O)4)cc(cc4)C(O1)=C(OC(C(O)3)OC(C(O)C3O)C(O)=O)C(=O)c(c2O)c(cc(c2)OS(O)(=O)=O)1)C
M END