Mol:FL5FADGL0018
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 56 61 0 0 0 0 0 0 0 0999 V2000 | + | 56 61 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.8270 1.2407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8270 1.2407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8270 0.5983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8270 0.5983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2707 0.2771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2707 0.2771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7144 0.5983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7144 0.5983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7144 1.2407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7144 1.2407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2707 1.5619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2707 1.5619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1581 0.2771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1581 0.2771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6018 0.5983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6018 0.5983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6018 1.2407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6018 1.2407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1581 1.5619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1581 1.5619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1581 -0.2237 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1581 -0.2237 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0987 1.6855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0987 1.6855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6657 1.3581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6657 1.3581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2326 1.6855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2326 1.6855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2326 2.3402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2326 2.3402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6657 2.6675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6657 2.6675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0987 2.3402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0987 2.3402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2707 -0.3650 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2707 -0.3650 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0986 2.8403 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0986 2.8403 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4915 1.6243 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4915 1.6243 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1578 0.1543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1578 0.1543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0749 0.5696 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.0749 0.5696 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.7740 0.0485 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.7740 0.0485 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.3525 0.2139 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3525 0.2139 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9345 0.0485 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.9345 0.0485 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.2354 0.5696 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.2354 0.5696 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.6569 0.4043 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.6569 0.4043 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.5162 0.4199 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5162 0.4199 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6984 0.8788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6984 0.8788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9592 0.3756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9592 0.3756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4861 0.0485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4861 0.0485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2789 -2.3943 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 2.2789 -2.3943 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 2.9279 -2.2268 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.9279 -2.2268 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.8969 -1.5639 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.8969 -1.5639 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.2210 -0.9917 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.2210 -0.9917 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.5092 -1.2429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5092 -1.2429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5720 -1.9338 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.5720 -1.9338 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.2789 -2.9214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2789 -2.9214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6554 -3.2436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6554 -3.2436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4915 -1.5557 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4915 -1.5557 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9002 -2.1006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9002 -2.1006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4282 -1.8767 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -0.4282 -1.8767 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -0.0570 -2.3666 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.0570 -2.3666 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.4775 -2.1588 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.4775 -2.1588 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.9932 -2.1532 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.9932 -2.1532 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.6184 -1.7784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6184 -1.7784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1508 -2.0252 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.1508 -2.0252 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -1.0191 -1.8249 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0191 -1.8249 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4074 -2.8279 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4074 -2.8279 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7837 -2.6729 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7837 -2.6729 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0209 -0.4863 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0209 -0.4863 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4052 -1.9768 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4052 -1.9768 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5417 -1.4828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5417 -1.4828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3170 -1.2007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3170 -1.2007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9491 3.2436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9491 3.2436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3905 4.1409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3905 4.1409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 25 31 1 0 0 0 0 | + | 25 31 1 0 0 0 0 |
| − | 23 21 1 0 0 0 0 | + | 23 21 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 37 41 1 0 0 0 0 | + | 37 41 1 0 0 0 0 |
| − | 42 43 1 1 0 0 0 | + | 42 43 1 1 0 0 0 |
| − | 43 44 1 1 0 0 0 | + | 43 44 1 1 0 0 0 |
| − | 45 44 1 1 0 0 0 | + | 45 44 1 1 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 42 1 0 0 0 0 | + | 47 42 1 0 0 0 0 |
| − | 42 48 1 0 0 0 0 | + | 42 48 1 0 0 0 0 |
| − | 43 49 1 0 0 0 0 | + | 43 49 1 0 0 0 0 |
| − | 44 50 1 0 0 0 0 | + | 44 50 1 0 0 0 0 |
| − | 31 51 1 0 0 0 0 | + | 31 51 1 0 0 0 0 |
| − | 51 35 1 0 0 0 0 | + | 51 35 1 0 0 0 0 |
| − | 41 52 1 0 0 0 0 | + | 41 52 1 0 0 0 0 |
| − | 52 45 1 0 0 0 0 | + | 52 45 1 0 0 0 0 |
| − | 47 53 1 0 0 0 0 | + | 47 53 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 16 55 1 0 0 0 0 | + | 16 55 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 53 54 | + | M SAL 2 2 53 54 |
| − | M SBL 2 1 58 | + | M SBL 2 1 58 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 58 -0.2476 -1.241 | + | M SVB 2 58 -0.2476 -1.241 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 55 56 | + | M SAL 1 2 55 56 |
| − | M SBL 1 1 60 | + | M SBL 1 1 60 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 60 0.9491 3.2436 | + | M SVB 1 60 0.9491 3.2436 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FADGL0018 | + | ID FL5FADGL0018 |
| − | KNApSAcK_ID C00005569 | + | KNApSAcK_ID C00005569 |
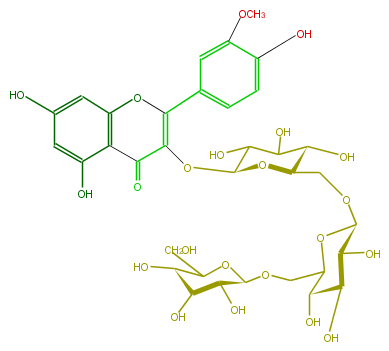
| − | NAME Isorhamnetin 3-gentiotrioside | + | NAME Isorhamnetin 3-gentiotrioside |
| − | CAS_RN 84534-25-8 | + | CAS_RN 84534-25-8 |
| − | FORMULA C34H42O22 | + | FORMULA C34H42O22 |
| − | EXACTMASS 802.216773028 | + | EXACTMASS 802.216773028 |
| − | AVERAGEMASS 802.68408 | + | AVERAGEMASS 802.68408 |
| − | SMILES c(c(C(O5)=C(C(=O)c(c6O)c5cc(c6)O)O[C@@H](C2O)O[C@H](CO[C@H](O3)[C@@H](O)[C@H]([C@@H](O)C(CO[C@H](O4)[C@H]([C@@H](O)[C@H](C4CO)O)O)3)O)[C@H](O)C(O)2)1)cc(c(OC)c1)O | + | SMILES c(c(C(O5)=C(C(=O)c(c6O)c5cc(c6)O)O[C@@H](C2O)O[C@H](CO[C@H](O3)[C@@H](O)[C@H]([C@@H](O)C(CO[C@H](O4)[C@H]([C@@H](O)[C@H](C4CO)O)O)3)O)[C@H](O)C(O)2)1)cc(c(OC)c1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
56 61 0 0 0 0 0 0 0 0999 V2000
-2.8270 1.2407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8270 0.5983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2707 0.2771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7144 0.5983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7144 1.2407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2707 1.5619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1581 0.2771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6018 0.5983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6018 1.2407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1581 1.5619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1581 -0.2237 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0987 1.6855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6657 1.3581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2326 1.6855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2326 2.3402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6657 2.6675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0987 2.3402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2707 -0.3650 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0986 2.8403 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4915 1.6243 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1578 0.1543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0749 0.5696 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.7740 0.0485 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.3525 0.2139 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9345 0.0485 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.2354 0.5696 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.6569 0.4043 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.5162 0.4199 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6984 0.8788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9592 0.3756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4861 0.0485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2789 -2.3943 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
2.9279 -2.2268 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.8969 -1.5639 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.2210 -0.9917 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.5092 -1.2429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5720 -1.9338 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.2789 -2.9214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6554 -3.2436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4915 -1.5557 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9002 -2.1006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4282 -1.8767 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-0.0570 -2.3666 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.4775 -2.1588 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.9932 -2.1532 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.6184 -1.7784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1508 -2.0252 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-1.0191 -1.8249 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4074 -2.8279 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7837 -2.6729 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0209 -0.4863 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4052 -1.9768 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5417 -1.4828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3170 -1.2007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9491 3.2436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3905 4.1409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
4 3 1 0 0 0 0
1 20 1 0 0 0 0
21 8 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
27 29 1 0 0 0 0
26 30 1 0 0 0 0
25 31 1 0 0 0 0
23 21 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 40 1 0 0 0 0
37 41 1 0 0 0 0
42 43 1 1 0 0 0
43 44 1 1 0 0 0
45 44 1 1 0 0 0
45 46 1 0 0 0 0
46 47 1 0 0 0 0
47 42 1 0 0 0 0
42 48 1 0 0 0 0
43 49 1 0 0 0 0
44 50 1 0 0 0 0
31 51 1 0 0 0 0
51 35 1 0 0 0 0
41 52 1 0 0 0 0
52 45 1 0 0 0 0
47 53 1 0 0 0 0
53 54 1 0 0 0 0
16 55 1 0 0 0 0
55 56 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 53 54
M SBL 2 1 58
M SMT 2 CH2OH
M SVB 2 58 -0.2476 -1.241
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 55 56
M SBL 1 1 60
M SMT 1 OCH3
M SVB 1 60 0.9491 3.2436
S SKP 8
ID FL5FADGL0018
KNApSAcK_ID C00005569
NAME Isorhamnetin 3-gentiotrioside
CAS_RN 84534-25-8
FORMULA C34H42O22
EXACTMASS 802.216773028
AVERAGEMASS 802.68408
SMILES c(c(C(O5)=C(C(=O)c(c6O)c5cc(c6)O)O[C@@H](C2O)O[C@H](CO[C@H](O3)[C@@H](O)[C@H]([C@@H](O)C(CO[C@H](O4)[C@H]([C@@H](O)[C@H](C4CO)O)O)3)O)[C@H](O)C(O)2)1)cc(c(OC)c1)O
M END