Mol:FL5FADGA0023
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 48 52 0 0 0 0 0 0 0 0999 V2000 | + | 48 52 0 0 0 0 0 0 0 0999 V2000 |
| − | -4.6266 0.0760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6266 0.0760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6266 -0.5663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6266 -0.5663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0703 -0.8875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0703 -0.8875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5140 -0.5663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5140 -0.5663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5140 0.0760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5140 0.0760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0703 0.3972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0703 0.3972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9577 -0.8875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9577 -0.8875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4014 -0.5663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4014 -0.5663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4014 0.0760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4014 0.0760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9577 0.3972 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9577 0.3972 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9577 -1.3883 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9577 -1.3883 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7009 0.5208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7009 0.5208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1339 0.1935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1339 0.1935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5670 0.5208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5670 0.5208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5670 1.1755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5670 1.1755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1339 1.5029 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1339 1.5029 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7009 1.1755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7009 1.1755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0703 -1.5296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0703 -1.5296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2990 1.6755 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2990 1.6755 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1422 0.5298 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1422 0.5298 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6772 -1.3471 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6772 -1.3471 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4979 -1.0282 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -0.4979 -1.0282 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.7987 -1.5492 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.7987 -1.5492 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.2202 -1.3839 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2202 -1.3839 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3618 -1.5492 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.3618 -1.5492 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.6627 -1.0282 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.6627 -1.0282 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.0841 -1.1935 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.0841 -1.1935 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -1.0566 -1.1779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0566 -1.1779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1256 -0.7189 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1256 -0.7189 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6627 -0.4146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6627 -0.4146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9133 -1.5492 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9133 -1.5492 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5376 -1.2173 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 2.5376 -1.2173 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 2.9393 -1.7476 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.9393 -1.7476 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.5177 -1.5226 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.5177 -1.5226 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 4.0759 -1.5166 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 4.0759 -1.5166 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.6703 -1.1109 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 3.6703 -1.1109 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 3.1642 -1.3780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1642 -1.3780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7878 -1.5533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7878 -1.5533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3131 -1.7780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3131 -1.7780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8492 -2.0790 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8492 -2.0790 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6703 -1.1109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6703 -1.1109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0671 -1.6487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0671 -1.6487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1597 -0.5015 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1597 -0.5015 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6287 -0.2595 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6287 -0.2595 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1422 -0.5560 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1422 -0.5560 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6287 0.2442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6287 0.2442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8505 2.0790 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8505 2.0790 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4091 2.9763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4091 2.9763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 25 31 1 0 0 0 0 | + | 25 31 1 0 0 0 0 |
| − | 21 23 1 0 0 0 0 | + | 21 23 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 36 41 1 0 0 0 0 | + | 36 41 1 0 0 0 0 |
| − | 35 42 1 0 0 0 0 | + | 35 42 1 0 0 0 0 |
| − | 38 31 1 0 0 0 0 | + | 38 31 1 0 0 0 0 |
| − | 41 43 1 0 0 0 0 | + | 41 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 2 0 0 0 0 | + | 44 45 2 0 0 0 0 |
| − | 44 46 1 0 0 0 0 | + | 44 46 1 0 0 0 0 |
| − | 16 47 1 0 0 0 0 | + | 16 47 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 47 48 | + | M SAL 1 2 47 48 |
| − | M SBL 1 1 51 | + | M SBL 1 1 51 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 51 -0.8505 2.079 | + | M SVB 1 51 -0.8505 2.079 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FADGA0023 | + | ID FL5FADGA0023 |
| − | KNApSAcK_ID C00006014 | + | KNApSAcK_ID C00006014 |
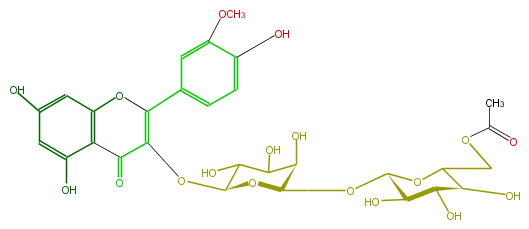
| − | NAME Isorhamnetin 3-(6'''-acetylglucosyl)(1->6)-galactoside | + | NAME Isorhamnetin 3-(6'''-acetylglucosyl)(1->6)-galactoside |
| − | CAS_RN - | + | CAS_RN - |
| − | FORMULA C30H34O18 | + | FORMULA C30H34O18 |
| − | EXACTMASS 682.174514284 | + | EXACTMASS 682.174514284 |
| − | AVERAGEMASS 682.58016 | + | AVERAGEMASS 682.58016 |
| − | SMILES [C@@H](O)([C@@H](O)1)C(O[C@H](OC[C@@H](O2)[C@H](O)C(O)C([C@@H]2OC(=C4c(c5)cc(c(c5)O)OC)C(c(c(O)3)c(O4)cc(O)c3)=O)O)[C@@H]1O)COC(C)=O | + | SMILES [C@@H](O)([C@@H](O)1)C(O[C@H](OC[C@@H](O2)[C@H](O)C(O)C([C@@H]2OC(=C4c(c5)cc(c(c5)O)OC)C(c(c(O)3)c(O4)cc(O)c3)=O)O)[C@@H]1O)COC(C)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
48 52 0 0 0 0 0 0 0 0999 V2000
-4.6266 0.0760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6266 -0.5663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0703 -0.8875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5140 -0.5663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5140 0.0760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0703 0.3972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9577 -0.8875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4014 -0.5663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4014 0.0760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9577 0.3972 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9577 -1.3883 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7009 0.5208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1339 0.1935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5670 0.5208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5670 1.1755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1339 1.5029 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7009 1.1755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0703 -1.5296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2990 1.6755 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.1422 0.5298 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6772 -1.3471 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4979 -1.0282 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.7987 -1.5492 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.2202 -1.3839 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3618 -1.5492 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.6627 -1.0282 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.0841 -1.1935 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-1.0566 -1.1779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1256 -0.7189 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6627 -0.4146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9133 -1.5492 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5376 -1.2173 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
2.9393 -1.7476 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.5177 -1.5226 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
4.0759 -1.5166 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.6703 -1.1109 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
3.1642 -1.3780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7878 -1.5533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3131 -1.7780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8492 -2.0790 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6703 -1.1109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0671 -1.6487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1597 -0.5015 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6287 -0.2595 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1422 -0.5560 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6287 0.2442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8505 2.0790 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4091 2.9763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
4 3 1 0 0 0 0
1 20 1 0 0 0 0
21 8 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
27 29 1 0 0 0 0
26 30 1 0 0 0 0
25 31 1 0 0 0 0
21 23 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 40 1 0 0 0 0
36 41 1 0 0 0 0
35 42 1 0 0 0 0
38 31 1 0 0 0 0
41 43 1 0 0 0 0
43 44 1 0 0 0 0
44 45 2 0 0 0 0
44 46 1 0 0 0 0
16 47 1 0 0 0 0
47 48 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 47 48
M SBL 1 1 51
M SMT 1 OCH3
M SVB 1 51 -0.8505 2.079
S SKP 8
ID FL5FADGA0023
KNApSAcK_ID C00006014
NAME Isorhamnetin 3-(6'''-acetylglucosyl)(1->6)-galactoside
CAS_RN -
FORMULA C30H34O18
EXACTMASS 682.174514284
AVERAGEMASS 682.58016
SMILES [C@@H](O)([C@@H](O)1)C(O[C@H](OC[C@@H](O2)[C@H](O)C(O)C([C@@H]2OC(=C4c(c5)cc(c(c5)O)OC)C(c(c(O)3)c(O4)cc(O)c3)=O)O)[C@@H]1O)COC(C)=O
M END