Mol:FL5FADGA0016
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 56 61 0 0 0 0 0 0 0 0999 V2000 | + | 56 61 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.5363 -4.5718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5363 -4.5718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1688 -4.4602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1688 -4.4602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3885 -3.8566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3885 -3.8566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9756 -3.3645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9756 -3.3645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3431 -3.4761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3431 -3.4761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1233 -4.0797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1233 -4.0797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1953 -2.7609 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1953 -2.7609 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7824 -2.2688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7824 -2.2688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1499 -2.3804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1499 -2.3804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9301 -2.9840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9301 -2.9840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6886 -2.6739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6886 -2.6739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7371 -1.8885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7371 -1.8885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9610 -1.2732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9610 -1.2732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5402 -0.7718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5402 -0.7718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1046 -0.8855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1046 -0.8855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3285 -1.5006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3285 -1.5006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0923 -2.0022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0923 -2.0022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3166 -5.1752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3166 -5.1752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0208 -3.7450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0208 -3.7450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7474 -0.1195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7474 -0.1195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7871 -0.2803 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.7871 -0.2803 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.4739 -0.8228 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.4739 -0.8228 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.0762 -0.6507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0762 -0.6507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6822 -0.8228 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.6822 -0.8228 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.9955 -0.2803 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.9955 -0.2803 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.3931 -0.4524 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 3.3931 -0.4524 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.2452 -0.2803 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2452 -0.2803 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4302 -0.0288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4302 -0.0288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8385 0.3056 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8385 0.3056 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2027 -0.5801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2027 -0.5801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.6278 -0.9928 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 6.6278 -0.9928 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 6.5399 -0.2913 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 6.5399 -0.2913 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 5.8879 -0.1910 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 5.8879 -0.1910 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 5.3712 0.1001 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 5.3712 0.1001 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 5.5286 -0.4888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5286 -0.4888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1852 -0.6075 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 6.1852 -0.6075 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 7.1789 -1.3788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.1789 -1.3788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.1541 -0.0332 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.1541 -0.0332 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8787 0.4973 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8787 0.4973 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6830 -0.2972 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6830 -0.2972 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2612 -1.5507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2612 -1.5507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1568 -7.4265 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 2.1568 -7.4265 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 2.6376 -6.9081 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.6376 -6.9081 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.2954 -6.3442 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.2954 -6.3442 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.1861 -5.7611 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.1861 -5.7611 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.8364 -6.2605 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8364 -6.2605 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1675 -6.8398 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.1675 -6.8398 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.2049 -7.9755 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2049 -7.9755 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2302 -7.2129 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2302 -7.2129 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8166 -5.8946 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8166 -5.8946 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8211 -1.3305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8211 -1.3305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7908 -2.3301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7908 -2.3301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9450 -1.3216 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9450 -1.3216 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9054 -1.0427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9054 -1.0427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4936 -7.1975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4936 -7.1975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4579 -8.1968 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4579 -8.1968 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 15 1 0 0 0 0 | + | 20 15 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 40 30 1 0 0 0 0 | + | 40 30 1 0 0 0 0 |
| − | 22 41 1 0 0 0 0 | + | 22 41 1 0 0 0 0 |
| − | 41 8 1 0 0 0 0 | + | 41 8 1 0 0 0 0 |
| − | 42 43 1 1 0 0 0 | + | 42 43 1 1 0 0 0 |
| − | 43 44 1 1 0 0 0 | + | 43 44 1 1 0 0 0 |
| − | 45 44 1 1 0 0 0 | + | 45 44 1 1 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 42 1 0 0 0 0 | + | 47 42 1 0 0 0 0 |
| − | 42 48 1 0 0 0 0 | + | 42 48 1 0 0 0 0 |
| − | 43 49 1 0 0 0 0 | + | 43 49 1 0 0 0 0 |
| − | 44 50 1 0 0 0 0 | + | 44 50 1 0 0 0 0 |
| − | 45 18 1 0 0 0 0 | + | 45 18 1 0 0 0 0 |
| − | 36 51 1 0 0 0 0 | + | 36 51 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 16 53 1 0 0 0 0 | + | 16 53 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 47 55 1 0 0 0 0 | + | 47 55 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 55 56 | + | M SAL 3 2 55 56 |
| − | M SBL 3 1 60 | + | M SBL 3 1 60 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SVB 3 60 -3.6337 1.2496 | + | M SVB 3 60 -3.6337 1.2496 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 51 52 | + | M SAL 2 2 51 52 |
| − | M SBL 2 1 56 | + | M SBL 2 1 56 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 56 2.8095 -1.0393 | + | M SVB 2 56 2.8095 -1.0393 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 53 54 | + | M SAL 1 2 53 54 |
| − | M SBL 1 1 58 | + | M SBL 1 1 58 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 58 2.5764 2.6308 | + | M SVB 1 58 2.5764 2.6308 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FADGA0016 | + | ID FL5FADGA0016 |
| − | KNApSAcK_ID C00005575 | + | KNApSAcK_ID C00005575 |
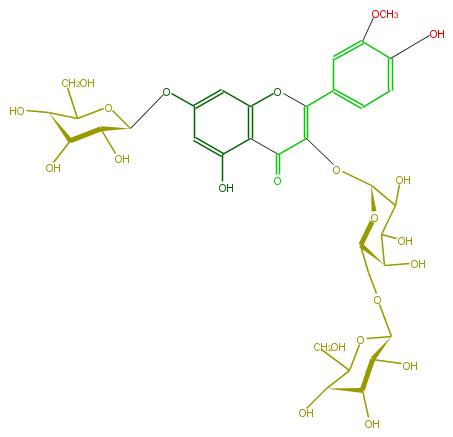
| − | NAME Isorhamnetin 3-glucosyl-(1->6)-galactoside-7-glucoside;Quercetin 3'-methyl ether 3-glucosyl-(1->6)-galactoside-7-glucoside;3-[(6-O-beta-D-Glucopyranosyl-beta-D-galactopyranosyl)oxy]-7-(beta-D-glucopyranosyloxy)-5-hydroxy-2-(4-hydroxy-3-methoxyphenyl)-4H-1-benzopyran-4-one | + | NAME Isorhamnetin 3-glucosyl-(1->6)-galactoside-7-glucoside;Quercetin 3'-methyl ether 3-glucosyl-(1->6)-galactoside-7-glucoside;3-[(6-O-beta-D-Glucopyranosyl-beta-D-galactopyranosyl)oxy]-7-(beta-D-glucopyranosyloxy)-5-hydroxy-2-(4-hydroxy-3-methoxyphenyl)-4H-1-benzopyran-4-one |
| − | CAS_RN 157201-73-5 | + | CAS_RN 157201-73-5 |
| − | FORMULA C34H42O22 | + | FORMULA C34H42O22 |
| − | EXACTMASS 802.216773028 | + | EXACTMASS 802.216773028 |
| − | AVERAGEMASS 802.68408 | + | AVERAGEMASS 802.68408 |
| − | SMILES O[C@H]([C@@H]1OC[C@@H](O2)[C@@H](C(C(O)[C@H](OC(=C(c(c6)cc(c(c6)O)OC)3)C(c(c4O)c(cc(O[C@@H]([C@H]5O)OC([C@@H]([C@H](O)5)O)CO)c4)O3)=O)2)O)O)[C@@H](O)[C@@H](O)C(O1)CO | + | SMILES O[C@H]([C@@H]1OC[C@@H](O2)[C@@H](C(C(O)[C@H](OC(=C(c(c6)cc(c(c6)O)OC)3)C(c(c4O)c(cc(O[C@@H]([C@H]5O)OC([C@@H]([C@H](O)5)O)CO)c4)O3)=O)2)O)O)[C@@H](O)[C@@H](O)C(O1)CO |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
56 61 0 0 0 0 0 0 0 0999 V2000
1.5363 -4.5718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1688 -4.4602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3885 -3.8566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9756 -3.3645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3431 -3.4761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1233 -4.0797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1953 -2.7609 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7824 -2.2688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1499 -2.3804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9301 -2.9840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6886 -2.6739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7371 -1.8885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9610 -1.2732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5402 -0.7718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1046 -0.8855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3285 -1.5006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0923 -2.0022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3166 -5.1752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0208 -3.7450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7474 -0.1195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7871 -0.2803 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.4739 -0.8228 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.0762 -0.6507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6822 -0.8228 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.9955 -0.2803 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.3931 -0.4524 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.2452 -0.2803 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4302 -0.0288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8385 0.3056 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2027 -0.5801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.6278 -0.9928 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
6.5399 -0.2913 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
5.8879 -0.1910 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
5.3712 0.1001 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
5.5286 -0.4888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.1852 -0.6075 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
7.1789 -1.3788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.1541 -0.0332 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.8787 0.4973 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6830 -0.2972 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2612 -1.5507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1568 -7.4265 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
2.6376 -6.9081 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.2954 -6.3442 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.1861 -5.7611 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.8364 -6.2605 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1675 -6.8398 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.2049 -7.9755 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2302 -7.2129 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8166 -5.8946 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.8211 -1.3305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.7908 -2.3301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9450 -1.3216 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9054 -1.0427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4936 -7.1975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4579 -8.1968 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
3 19 1 0 0 0 0
20 15 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
24 30 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 40 1 0 0 0 0
40 30 1 0 0 0 0
22 41 1 0 0 0 0
41 8 1 0 0 0 0
42 43 1 1 0 0 0
43 44 1 1 0 0 0
45 44 1 1 0 0 0
45 46 1 0 0 0 0
46 47 1 0 0 0 0
47 42 1 0 0 0 0
42 48 1 0 0 0 0
43 49 1 0 0 0 0
44 50 1 0 0 0 0
45 18 1 0 0 0 0
36 51 1 0 0 0 0
51 52 1 0 0 0 0
16 53 1 0 0 0 0
53 54 1 0 0 0 0
47 55 1 0 0 0 0
55 56 1 0 0 0 0
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 55 56
M SBL 3 1 60
M SMT 3 CH2OH
M SVB 3 60 -3.6337 1.2496
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 51 52
M SBL 2 1 56
M SMT 2 CH2OH
M SVB 2 56 2.8095 -1.0393
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 53 54
M SBL 1 1 58
M SMT 1 OCH3
M SVB 1 58 2.5764 2.6308
S SKP 8
ID FL5FADGA0016
KNApSAcK_ID C00005575
NAME Isorhamnetin 3-glucosyl-(1->6)-galactoside-7-glucoside;Quercetin 3'-methyl ether 3-glucosyl-(1->6)-galactoside-7-glucoside;3-[(6-O-beta-D-Glucopyranosyl-beta-D-galactopyranosyl)oxy]-7-(beta-D-glucopyranosyloxy)-5-hydroxy-2-(4-hydroxy-3-methoxyphenyl)-4H-1-benzopyran-4-one
CAS_RN 157201-73-5
FORMULA C34H42O22
EXACTMASS 802.216773028
AVERAGEMASS 802.68408
SMILES O[C@H]([C@@H]1OC[C@@H](O2)[C@@H](C(C(O)[C@H](OC(=C(c(c6)cc(c(c6)O)OC)3)C(c(c4O)c(cc(O[C@@H]([C@H]5O)OC([C@@H]([C@H](O)5)O)CO)c4)O3)=O)2)O)O)[C@@H](O)[C@@H](O)C(O1)CO
M END