Mol:FL5FADGA0003
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 45 49 0 0 0 0 0 0 0 0999 V2000 | + | 45 49 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.8374 1.0714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8374 1.0714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8374 0.4291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8374 0.4291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2811 0.1079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2811 0.1079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7248 0.4291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7248 0.4291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7248 1.0714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7248 1.0714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2811 1.3926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2811 1.3926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1685 0.1079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1685 0.1079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6122 0.4291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6122 0.4291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6122 1.0714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6122 1.0714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1685 1.3926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1685 1.3926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1685 -0.3930 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1685 -0.3930 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0883 1.5162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0883 1.5162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6553 1.1889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6553 1.1889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2222 1.5162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2222 1.5162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2222 2.1709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2222 2.1709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6553 2.4983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6553 2.4983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0883 2.1709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0883 2.1709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2811 -0.5343 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2811 -0.5343 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0882 2.6709 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0882 2.6709 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0489 0.0195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0489 0.0195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5019 1.4551 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5019 1.4551 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5968 -1.2522 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.5968 -1.2522 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.0435 -0.9327 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.0435 -0.9327 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.2191 -1.5470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2191 -1.5470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0435 -2.1650 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.0435 -2.1650 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.5968 -2.4845 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.5968 -2.4845 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.4213 -1.8702 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.4213 -1.8702 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.4378 -0.6589 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4378 -0.6589 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9251 -1.9143 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9251 -1.9143 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4107 -2.2664 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4107 -2.2664 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5011 -0.3250 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 1.5011 -0.3250 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 1.9029 -0.8553 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.9029 -0.8553 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.4813 -0.6303 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.4813 -0.6303 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.0395 -0.6243 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.0395 -0.6243 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.6339 -0.2186 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.6339 -0.2186 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.1278 -0.4857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1278 -0.4857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2766 -0.8858 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2766 -0.8858 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8127 -1.1867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8127 -1.1867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0307 -0.7564 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0307 -0.7564 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3908 0.4349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3908 0.4349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2034 1.4172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2034 1.4172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1273 -3.0744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1273 -3.0744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6695 -2.8609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6695 -2.8609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9387 3.0744 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9387 3.0744 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3801 3.9717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3801 3.9717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 21 1 0 0 0 0 | + | 1 21 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 23 20 1 0 0 0 0 | + | 23 20 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 28 31 1 0 0 0 0 | + | 28 31 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 35 40 1 0 0 0 0 | + | 35 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 25 42 1 0 0 0 0 | + | 25 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 16 44 1 0 0 0 0 | + | 16 44 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 42 43 | + | M SAL 3 2 42 43 |
| − | M SBL 3 1 46 | + | M SBL 3 1 46 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SVB 3 46 0.1273 -3.0744 | + | M SVB 3 46 0.1273 -3.0744 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 40 41 | + | M SAL 2 2 40 41 |
| − | M SBL 2 1 44 | + | M SBL 2 1 44 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 44 3.3603 -0.7624 | + | M SVB 2 44 3.3603 -0.7624 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 44 45 | + | M SAL 1 2 44 45 |
| − | M SBL 1 1 48 | + | M SBL 1 1 48 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 48 0.9387 3.0744 | + | M SVB 1 48 0.9387 3.0744 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FADGA0003 | + | ID FL5FADGA0003 |
| − | KNApSAcK_ID C00005534 | + | KNApSAcK_ID C00005534 |
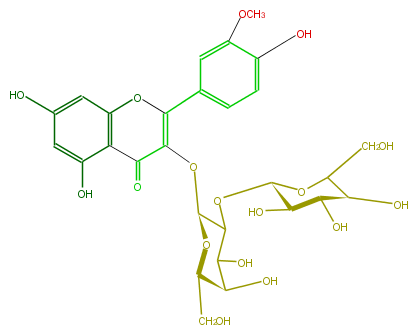
| − | NAME Isorhamnetin 3-glucosyl-(1->2)-galactoside | + | NAME Isorhamnetin 3-glucosyl-(1->2)-galactoside |
| − | CAS_RN 117383-33-2 | + | CAS_RN 117383-33-2 |
| − | FORMULA C28H32O17 | + | FORMULA C28H32O17 |
| − | EXACTMASS 640.163949598 | + | EXACTMASS 640.163949598 |
| − | AVERAGEMASS 640.54348 | + | AVERAGEMASS 640.54348 |
| − | SMILES OC[C@H]([C@H](O)1)O[C@@H](OC(=C4c(c5)cc(OC)c(O)c5)C(=O)c(c3O4)c(cc(c3)O)O)C(O[C@@H](O2)[C@H](O)[C@@H]([C@@H](C2CO)O)O)C1O | + | SMILES OC[C@H]([C@H](O)1)O[C@@H](OC(=C4c(c5)cc(OC)c(O)c5)C(=O)c(c3O4)c(cc(c3)O)O)C(O[C@@H](O2)[C@H](O)[C@@H]([C@@H](C2CO)O)O)C1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
45 49 0 0 0 0 0 0 0 0999 V2000
-2.8374 1.0714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8374 0.4291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2811 0.1079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7248 0.4291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7248 1.0714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2811 1.3926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1685 0.1079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6122 0.4291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6122 1.0714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1685 1.3926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1685 -0.3930 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0883 1.5162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6553 1.1889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2222 1.5162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2222 2.1709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6553 2.4983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0883 2.1709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2811 -0.5343 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0882 2.6709 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0489 0.0195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5019 1.4551 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5968 -1.2522 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.0435 -0.9327 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.2191 -1.5470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0435 -2.1650 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.5968 -2.4845 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.4213 -1.8702 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.4378 -0.6589 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9251 -1.9143 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4107 -2.2664 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5011 -0.3250 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
1.9029 -0.8553 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.4813 -0.6303 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.0395 -0.6243 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.6339 -0.2186 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.1278 -0.4857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2766 -0.8858 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8127 -1.1867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0307 -0.7564 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3908 0.4349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2034 1.4172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1273 -3.0744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6695 -2.8609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9387 3.0744 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3801 3.9717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
20 8 1 0 0 0 0
4 3 1 0 0 0 0
1 21 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
27 29 1 0 0 0 0
26 30 1 0 0 0 0
23 20 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
32 37 1 0 0 0 0
33 38 1 0 0 0 0
28 31 1 0 0 0 0
34 39 1 0 0 0 0
35 40 1 0 0 0 0
40 41 1 0 0 0 0
25 42 1 0 0 0 0
42 43 1 0 0 0 0
16 44 1 0 0 0 0
44 45 1 0 0 0 0
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 42 43
M SBL 3 1 46
M SMT 3 CH2OH
M SVB 3 46 0.1273 -3.0744
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 40 41
M SBL 2 1 44
M SMT 2 CH2OH
M SVB 2 44 3.3603 -0.7624
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 44 45
M SBL 1 1 48
M SMT 1 OCH3
M SVB 1 48 0.9387 3.0744
S SKP 8
ID FL5FADGA0003
KNApSAcK_ID C00005534
NAME Isorhamnetin 3-glucosyl-(1->2)-galactoside
CAS_RN 117383-33-2
FORMULA C28H32O17
EXACTMASS 640.163949598
AVERAGEMASS 640.54348
SMILES OC[C@H]([C@H](O)1)O[C@@H](OC(=C4c(c5)cc(OC)c(O)c5)C(=O)c(c3O4)c(cc(c3)O)O)C(O[C@@H](O2)[C@H](O)[C@@H]([C@@H](C2CO)O)O)C1O
M END