Mol:FL5FADGA0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.8578 0.3142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8578 0.3142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8578 -0.5076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8578 -0.5076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1461 -0.9186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1461 -0.9186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4344 -0.5076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4344 -0.5076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4344 0.3142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4344 0.3142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1461 0.7250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1461 0.7250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7228 -0.9186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7228 -0.9186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0111 -0.5076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0111 -0.5076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0111 0.3142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0111 0.3142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7228 0.7250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7228 0.7250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7228 -1.5592 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7228 -1.5592 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8851 0.8832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8851 0.8832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6105 0.4644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6105 0.4644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3357 0.8832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3357 0.8832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3357 1.7208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3357 1.7208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6105 2.1395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6105 2.1395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8851 1.7208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8851 1.7208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1461 -1.7400 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1461 -1.7400 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9500 2.1323 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9500 2.1323 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6476 -0.9341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6476 -0.9341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5622 0.7623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5622 0.7623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8708 -1.0662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8708 -1.0662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1564 -1.4787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1564 -1.4787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9567 -1.6790 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9567 -1.6790 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5345 -2.2744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5345 -2.2744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2491 -1.8619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2491 -1.8619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4487 -1.6615 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4487 -1.6615 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2564 -0.8072 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2564 -0.8072 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9174 -1.4233 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9174 -1.4233 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5622 -1.2811 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5622 -1.2811 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1247 -2.5046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1247 -2.5046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3607 -3.0837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3607 -3.0837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9229 -3.8260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9229 -3.8260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7326 -3.5111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7326 -3.5111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5139 -3.5027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5139 -3.5027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9463 -2.9348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9463 -2.9348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1192 -3.2317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1192 -3.2317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3827 -2.5092 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3827 -2.5092 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2335 -3.7462 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2335 -3.7462 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3836 -3.9612 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3836 -3.9612 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1858 -3.2192 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1858 -3.2192 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6031 2.8133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6031 2.8133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1678 3.9612 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1678 3.9612 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 21 1 0 0 0 0 | + | 1 21 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 25 31 1 0 0 0 0 | + | 25 31 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 35 41 1 0 0 0 0 | + | 35 41 1 0 0 0 0 |
| − | 41 31 1 0 0 0 0 | + | 41 31 1 0 0 0 0 |
| − | 23 20 1 0 0 0 0 | + | 23 20 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 16 42 1 0 0 0 0 | + | 16 42 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 42 43 | + | M SAL 1 2 42 43 |
| − | M SBL 1 1 47 | + | M SBL 1 1 47 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 47 0.0073 -0.6738 | + | M SBV 1 47 0.0073 -0.6738 |
| − | S SKP 5 | + | S SKP 5 |
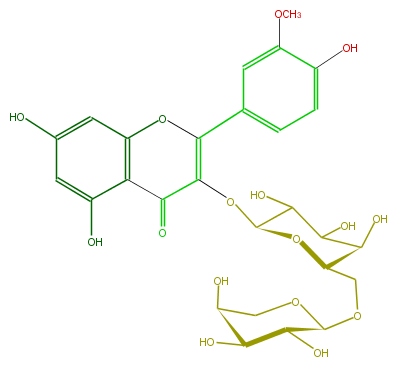
| − | ID FL5FADGA0002 | + | ID FL5FADGA0002 |
| − | FORMULA C27H30O16 | + | FORMULA C27H30O16 |
| − | EXACTMASS 610.153384912 | + | EXACTMASS 610.153384912 |
| − | AVERAGEMASS 610.5175 | + | AVERAGEMASS 610.5175 |
| − | SMILES C(O)(C(O)1)C(O)C(OC(C4=O)=C(Oc(c5)c(c(O)cc5O)4)c(c3)ccc(c3OC)O)OC(COC(O2)C(O)C(C(O)C2)O)1 | + | SMILES C(O)(C(O)1)C(O)C(OC(C4=O)=C(Oc(c5)c(c(O)cc5O)4)c(c3)ccc(c3OC)O)OC(COC(O2)C(O)C(C(O)C2)O)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-2.8578 0.3142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8578 -0.5076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1461 -0.9186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4344 -0.5076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4344 0.3142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1461 0.7250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7228 -0.9186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0111 -0.5076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0111 0.3142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7228 0.7250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7228 -1.5592 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8851 0.8832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6105 0.4644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3357 0.8832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3357 1.7208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6105 2.1395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8851 1.7208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1461 -1.7400 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9500 2.1323 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6476 -0.9341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5622 0.7623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8708 -1.0662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1564 -1.4787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9567 -1.6790 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5345 -2.2744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2491 -1.8619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4487 -1.6615 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2564 -0.8072 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9174 -1.4233 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5622 -1.2811 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1247 -2.5046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3607 -3.0837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9229 -3.8260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7326 -3.5111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5139 -3.5027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9463 -2.9348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1192 -3.2317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3827 -2.5092 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2335 -3.7462 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3836 -3.9612 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1858 -3.2192 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6031 2.8133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1678 3.9612 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
20 8 1 0 0 0 0
4 3 1 0 0 0 0
1 21 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
27 29 1 0 0 0 0
26 30 1 0 0 0 0
25 31 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 40 1 0 0 0 0
35 41 1 0 0 0 0
41 31 1 0 0 0 0
23 20 1 0 0 0 0
42 43 1 0 0 0 0
16 42 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 42 43
M SBL 1 1 47
M SMT 1 OCH3
M SBV 1 47 0.0073 -0.6738
S SKP 5
ID FL5FADGA0002
FORMULA C27H30O16
EXACTMASS 610.153384912
AVERAGEMASS 610.5175
SMILES C(O)(C(O)1)C(O)C(OC(C4=O)=C(Oc(c5)c(c(O)cc5O)4)c(c3)ccc(c3OC)O)OC(COC(O2)C(O)C(C(O)C2)O)1
M END