Mol:FL5FACNI0004
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 37 39 0 0 0 0 0 0 0 0999 V2000 | + | 37 39 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.8114 -0.1399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8114 -0.1399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8114 -0.7823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8114 -0.7823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2551 -1.1035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2551 -1.1035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6988 -0.7823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6988 -0.7823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6988 -0.1399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6988 -0.1399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2551 0.1813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2551 0.1813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1425 -1.1035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1425 -1.1035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5862 -0.7823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5862 -0.7823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5862 -0.1399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5862 -0.1399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1425 0.1813 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1425 0.1813 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1425 -1.6043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1425 -1.6043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0301 0.1811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0301 0.1811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5369 -0.1462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5369 -0.1462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1038 0.1811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1038 0.1811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1038 0.8358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1038 0.8358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5369 1.1632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5369 1.1632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0301 0.8358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0301 0.8358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0064 -1.0887 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0064 -1.0887 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2551 -1.7456 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2551 -1.7456 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6706 1.1631 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6706 1.1631 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3675 0.1811 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3675 0.1811 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5369 1.8177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5369 1.8177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2551 0.8234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2551 0.8234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8112 1.1444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8112 1.1444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6706 -0.1461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6706 -0.1461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8112 1.7866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8112 1.7866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3673 2.1076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3673 2.1076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2551 2.1076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2551 2.1076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2363 0.1805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2363 0.1805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8019 -0.1461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8019 -0.1461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3675 0.1805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3675 0.1805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8019 -0.7992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8019 -0.7992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5369 -0.8007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5369 -0.8007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1037 -1.1279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1037 -1.1279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1037 -1.7811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1037 -1.7811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6693 -2.1076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6693 -2.1076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5380 -2.1076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5380 -2.1076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 8 18 1 0 0 0 0 | + | 8 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 1 21 1 0 0 0 0 | + | 1 21 1 0 0 0 0 |
| − | 16 22 1 0 0 0 0 | + | 16 22 1 0 0 0 0 |
| − | 6 23 1 0 0 0 0 | + | 6 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 14 25 1 0 0 0 0 | + | 14 25 1 0 0 0 0 |
| − | 24 26 2 0 0 0 0 | + | 24 26 2 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 29 30 2 0 0 0 0 | + | 29 30 2 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 30 32 1 0 0 0 0 | + | 30 32 1 0 0 0 0 |
| − | 13 33 1 0 0 0 0 | + | 13 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 35 37 1 0 0 0 0 | + | 35 37 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FACNI0004 | + | ID FL5FACNI0004 |
| − | KNApSAcK_ID C00005034 | + | KNApSAcK_ID C00005034 |
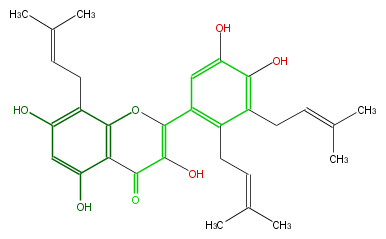
| − | NAME Broussoflavonol G | + | NAME Broussoflavonol G |
| − | CAS_RN 128341-11-7 | + | CAS_RN 128341-11-7 |
| − | FORMULA C30H34O7 | + | FORMULA C30H34O7 |
| − | EXACTMASS 506.230453442 | + | EXACTMASS 506.230453442 |
| − | AVERAGEMASS 506.58676 | + | AVERAGEMASS 506.58676 |
| − | SMILES c(c3O)c(O)c(c(c23)OC(=C(C2=O)O)c(c1)c(c(c(O)c(O)1)CC=C(C)C)CC=C(C)C)CC=C(C)C | + | SMILES c(c3O)c(O)c(c(c23)OC(=C(C2=O)O)c(c1)c(c(c(O)c(O)1)CC=C(C)C)CC=C(C)C)CC=C(C)C |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
37 39 0 0 0 0 0 0 0 0999 V2000
-2.8114 -0.1399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8114 -0.7823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2551 -1.1035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6988 -0.7823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6988 -0.1399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2551 0.1813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1425 -1.1035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5862 -0.7823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5862 -0.1399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1425 0.1813 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1425 -1.6043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0301 0.1811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5369 -0.1462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1038 0.1811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1038 0.8358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5369 1.1632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0301 0.8358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0064 -1.0887 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2551 -1.7456 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6706 1.1631 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3675 0.1811 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5369 1.8177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2551 0.8234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8112 1.1444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6706 -0.1461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8112 1.7866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3673 2.1076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2551 2.1076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2363 0.1805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8019 -0.1461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3675 0.1805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8019 -0.7992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5369 -0.8007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1037 -1.1279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1037 -1.7811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6693 -2.1076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5380 -2.1076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
8 18 1 0 0 0 0
3 19 1 0 0 0 0
15 20 1 0 0 0 0
1 21 1 0 0 0 0
16 22 1 0 0 0 0
6 23 1 0 0 0 0
23 24 1 0 0 0 0
14 25 1 0 0 0 0
24 26 2 0 0 0 0
26 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
29 30 2 0 0 0 0
30 31 1 0 0 0 0
30 32 1 0 0 0 0
13 33 1 0 0 0 0
33 34 1 0 0 0 0
34 35 2 0 0 0 0
35 36 1 0 0 0 0
35 37 1 0 0 0 0
S SKP 8
ID FL5FACNI0004
KNApSAcK_ID C00005034
NAME Broussoflavonol G
CAS_RN 128341-11-7
FORMULA C30H34O7
EXACTMASS 506.230453442
AVERAGEMASS 506.58676
SMILES c(c3O)c(O)c(c(c23)OC(=C(C2=O)O)c(c1)c(c(c(O)c(O)1)CC=C(C)C)CC=C(C)C)CC=C(C)C
M END