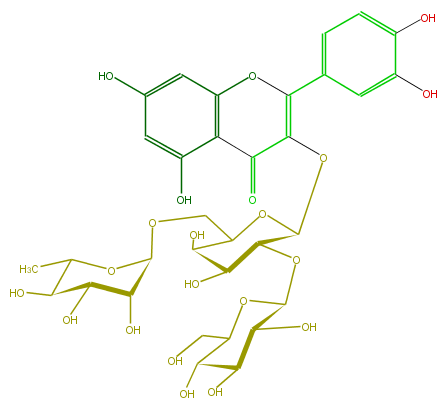
Mol:FL5FACGS0078
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 54 59 0 0 0 0 0 0 0 0999 V2000 | + | 54 59 0 0 0 0 0 0 0 0999 V2000 |
| − | 2.0039 3.3832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0039 3.3832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0039 2.5582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0039 2.5582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7183 2.1457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7183 2.1457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4328 2.5582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4328 2.5582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4328 3.3832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4328 3.3832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7183 3.7957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7183 3.7957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2894 2.1457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2894 2.1457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5750 2.5582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5750 2.5582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1395 2.1457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1395 2.1457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1395 1.3208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1395 1.3208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5750 0.9083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5750 0.9083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2894 1.3208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2894 1.3208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8539 2.5582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8539 2.5582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5684 2.1457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5684 2.1457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5684 1.3208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5684 1.3208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8539 0.9083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8539 0.9083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5750 0.0833 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5750 0.0833 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2829 2.5582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2829 2.5582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0039 0.9083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0039 0.9083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8539 0.0833 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8539 0.0833 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0247 3.7249 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0247 3.7249 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0614 2.1953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0614 2.1953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6288 -0.9395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6288 -0.9395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0728 -1.3740 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0728 -1.3740 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6772 -0.8123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6772 -0.8123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4836 -0.6370 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4836 -0.6370 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7821 -0.2023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7821 -0.2023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1776 -0.7640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1776 -0.7640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3781 -0.2636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3781 -0.2636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4158 -0.3801 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4158 -0.3801 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6004 -0.6161 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6004 -0.6161 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5698 -1.6023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5698 -1.6023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4590 -1.1278 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4590 -1.1278 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0466 -1.1376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0466 -1.1376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4592 -1.8523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4592 -1.8523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6657 -1.6255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6657 -1.6255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8674 -1.8348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8674 -1.8348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4547 -1.1201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4547 -1.1201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2483 -1.3468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2483 -1.3468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8721 -2.5327 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8721 -2.5327 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1405 -2.3320 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1405 -2.3320 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0614 -1.7911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0614 -1.7911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7810 -1.3031 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7810 -1.3031 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5402 -3.2213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5402 -3.2213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2806 -3.3067 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2806 -3.3067 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5798 -2.5376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5798 -2.5376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2291 -2.0283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2291 -2.0283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4084 -1.9428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4084 -1.9428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1090 -2.7119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1090 -2.7119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4340 -2.6630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4340 -2.6630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0417 -3.1377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0417 -3.1377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8011 -3.7957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8011 -3.7957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2324 -3.7668 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2324 -3.7668 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6394 -2.4566 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6394 -2.4566 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 5 21 1 0 0 0 0 | + | 5 21 1 0 0 0 0 |
| − | 4 22 1 0 0 0 0 | + | 4 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 33 1 0 0 0 0 | + | 25 33 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 37 36 1 1 0 0 0 | + | 37 36 1 1 0 0 0 |
| − | 38 37 1 1 0 0 0 | + | 38 37 1 1 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 37 40 1 0 0 0 0 | + | 37 40 1 0 0 0 0 |
| − | 36 41 1 0 0 0 0 | + | 36 41 1 0 0 0 0 |
| − | 35 42 1 0 0 0 0 | + | 35 42 1 0 0 0 0 |
| − | 34 43 1 0 0 0 0 | + | 34 43 1 0 0 0 0 |
| − | 38 30 1 0 0 0 0 | + | 38 30 1 0 0 0 0 |
| − | 44 45 1 1 0 0 0 | + | 44 45 1 1 0 0 0 |
| − | 45 46 1 1 0 0 0 | + | 45 46 1 1 0 0 0 |
| − | 47 46 1 1 0 0 0 | + | 47 46 1 1 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 44 1 0 0 0 0 | + | 49 44 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 44 52 1 0 0 0 0 | + | 44 52 1 0 0 0 0 |
| − | 45 53 1 0 0 0 0 | + | 45 53 1 0 0 0 0 |
| − | 46 54 1 0 0 0 0 | + | 46 54 1 0 0 0 0 |
| − | 47 33 1 0 0 0 0 | + | 47 33 1 0 0 0 0 |
| − | 26 19 1 0 0 0 0 | + | 26 19 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FACGS0078 | + | ID FL5FACGS0078 |
| − | FORMULA C33H40O21 | + | FORMULA C33H40O21 |
| − | EXACTMASS 772.206208342 | + | EXACTMASS 772.206208342 |
| − | AVERAGEMASS 772.6581 | + | AVERAGEMASS 772.6581 |
| − | SMILES C(O)C(O1)C(C(O)C(C(OC(C3OC(=C(c(c6)cc(c(c6)O)O)5)C(=O)c(c(O5)4)c(cc(O)c4)O)C(C(O)C(O3)COC(C(O)2)OC(C)C(O)C(O)2)O)1)O)O | + | SMILES C(O)C(O1)C(C(O)C(C(OC(C3OC(=C(c(c6)cc(c(c6)O)O)5)C(=O)c(c(O5)4)c(cc(O)c4)O)C(C(O)C(O3)COC(C(O)2)OC(C)C(O)C(O)2)O)1)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
54 59 0 0 0 0 0 0 0 0999 V2000
2.0039 3.3832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0039 2.5582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7183 2.1457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4328 2.5582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4328 3.3832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7183 3.7957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2894 2.1457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5750 2.5582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1395 2.1457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1395 1.3208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5750 0.9083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2894 1.3208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8539 2.5582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5684 2.1457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5684 1.3208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8539 0.9083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5750 0.0833 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2829 2.5582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0039 0.9083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8539 0.0833 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0247 3.7249 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0614 2.1953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6288 -0.9395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0728 -1.3740 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6772 -0.8123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4836 -0.6370 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7821 -0.2023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1776 -0.7640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3781 -0.2636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4158 -0.3801 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6004 -0.6161 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5698 -1.6023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4590 -1.1278 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0466 -1.1376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4592 -1.8523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6657 -1.6255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8674 -1.8348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4547 -1.1201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2483 -1.3468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8721 -2.5327 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1405 -2.3320 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0614 -1.7911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7810 -1.3031 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5402 -3.2213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2806 -3.3067 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5798 -2.5376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2291 -2.0283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4084 -1.9428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1090 -2.7119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4340 -2.6630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0417 -3.1377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8011 -3.7957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2324 -3.7668 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6394 -2.4566 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
16 20 1 0 0 0 0
5 21 1 0 0 0 0
4 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
28 29 1 0 0 0 0
29 30 1 0 0 0 0
23 31 1 0 0 0 0
24 32 1 0 0 0 0
25 33 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 1 0 0 0
37 36 1 1 0 0 0
38 37 1 1 0 0 0
38 39 1 0 0 0 0
39 34 1 0 0 0 0
37 40 1 0 0 0 0
36 41 1 0 0 0 0
35 42 1 0 0 0 0
34 43 1 0 0 0 0
38 30 1 0 0 0 0
44 45 1 1 0 0 0
45 46 1 1 0 0 0
47 46 1 1 0 0 0
47 48 1 0 0 0 0
48 49 1 0 0 0 0
49 44 1 0 0 0 0
49 50 1 0 0 0 0
50 51 1 0 0 0 0
44 52 1 0 0 0 0
45 53 1 0 0 0 0
46 54 1 0 0 0 0
47 33 1 0 0 0 0
26 19 1 0 0 0 0
S SKP 5
ID FL5FACGS0078
FORMULA C33H40O21
EXACTMASS 772.206208342
AVERAGEMASS 772.6581
SMILES C(O)C(O1)C(C(O)C(C(OC(C3OC(=C(c(c6)cc(c(c6)O)O)5)C(=O)c(c(O5)4)c(cc(O)c4)O)C(C(O)C(O3)COC(C(O)2)OC(C)C(O)C(O)2)O)1)O)O
M END