Mol:FL5FACGS0023
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 40 44 0 0 0 0 0 0 0 0999 V2000 | + | 40 44 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.8956 1.0045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8956 1.0045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6101 1.4170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6101 1.4170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6101 2.2420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6101 2.2420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8956 2.6545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8956 2.6545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1810 2.2420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1810 2.2420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1810 1.4170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1810 1.4170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8956 3.2403 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8956 3.2403 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2073 2.5868 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2073 2.5868 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4441 0.9914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4441 0.9914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3266 1.4364 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3266 1.4364 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0975 0.9914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0975 0.9914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0975 0.1015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0975 0.1015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3266 -0.3435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3266 -0.3435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4441 0.1015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4441 0.1015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8941 1.4514 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8941 1.4514 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6909 0.9914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6909 0.9914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6909 0.0715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6909 0.0715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8941 -0.3885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8941 -0.3885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2073 1.2896 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2073 1.2896 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8941 -0.9851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8941 -0.9851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3266 -0.8727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3266 -0.8727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0471 -0.2466 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0471 -0.2466 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6697 -1.1868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6697 -1.1868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9154 -0.9847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9154 -0.9847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3170 -1.6545 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3170 -1.6545 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3212 -2.4352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3212 -2.4352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0755 -2.6374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0755 -2.6374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6740 -1.9676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6740 -1.9676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2526 -2.3017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2526 -2.3017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9139 -3.2403 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9139 -3.2403 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4484 -0.5348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4484 -0.5348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7307 -0.1269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7307 -0.1269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0695 0.1838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0695 0.1838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4156 0.1838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4156 0.1838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7522 -0.1269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7522 -0.1269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2432 0.4069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2432 0.4069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7307 0.7305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7307 0.7305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7522 0.7305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7522 0.7305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7522 -0.7568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7522 -0.7568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8053 -0.7568 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8053 -0.7568 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 3 8 1 0 0 0 0 | + | 3 8 1 0 0 0 0 |
| − | 6 9 1 0 0 0 0 | + | 6 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 9 2 0 0 0 0 | + | 14 9 2 0 0 0 0 |
| − | 11 15 1 0 0 0 0 | + | 11 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 12 1 0 0 0 0 | + | 18 12 1 0 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 18 20 1 0 0 0 0 | + | 18 20 1 0 0 0 0 |
| − | 13 21 2 0 0 0 0 | + | 13 21 2 0 0 0 0 |
| − | 14 22 1 0 0 0 0 | + | 14 22 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 22 24 1 0 0 0 0 | + | 22 24 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 27 30 1 0 0 0 0 | + | 27 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 35 32 1 1 0 0 0 | + | 35 32 1 1 0 0 0 |
| − | 34 32 1 1 0 0 0 | + | 34 32 1 1 0 0 0 |
| − | 33 35 1 1 0 0 0 | + | 33 35 1 1 0 0 0 |
| − | 36 33 1 0 0 0 0 | + | 36 33 1 0 0 0 0 |
| − | 34 36 1 0 0 0 0 | + | 34 36 1 0 0 0 0 |
| − | 31 34 1 0 0 0 0 | + | 31 34 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 35 38 1 0 0 0 0 | + | 35 38 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 39 40 | + | M SAL 1 2 39 40 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 44 0.0000 0.6299 | + | M SBV 1 44 0.0000 0.6299 |
| − | S SKP 5 | + | S SKP 5 |
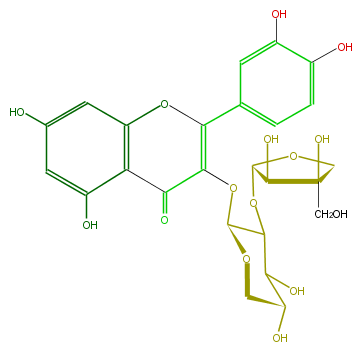
| − | ID FL5FACGS0023 | + | ID FL5FACGS0023 |
| − | FORMULA C25H26O15 | + | FORMULA C25H26O15 |
| − | EXACTMASS 566.127170162 | + | EXACTMASS 566.127170162 |
| − | AVERAGEMASS 566.46494 | + | AVERAGEMASS 566.46494 |
| − | SMILES C(C(O)5)(C(OCC5O)OC(C2=O)=C(c(c4)cc(c(c4)O)O)Oc(c3)c2c(O)cc3O)OC(C(O)1)OCC1(O)CO | + | SMILES C(C(O)5)(C(OCC5O)OC(C2=O)=C(c(c4)cc(c(c4)O)O)Oc(c3)c2c(O)cc3O)OC(C(O)1)OCC1(O)CO |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
40 44 0 0 0 0 0 0 0 0999 V2000
1.8956 1.0045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6101 1.4170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6101 2.2420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8956 2.6545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1810 2.2420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1810 1.4170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8956 3.2403 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2073 2.5868 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4441 0.9914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3266 1.4364 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0975 0.9914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0975 0.1015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3266 -0.3435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4441 0.1015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8941 1.4514 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6909 0.9914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6909 0.0715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8941 -0.3885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2073 1.2896 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8941 -0.9851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3266 -0.8727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0471 -0.2466 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6697 -1.1868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9154 -0.9847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3170 -1.6545 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3212 -2.4352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0755 -2.6374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6740 -1.9676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2526 -2.3017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9139 -3.2403 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4484 -0.5348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7307 -0.1269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0695 0.1838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4156 0.1838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7522 -0.1269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2432 0.4069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7307 0.7305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7522 0.7305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7522 -0.7568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8053 -0.7568 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
3 8 1 0 0 0 0
6 9 1 0 0 0 0
9 10 1 0 0 0 0
10 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 1 0 0 0 0
14 9 2 0 0 0 0
11 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 12 1 0 0 0 0
16 19 1 0 0 0 0
18 20 1 0 0 0 0
13 21 2 0 0 0 0
14 22 1 0 0 0 0
23 24 1 0 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
27 26 1 1 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
22 24 1 0 0 0 0
28 29 1 0 0 0 0
27 30 1 0 0 0 0
23 31 1 0 0 0 0
35 32 1 1 0 0 0
34 32 1 1 0 0 0
33 35 1 1 0 0 0
36 33 1 0 0 0 0
34 36 1 0 0 0 0
31 34 1 0 0 0 0
32 37 1 0 0 0 0
35 38 1 0 0 0 0
39 40 1 0 0 0 0
35 39 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 39 40
M SBL 1 1 44
M SMT 1 CH2OH
M SBV 1 44 0.0000 0.6299
S SKP 5
ID FL5FACGS0023
FORMULA C25H26O15
EXACTMASS 566.127170162
AVERAGEMASS 566.46494
SMILES C(C(O)5)(C(OCC5O)OC(C2=O)=C(c(c4)cc(c(c4)O)O)Oc(c3)c2c(O)cc3O)OC(C(O)1)OCC1(O)CO
M END