Mol:FL5FACGS0016
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 34 37 0 0 0 0 0 0 0 0999 V2000 | + | 34 37 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.7330 -0.2829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7330 -0.2829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7330 -1.1079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7330 -1.1079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0185 -1.5205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0185 -1.5205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6961 -1.1079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6961 -1.1079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6961 -0.2829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6961 -0.2829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0185 0.1297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0185 0.1297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4106 -1.5205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4106 -1.5205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1251 -1.1079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1251 -1.1079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1251 -0.2829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1251 -0.2829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4106 0.1297 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4106 0.1297 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4106 -2.2315 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4106 -2.2315 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8394 0.1295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8394 0.1295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5677 -0.2909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5677 -0.2909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2960 0.1295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2960 0.1295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2960 0.9705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2960 0.9705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5677 1.3909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5677 1.3909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8394 0.9705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8394 0.9705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4473 0.1295 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4473 0.1295 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0185 -2.2128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0185 -2.2128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9180 1.5060 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9180 1.5060 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8394 -1.5203 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8394 -1.5203 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9827 0.0697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9827 0.0697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5059 -0.5597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5059 -0.5597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8194 -0.2926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8194 -0.2926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1570 -0.2856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1570 -0.2856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6383 0.1960 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6383 0.1960 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2390 -0.1210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2390 -0.1210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7650 -0.2097 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7650 -0.2097 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1859 -0.5148 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1859 -0.5148 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1495 -0.6794 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1495 -0.6794 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5677 2.2315 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5677 2.2315 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8584 0.4983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8584 0.4983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9180 0.4983 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9180 0.4983 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3285 1.4160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3285 1.4160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 15 1 0 0 0 0 | + | 20 15 1 0 0 0 0 |
| − | 8 21 1 0 0 0 0 | + | 8 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 25 18 1 0 0 0 0 | + | 25 18 1 0 0 0 0 |
| − | 16 31 1 0 0 0 0 | + | 16 31 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 27 32 1 0 0 0 0 | + | 27 32 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 3 32 33 34 | + | M SAL 1 3 32 33 34 |
| − | M SBL 1 1 37 | + | M SBL 1 1 37 |
| − | M SMT 1 ^COOH | + | M SMT 1 ^COOH |
| − | M SBV 1 37 0.6193 -0.6193 | + | M SBV 1 37 0.6193 -0.6193 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FACGS0016 | + | ID FL5FACGS0016 |
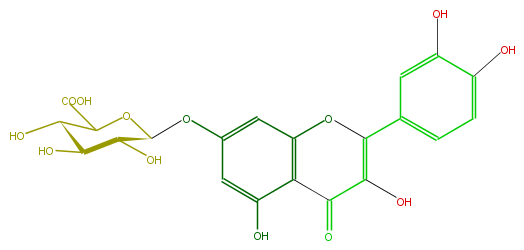
| − | FORMULA C21H18O13 | + | FORMULA C21H18O13 |
| − | EXACTMASS 478.07474066199995 | + | EXACTMASS 478.07474066199995 |
| − | AVERAGEMASS 478.35982 | + | AVERAGEMASS 478.35982 |
| − | SMILES c(c3)(OC(C(O)4)OC(C(O)=O)C(O)C4O)cc(c1c3O)OC(c(c2)cc(c(O)c2)O)=C(C(=O)1)O | + | SMILES c(c3)(OC(C(O)4)OC(C(O)=O)C(O)C4O)cc(c1c3O)OC(c(c2)cc(c(O)c2)O)=C(C(=O)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
34 37 0 0 0 0 0 0 0 0999 V2000
-0.7330 -0.2829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7330 -1.1079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0185 -1.5205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6961 -1.1079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6961 -0.2829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0185 0.1297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4106 -1.5205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1251 -1.1079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1251 -0.2829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4106 0.1297 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4106 -2.2315 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8394 0.1295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5677 -0.2909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2960 0.1295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2960 0.9705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5677 1.3909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8394 0.9705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4473 0.1295 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0185 -2.2128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9180 1.5060 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8394 -1.5203 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9827 0.0697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5059 -0.5597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8194 -0.2926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1570 -0.2856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6383 0.1960 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2390 -0.1210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7650 -0.2097 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1859 -0.5148 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1495 -0.6794 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5677 2.2315 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8584 0.4983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9180 0.4983 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3285 1.4160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
3 19 1 0 0 0 0
20 15 1 0 0 0 0
8 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
25 18 1 0 0 0 0
16 31 1 0 0 0 0
32 33 2 0 0 0 0
32 34 1 0 0 0 0
27 32 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 3 32 33 34
M SBL 1 1 37
M SMT 1 ^COOH
M SBV 1 37 0.6193 -0.6193
S SKP 5
ID FL5FACGS0016
FORMULA C21H18O13
EXACTMASS 478.07474066199995
AVERAGEMASS 478.35982
SMILES c(c3)(OC(C(O)4)OC(C(O)=O)C(O)C4O)cc(c1c3O)OC(c(c2)cc(c(O)c2)O)=C(C(=O)1)O
M END