Mol:FL5FACGLS001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 37 40 0 0 0 0 0 0 0 0999 V2000 | + | 37 40 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.1034 -2.2077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1034 -2.2077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6114 -2.6206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6114 -2.6206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0078 -2.4008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0078 -2.4008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8962 -1.7683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8962 -1.7683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3883 -1.3554 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3883 -1.3554 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9919 -1.5751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9919 -1.5751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2926 -1.5485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2926 -1.5485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1811 -0.9160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1811 -0.9160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6731 -0.5031 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6731 -0.5031 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2767 -0.7228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2767 -0.7228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0911 -1.8704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0911 -1.8704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5636 0.3193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5636 0.3193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0516 0.5433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0516 0.5433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1653 1.1880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1653 1.1880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3362 1.6089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3362 1.6089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9515 1.3850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9515 1.3850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0652 0.7402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0652 0.7402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8245 -2.4701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8245 -2.4701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3634 2.1973 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3634 2.1973 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5766 -2.7625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5766 -2.7625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1578 -0.6296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1578 -0.6296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7579 -2.0832 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7579 -2.0832 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5518 -1.6412 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5518 -1.6412 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9210 -1.5423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9210 -1.5423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3686 -1.2485 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3686 -1.2485 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0942 -1.8545 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0942 -1.8545 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9883 -0.4214 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 0.9883 -0.4214 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 1.3900 -0.9516 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.3900 -0.9516 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.9684 -0.7267 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.9684 -0.7267 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.5266 -0.7206 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.5266 -0.7206 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.1210 -0.3150 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.1210 -0.3150 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.6149 -0.5821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6149 -0.5821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7638 -0.9821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7638 -0.9821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5266 -0.7206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5266 -0.7206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4529 1.8057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4529 1.8057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3517 -0.0729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3517 -0.0729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0696 0.8865 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0696 0.8865 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 20 3 1 0 0 0 0 | + | 20 3 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 2 0 0 0 0 | + | 23 24 2 0 0 0 0 |
| − | 23 25 1 0 0 0 0 | + | 23 25 1 0 0 0 0 |
| − | 23 26 2 0 0 0 0 | + | 23 26 2 0 0 0 0 |
| − | 8 21 1 0 0 0 0 | + | 8 21 1 0 0 0 0 |
| − | 27 28 1 1 0 0 0 | + | 27 28 1 1 0 0 0 |
| − | 28 29 1 1 0 0 0 | + | 28 29 1 1 0 0 0 |
| − | 30 29 1 1 0 0 0 | + | 30 29 1 1 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 27 1 0 0 0 0 | + | 32 27 1 0 0 0 0 |
| − | 28 33 1 0 0 0 0 | + | 28 33 1 0 0 0 0 |
| − | 30 34 1 0 0 0 0 | + | 30 34 1 0 0 0 0 |
| − | 27 21 1 0 0 0 0 | + | 27 21 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 16 35 1 0 0 0 0 | + | 16 35 1 0 0 0 0 |
| − | 31 36 1 0 0 0 0 | + | 31 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 36 37 | + | M SAL 1 2 36 37 |
| − | M SBL 1 1 39 | + | M SBL 1 1 39 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 39 2.4547 -0.3361 | + | M SVB 1 39 2.4547 -0.3361 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FACGLS001 | + | ID FL5FACGLS001 |
| − | KNApSAcK_ID C00006068 | + | KNApSAcK_ID C00006068 |
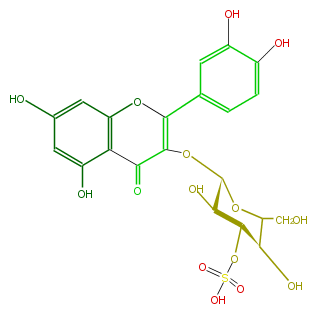
| − | NAME Quercetin 3-(3''-sulfatoglucoside) | + | NAME Quercetin 3-(3''-sulfatoglucoside) |
| − | CAS_RN 86203-13-6 | + | CAS_RN 86203-13-6 |
| − | FORMULA C21H20O15S | + | FORMULA C21H20O15S |
| − | EXACTMASS 544.0522906599999 | + | EXACTMASS 544.0522906599999 |
| − | AVERAGEMASS 544.4404999999999 | + | AVERAGEMASS 544.4404999999999 |
| − | SMILES OCC([C@@H](O)1)O[C@H](OC(C(=O)3)=C(Oc(c4)c3c(cc(O)4)O)c(c2)cc(c(O)c2)O)[C@H](O)[C@@H]1OS(O)(=O)=O | + | SMILES OCC([C@@H](O)1)O[C@H](OC(C(=O)3)=C(Oc(c4)c3c(cc(O)4)O)c(c2)cc(c(O)c2)O)[C@H](O)[C@@H]1OS(O)(=O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
37 40 0 0 0 0 0 0 0 0999 V2000
-2.1034 -2.2077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6114 -2.6206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0078 -2.4008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8962 -1.7683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3883 -1.3554 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9919 -1.5751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2926 -1.5485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1811 -0.9160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6731 -0.5031 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2767 -0.7228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0911 -1.8704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5636 0.3193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0516 0.5433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1653 1.1880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3362 1.6089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9515 1.3850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0652 0.7402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8245 -2.4701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3634 2.1973 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5766 -2.7625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1578 -0.6296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7579 -2.0832 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5518 -1.6412 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0
2.9210 -1.5423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3686 -1.2485 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0942 -1.8545 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9883 -0.4214 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
1.3900 -0.9516 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.9684 -0.7267 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.5266 -0.7206 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.1210 -0.3150 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.6149 -0.5821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7638 -0.9821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5266 -0.7206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4529 1.8057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3517 -0.0729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0696 0.8865 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
4 3 1 0 0 0 0
1 18 1 0 0 0 0
15 19 1 0 0 0 0
20 3 1 0 0 0 0
22 23 1 0 0 0 0
23 24 2 0 0 0 0
23 25 1 0 0 0 0
23 26 2 0 0 0 0
8 21 1 0 0 0 0
27 28 1 1 0 0 0
28 29 1 1 0 0 0
30 29 1 1 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
32 27 1 0 0 0 0
28 33 1 0 0 0 0
30 34 1 0 0 0 0
27 21 1 0 0 0 0
25 29 1 0 0 0 0
16 35 1 0 0 0 0
31 36 1 0 0 0 0
36 37 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 36 37
M SBL 1 1 39
M SMT 1 CH2OH
M SVB 1 39 2.4547 -0.3361
S SKP 8
ID FL5FACGLS001
KNApSAcK_ID C00006068
NAME Quercetin 3-(3''-sulfatoglucoside)
CAS_RN 86203-13-6
FORMULA C21H20O15S
EXACTMASS 544.0522906599999
AVERAGEMASS 544.4404999999999
SMILES OCC([C@@H](O)1)O[C@H](OC(C(=O)3)=C(Oc(c4)c3c(cc(O)4)O)c(c2)cc(c(O)c2)O)[C@H](O)[C@@H]1OS(O)(=O)=O
M END