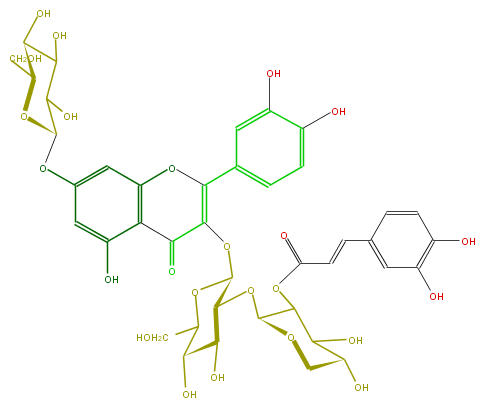
Mol:FL5FACGL0071
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 65 71 0 0 0 0 0 0 0 0999 V2000 | + | 65 71 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.0909 0.3779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0909 0.3779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0909 -0.4475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0909 -0.4475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3762 -0.8601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3762 -0.8601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6615 -0.4475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6615 -0.4475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6615 0.3779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6615 0.3779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3762 0.7905 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3762 0.7905 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9467 -0.8601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9467 -0.8601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2320 -0.4475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2320 -0.4475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2320 0.3779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2320 0.3779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9467 0.7905 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9467 0.7905 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9467 -1.5035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9467 -1.5035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4824 0.7904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4824 0.7904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2107 0.3698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2107 0.3698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9392 0.7904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9392 0.7904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9392 1.6314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9392 1.6314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2107 2.0520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2107 2.0520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4824 1.6314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4824 1.6314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8053 0.7904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8053 0.7904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6674 2.0519 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6674 2.0519 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2743 -0.9471 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2743 -0.9471 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3762 -1.6851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3762 -1.6851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2107 2.8929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2107 2.8929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6980 -3.4118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6980 -3.4118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0485 -3.0937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0485 -3.0937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0128 -2.3296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0128 -2.3296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3862 -1.6703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3862 -1.6703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4341 -1.9598 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4341 -1.9598 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3616 -2.7559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3616 -2.7559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6347 -4.2270 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6347 -4.2270 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0326 -3.8606 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0326 -3.8606 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8026 -1.9298 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8026 -1.9298 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7639 -2.3564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7639 -2.3564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9712 -2.5688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9712 -2.5688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6887 -2.9674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6887 -2.9674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0906 -3.6882 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0906 -3.6882 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8835 -3.4759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8835 -3.4759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1659 -3.0772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1659 -3.0772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0419 -3.0293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0419 -3.0293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1775 -4.0682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1775 -4.0682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8960 -1.3436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8960 -1.3436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3589 -1.8835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3589 -1.8835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5286 -0.7072 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5286 -0.7072 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5706 -1.3436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5706 -1.3436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8623 -0.8385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8623 -0.8385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4452 -0.8385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4452 -0.8385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7956 -1.4455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7956 -1.4455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4966 -1.4455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4966 -1.4455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8471 -0.8385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8471 -0.8385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4966 -0.2314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4966 -0.2314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7956 -0.2314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7956 -0.2314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5466 -0.8385 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5466 -0.8385 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2462 1.9269 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2462 1.9269 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5166 1.5056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5166 1.5056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7481 2.3158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7481 2.3158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5166 3.1307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5166 3.1307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2462 3.5521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2462 3.5521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0148 2.7419 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0148 2.7419 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8898 4.2270 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8898 4.2270 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5343 3.7222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5343 3.7222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2927 1.9427 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2927 1.9427 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8463 -2.0513 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8463 -2.0513 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5935 3.2273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5935 3.2273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5466 2.3657 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5466 2.3657 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9760 -2.9693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9760 -2.9693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1326 -3.5286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1326 -3.5286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 3 21 1 0 0 0 0 | + | 3 21 1 0 0 0 0 |
| − | 16 22 1 0 0 0 0 | + | 16 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 25 31 1 0 0 0 0 | + | 25 31 1 0 0 0 0 |
| − | 26 20 1 0 0 0 0 | + | 26 20 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 36 39 1 0 0 0 0 | + | 36 39 1 0 0 0 0 |
| − | 33 31 1 0 0 0 0 | + | 33 31 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 32 1 0 0 0 0 | + | 41 32 1 0 0 0 0 |
| − | 40 42 2 0 0 0 0 | + | 40 42 2 0 0 0 0 |
| − | 40 43 1 0 0 0 0 | + | 40 43 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 2 0 0 0 0 | + | 45 46 2 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 2 0 0 0 0 | + | 47 48 2 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 50 45 1 0 0 0 0 | + | 50 45 1 0 0 0 0 |
| − | 48 51 1 0 0 0 0 | + | 48 51 1 0 0 0 0 |
| − | 53 52 1 1 0 0 0 | + | 53 52 1 1 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 56 57 1 1 0 0 0 | + | 56 57 1 1 0 0 0 |
| − | 57 52 1 1 0 0 0 | + | 57 52 1 1 0 0 0 |
| − | 56 58 1 0 0 0 0 | + | 56 58 1 0 0 0 0 |
| − | 55 59 1 0 0 0 0 | + | 55 59 1 0 0 0 0 |
| − | 54 60 1 0 0 0 0 | + | 54 60 1 0 0 0 0 |
| − | 18 53 1 0 0 0 0 | + | 18 53 1 0 0 0 0 |
| − | 47 61 1 0 0 0 0 | + | 47 61 1 0 0 0 0 |
| − | 62 63 1 0 0 0 0 | + | 62 63 1 0 0 0 0 |
| − | 57 62 1 0 0 0 0 | + | 57 62 1 0 0 0 0 |
| − | 64 65 1 0 0 0 0 | + | 64 65 1 0 0 0 0 |
| − | 28 64 1 0 0 0 0 | + | 28 64 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 62 63 | + | M SAL 1 2 62 63 |
| − | M SBL 1 1 69 | + | M SBL 1 1 69 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 69 0.5786 -0.4853 | + | M SBV 1 69 0.5786 -0.4853 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 64 65 | + | M SAL 2 2 64 65 |
| − | M SBL 2 1 71 | + | M SBL 2 1 71 |
| − | M SMT 2 ^ CH2OH | + | M SMT 2 ^ CH2OH |
| − | M SBV 2 71 0.6144 0.2134 | + | M SBV 2 71 0.6144 0.2134 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FACGL0071 | + | ID FL5FACGL0071 |
| − | FORMULA C41H44O24 | + | FORMULA C41H44O24 |
| − | EXACTMASS 920.222252336 | + | EXACTMASS 920.222252336 |
| − | AVERAGEMASS 920.77366 | + | AVERAGEMASS 920.77366 |
| − | SMILES C(OC(C(OC(O7)C(C(C(C7)O)O)OC(C=Cc(c6)cc(O)c(O)c6)=O)5)OC(CO)C(C(O)5)O)(C(=O)2)=C(Oc(c3)c2c(cc(OC(C(O)4)OC(CO)C(O)C4O)3)O)c(c1)ccc(O)c1O | + | SMILES C(OC(C(OC(O7)C(C(C(C7)O)O)OC(C=Cc(c6)cc(O)c(O)c6)=O)5)OC(CO)C(C(O)5)O)(C(=O)2)=C(Oc(c3)c2c(cc(OC(C(O)4)OC(CO)C(O)C4O)3)O)c(c1)ccc(O)c1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
65 71 0 0 0 0 0 0 0 0999 V2000
-3.0909 0.3779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0909 -0.4475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3762 -0.8601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6615 -0.4475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6615 0.3779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3762 0.7905 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9467 -0.8601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2320 -0.4475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2320 0.3779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9467 0.7905 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9467 -1.5035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4824 0.7904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2107 0.3698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9392 0.7904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9392 1.6314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2107 2.0520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4824 1.6314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8053 0.7904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6674 2.0519 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2743 -0.9471 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3762 -1.6851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2107 2.8929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6980 -3.4118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0485 -3.0937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0128 -2.3296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3862 -1.6703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4341 -1.9598 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3616 -2.7559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6347 -4.2270 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0326 -3.8606 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8026 -1.9298 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7639 -2.3564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9712 -2.5688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6887 -2.9674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0906 -3.6882 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8835 -3.4759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1659 -3.0772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0419 -3.0293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1775 -4.0682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8960 -1.3436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3589 -1.8835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5286 -0.7072 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5706 -1.3436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8623 -0.8385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4452 -0.8385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7956 -1.4455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4966 -1.4455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8471 -0.8385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4966 -0.2314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7956 -0.2314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5466 -0.8385 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2462 1.9269 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5166 1.5056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7481 2.3158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5166 3.1307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2462 3.5521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0148 2.7419 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8898 4.2270 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5343 3.7222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2927 1.9427 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8463 -2.0513 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5935 3.2273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.5466 2.3657 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9760 -2.9693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1326 -3.5286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
15 19 1 0 0 0 0
8 20 1 0 0 0 0
3 21 1 0 0 0 0
16 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
25 31 1 0 0 0 0
26 20 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
37 38 1 0 0 0 0
36 39 1 0 0 0 0
33 31 1 0 0 0 0
40 41 1 0 0 0 0
41 32 1 0 0 0 0
40 42 2 0 0 0 0
40 43 1 0 0 0 0
43 44 2 0 0 0 0
44 45 1 0 0 0 0
45 46 2 0 0 0 0
46 47 1 0 0 0 0
47 48 2 0 0 0 0
48 49 1 0 0 0 0
49 50 2 0 0 0 0
50 45 1 0 0 0 0
48 51 1 0 0 0 0
53 52 1 1 0 0 0
53 54 1 0 0 0 0
54 55 1 0 0 0 0
55 56 1 0 0 0 0
56 57 1 1 0 0 0
57 52 1 1 0 0 0
56 58 1 0 0 0 0
55 59 1 0 0 0 0
54 60 1 0 0 0 0
18 53 1 0 0 0 0
47 61 1 0 0 0 0
62 63 1 0 0 0 0
57 62 1 0 0 0 0
64 65 1 0 0 0 0
28 64 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 62 63
M SBL 1 1 69
M SMT 1 ^ CH2OH
M SBV 1 69 0.5786 -0.4853
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 64 65
M SBL 2 1 71
M SMT 2 ^ CH2OH
M SBV 2 71 0.6144 0.2134
S SKP 5
ID FL5FACGL0071
FORMULA C41H44O24
EXACTMASS 920.222252336
AVERAGEMASS 920.77366
SMILES C(OC(C(OC(O7)C(C(C(C7)O)O)OC(C=Cc(c6)cc(O)c(O)c6)=O)5)OC(CO)C(C(O)5)O)(C(=O)2)=C(Oc(c3)c2c(cc(OC(C(O)4)OC(CO)C(O)C4O)3)O)c(c1)ccc(O)c1O
M END