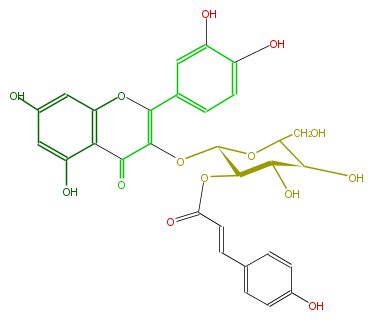
Mol:FL5FACGL0057
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.7098 0.9669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7098 0.9669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7098 0.3245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7098 0.3245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1535 0.0033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1535 0.0033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5972 0.3245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5972 0.3245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5972 0.9669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5972 0.9669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1535 1.2880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1535 1.2880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0409 0.0033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0409 0.0033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4846 0.3245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4846 0.3245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4846 0.9669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4846 0.9669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0409 1.2880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0409 1.2880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0409 -0.4975 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0409 -0.4975 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0715 1.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0715 1.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6385 0.9606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6385 0.9606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2055 1.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2055 1.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2055 1.9426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2055 1.9426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6385 2.2700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6385 2.2700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0715 1.9426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0715 1.9426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1535 -0.6388 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1535 -0.6388 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9992 2.3219 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9992 2.3219 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2047 1.2880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2047 1.2880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1292 -0.0299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1292 -0.0299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6385 2.9245 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6385 2.9245 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8601 0.2644 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 0.8601 0.2644 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 1.3025 -0.3196 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.3025 -0.3196 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.9396 -0.0718 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.9396 -0.0718 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.5955 -0.1477 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.5955 -0.1477 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.1076 0.3816 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.1076 0.3816 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.5503 0.0874 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5503 0.0874 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6128 -0.3532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6128 -0.3532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3046 -0.6846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3046 -0.6846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4912 -1.0750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4912 -1.0750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0628 -1.2441 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0628 -1.2441 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9466 -1.3379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9466 -1.3379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5747 -0.3505 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5747 -0.3505 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9466 -1.8603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9466 -1.8603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4323 -2.1407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4323 -2.1407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4323 -2.6632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4323 -2.6632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8848 -2.9245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8848 -2.9245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3373 -2.6632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3373 -2.6632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3373 -2.1407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3373 -2.1407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8848 -1.8794 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8848 -1.8794 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7770 -2.9170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7770 -2.9170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4578 0.5361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4578 0.5361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5658 1.5302 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5658 1.5302 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 1 1 0 0 0 0 | + | 20 1 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 16 22 1 0 0 0 0 | + | 16 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 24 29 1 0 0 0 0 | + | 24 29 1 0 0 0 0 |
| − | 25 30 1 0 0 0 0 | + | 25 30 1 0 0 0 0 |
| − | 21 23 1 0 0 0 0 | + | 21 23 1 0 0 0 0 |
| − | 29 31 1 0 0 0 0 | + | 29 31 1 0 0 0 0 |
| − | 31 32 2 0 0 0 0 | + | 31 32 2 0 0 0 0 |
| − | 31 33 1 0 0 0 0 | + | 31 33 1 0 0 0 0 |
| − | 26 34 1 0 0 0 0 | + | 26 34 1 0 0 0 0 |
| − | 33 35 2 0 0 0 0 | + | 33 35 2 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 2 0 0 0 0 | + | 40 41 2 0 0 0 0 |
| − | 41 36 1 0 0 0 0 | + | 41 36 1 0 0 0 0 |
| − | 39 42 1 0 0 0 0 | + | 39 42 1 0 0 0 0 |
| − | 27 43 1 0 0 0 0 | + | 27 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 43 44 | + | M SAL 1 2 43 44 |
| − | M SBL 1 1 47 | + | M SBL 1 1 47 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 47 2.4903 0.3736 | + | M SVB 1 47 2.4903 0.3736 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FACGL0057 | + | ID FL5FACGL0057 |
| − | KNApSAcK_ID C00005959 | + | KNApSAcK_ID C00005959 |
| − | NAME Quercetin 3-(2''-p-coumarylglucoside) | + | NAME Quercetin 3-(2''-p-coumarylglucoside) |
| − | CAS_RN 115909-21-2 | + | CAS_RN 115909-21-2 |
| − | FORMULA C30H26O14 | + | FORMULA C30H26O14 |
| − | EXACTMASS 610.13225554 | + | EXACTMASS 610.13225554 |
| − | AVERAGEMASS 610.51904 | + | AVERAGEMASS 610.51904 |
| − | SMILES c(c1)c(C(=C3O[C@@H](O4)[C@H](OC(C=Cc(c5)ccc(c5)O)=O)[C@H](O)[C@H](O)C(CO)4)Oc(c2)c(C3=O)c(O)cc(O)2)cc(c1O)O | + | SMILES c(c1)c(C(=C3O[C@@H](O4)[C@H](OC(C=Cc(c5)ccc(c5)O)=O)[C@H](O)[C@H](O)C(CO)4)Oc(c2)c(C3=O)c(O)cc(O)2)cc(c1O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
-2.7098 0.9669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7098 0.3245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1535 0.0033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5972 0.3245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5972 0.9669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1535 1.2880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0409 0.0033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4846 0.3245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4846 0.9669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0409 1.2880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0409 -0.4975 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0715 1.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6385 0.9606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2055 1.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2055 1.9426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6385 2.2700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0715 1.9426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1535 -0.6388 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9992 2.3219 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2047 1.2880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1292 -0.0299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6385 2.9245 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8601 0.2644 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
1.3025 -0.3196 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.9396 -0.0718 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.5955 -0.1477 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.1076 0.3816 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.5503 0.0874 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6128 -0.3532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3046 -0.6846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4912 -1.0750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0628 -1.2441 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9466 -1.3379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5747 -0.3505 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9466 -1.8603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4323 -2.1407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4323 -2.6632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8848 -2.9245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3373 -2.6632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3373 -2.1407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8848 -1.8794 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7770 -2.9170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4578 0.5361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5658 1.5302 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
20 1 1 0 0 0 0
21 8 1 0 0 0 0
16 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
24 29 1 0 0 0 0
25 30 1 0 0 0 0
21 23 1 0 0 0 0
29 31 1 0 0 0 0
31 32 2 0 0 0 0
31 33 1 0 0 0 0
26 34 1 0 0 0 0
33 35 2 0 0 0 0
35 36 1 0 0 0 0
36 37 2 0 0 0 0
37 38 1 0 0 0 0
38 39 2 0 0 0 0
39 40 1 0 0 0 0
40 41 2 0 0 0 0
41 36 1 0 0 0 0
39 42 1 0 0 0 0
27 43 1 0 0 0 0
43 44 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 43 44
M SBL 1 1 47
M SMT 1 CH2OH
M SVB 1 47 2.4903 0.3736
S SKP 8
ID FL5FACGL0057
KNApSAcK_ID C00005959
NAME Quercetin 3-(2''-p-coumarylglucoside)
CAS_RN 115909-21-2
FORMULA C30H26O14
EXACTMASS 610.13225554
AVERAGEMASS 610.51904
SMILES c(c1)c(C(=C3O[C@@H](O4)[C@H](OC(C=Cc(c5)ccc(c5)O)=O)[C@H](O)[C@H](O)C(CO)4)Oc(c2)c(C3=O)c(O)cc(O)2)cc(c1O)O
M END