Mol:FL5FACGA0029
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 46 0 0 0 0 0 0 0 0999 V2000 | + | 43 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -4.4387 0.8179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4387 0.8179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4387 0.1756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4387 0.1756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8824 -0.1456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8824 -0.1456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3261 0.1756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3261 0.1756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3261 0.8179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3261 0.8179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8824 1.1391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8824 1.1391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7698 -0.1456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7698 -0.1456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2135 0.1756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2135 0.1756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2135 0.8179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2135 0.8179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7698 1.1391 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7698 1.1391 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7698 -0.6465 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7698 -0.6465 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6574 1.1390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6574 1.1390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0904 0.8116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0904 0.8116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5234 1.1390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5234 1.1390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5234 1.7937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5234 1.7937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0904 2.1210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0904 2.1210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6574 1.7937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6574 1.7937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5653 -0.3747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5653 -0.3747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5199 0.1167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5199 0.1167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9076 -0.5547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9076 -0.5547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1621 -0.3417 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1621 -0.3417 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5879 -0.5547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5879 -0.5547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9756 0.1167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9756 0.1167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2301 -0.0963 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2301 -0.0963 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2470 -0.0781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2470 -0.0781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4989 0.3693 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4989 0.3693 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9756 0.8799 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9756 0.8799 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0434 2.1209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0434 2.1209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8824 -0.7877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8824 -0.7877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0496 1.0164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0496 1.0164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2168 -0.5547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2168 -0.5547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5624 -1.1533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5624 -1.1533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5632 -1.8839 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5632 -1.8839 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9074 -2.2626 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9074 -2.2626 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3057 -2.3583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3057 -2.3583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0904 2.7755 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0904 2.7755 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0276 -2.0489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0276 -2.0489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7016 -2.4381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7016 -2.4381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0276 -1.1412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0276 -1.1412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3756 -2.0490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3756 -2.0490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0496 -2.4381 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0496 -2.4381 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3756 -1.2707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3756 -1.2707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0276 -2.7755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0276 -2.7755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 24 26 1 0 0 0 0 | + | 24 26 1 0 0 0 0 |
| − | 23 27 1 0 0 0 0 | + | 23 27 1 0 0 0 0 |
| − | 20 18 1 0 0 0 0 | + | 20 18 1 0 0 0 0 |
| − | 18 8 1 0 0 0 0 | + | 18 8 1 0 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 15 28 1 0 0 0 0 | + | 15 28 1 0 0 0 0 |
| − | 3 29 1 0 0 0 0 | + | 3 29 1 0 0 0 0 |
| − | 1 30 1 0 0 0 0 | + | 1 30 1 0 0 0 0 |
| − | 22 31 1 0 0 0 0 | + | 22 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 2 0 0 0 0 | + | 33 34 2 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 16 36 1 0 0 0 0 | + | 16 36 1 0 0 0 0 |
| − | 35 37 1 0 0 0 0 | + | 35 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 37 39 1 0 0 0 0 | + | 37 39 1 0 0 0 0 |
| − | 38 40 1 0 0 0 0 | + | 38 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 40 42 2 0 0 0 0 | + | 40 42 2 0 0 0 0 |
| − | 37 43 1 0 0 0 0 | + | 37 43 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FACGA0029 | + | ID FL5FACGA0029 |
| − | KNApSAcK_ID C00005948 | + | KNApSAcK_ID C00005948 |
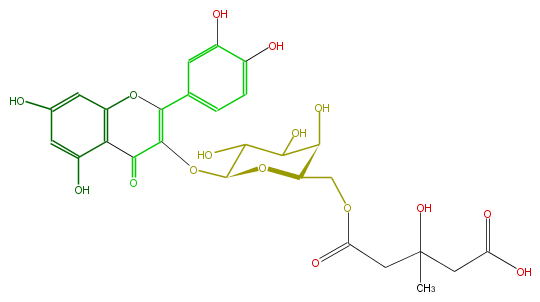
| − | NAME Quercetin 3-[6''-(3-hydroxy-3-methylglutaryl)galactoside] | + | NAME Quercetin 3-[6''-(3-hydroxy-3-methylglutaryl)galactoside] |
| − | CAS_RN 106915-93-9 | + | CAS_RN 106915-93-9 |
| − | FORMULA C27H28O16 | + | FORMULA C27H28O16 |
| − | EXACTMASS 608.137734848 | + | EXACTMASS 608.137734848 |
| − | AVERAGEMASS 608.50162 | + | AVERAGEMASS 608.50162 |
| − | SMILES OC(C(O)1)C(C(OC1OC(=C2c(c4)ccc(O)c4O)C(c(c(O)3)c(cc(O)c3)O2)=O)COC(=O)CC(CC(O)=O)(C)O)O | + | SMILES OC(C(O)1)C(C(OC1OC(=C2c(c4)ccc(O)c4O)C(c(c(O)3)c(cc(O)c3)O2)=O)COC(=O)CC(CC(O)=O)(C)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 46 0 0 0 0 0 0 0 0999 V2000
-4.4387 0.8179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4387 0.1756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8824 -0.1456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3261 0.1756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3261 0.8179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8824 1.1391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7698 -0.1456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2135 0.1756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2135 0.8179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7698 1.1391 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7698 -0.6465 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6574 1.1390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0904 0.8116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5234 1.1390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5234 1.7937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0904 2.1210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6574 1.7937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5653 -0.3747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5199 0.1167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9076 -0.5547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1621 -0.3417 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5879 -0.5547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9756 0.1167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2301 -0.0963 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2470 -0.0781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4989 0.3693 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9756 0.8799 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0434 2.1209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8824 -0.7877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0496 1.0164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2168 -0.5547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5624 -1.1533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5632 -1.8839 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9074 -2.2626 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3057 -2.3583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0904 2.7755 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0276 -2.0489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7016 -2.4381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0276 -1.1412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3756 -2.0490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0496 -2.4381 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3756 -1.2707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0276 -2.7755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
19 20 1 0 0 0 0
20 21 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
19 25 1 0 0 0 0
24 26 1 0 0 0 0
23 27 1 0 0 0 0
20 18 1 0 0 0 0
18 8 1 0 0 0 0
22 21 1 1 0 0 0
15 28 1 0 0 0 0
3 29 1 0 0 0 0
1 30 1 0 0 0 0
22 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 2 0 0 0 0
33 35 1 0 0 0 0
16 36 1 0 0 0 0
35 37 1 0 0 0 0
37 38 1 0 0 0 0
37 39 1 0 0 0 0
38 40 1 0 0 0 0
40 41 1 0 0 0 0
40 42 2 0 0 0 0
37 43 1 0 0 0 0
S SKP 8
ID FL5FACGA0029
KNApSAcK_ID C00005948
NAME Quercetin 3-[6''-(3-hydroxy-3-methylglutaryl)galactoside]
CAS_RN 106915-93-9
FORMULA C27H28O16
EXACTMASS 608.137734848
AVERAGEMASS 608.50162
SMILES OC(C(O)1)C(C(OC1OC(=C2c(c4)ccc(O)c4O)C(c(c(O)3)c(cc(O)c3)O2)=O)COC(=O)CC(CC(O)=O)(C)O)O
M END