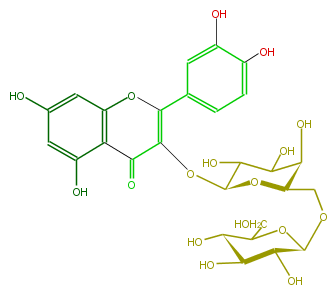
Mol:FL5FACGA0008
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.4755 0.7124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4755 0.7124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4755 0.0701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4755 0.0701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9192 -0.2511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9192 -0.2511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3629 0.0701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3629 0.0701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3629 0.7124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3629 0.7124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9192 1.0336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9192 1.0336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8066 -0.2511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8066 -0.2511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2503 0.0701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2503 0.0701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2503 0.7124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2503 0.7124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8066 1.0336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8066 1.0336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8066 -0.7520 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8066 -0.7520 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3058 1.0335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3058 1.0335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8728 0.7061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8728 0.7061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4398 1.0335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4398 1.0335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4398 1.6882 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4398 1.6882 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8728 2.0155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8728 2.0155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3058 1.6882 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3058 1.6882 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0316 1.0335 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0316 1.0335 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9192 -0.8932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9192 -0.8932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8194 1.9074 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8194 1.9074 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3669 -0.3195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3669 -0.3195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0537 -0.8620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0537 -0.8620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6560 -0.6899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6560 -0.6899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2620 -0.8620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2620 -0.8620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5753 -0.3195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5753 -0.3195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9729 -0.4916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9729 -0.4916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8250 -0.3195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8250 -0.3195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2209 -0.0620 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2209 -0.0620 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5753 0.4650 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5753 0.4650 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8363 -0.8620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8363 -0.8620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9993 -1.7542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9993 -1.7542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4262 -2.3178 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4262 -2.3178 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0410 -2.0787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0410 -2.0787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6342 -2.0723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6342 -2.0723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2031 -1.6411 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2031 -1.6411 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5751 -1.8666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5751 -1.8666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5250 -1.8813 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5250 -1.8813 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7606 -2.3502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7606 -2.3502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3932 -2.6700 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3932 -2.6700 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0316 -1.3840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0316 -1.3840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3739 -0.5261 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3739 -0.5261 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8728 2.6700 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8728 2.6700 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8438 -1.5314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8438 -1.5314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0469 -1.3179 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0469 -1.3179 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 15 1 0 0 0 0 | + | 20 15 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 40 30 1 0 0 0 0 | + | 40 30 1 0 0 0 0 |
| − | 22 41 1 0 0 0 0 | + | 22 41 1 0 0 0 0 |
| − | 41 8 1 0 0 0 0 | + | 41 8 1 0 0 0 0 |
| − | 16 42 1 0 0 0 0 | + | 16 42 1 0 0 0 0 |
| − | 36 43 1 0 0 0 0 | + | 36 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 43 44 | + | M SAL 1 2 43 44 |
| − | M SBL 1 1 47 | + | M SBL 1 1 47 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 47 -7.9776 6.8433 | + | M SBV 1 47 -7.9776 6.8433 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FACGA0008 | + | ID FL5FACGA0008 |
| − | KNApSAcK_ID C00005399 | + | KNApSAcK_ID C00005399 |
| − | NAME Quercetin 3-glucosyl-(1->6)-galactoside | + | NAME Quercetin 3-glucosyl-(1->6)-galactoside |
| − | CAS_RN 90366-14-6 | + | CAS_RN 90366-14-6 |
| − | FORMULA C27H30O17 | + | FORMULA C27H30O17 |
| − | EXACTMASS 626.148299534 | + | EXACTMASS 626.148299534 |
| − | AVERAGEMASS 626.5169000000001 | + | AVERAGEMASS 626.5169000000001 |
| − | SMILES c(c(O)5)cc(cc5O)C(O3)=C(C(c(c(O)4)c(cc(O)c4)3)=O)OC(O1)C(C(O)C(C(COC(C2O)OC(C(C(O)2)O)CO)1)O)O | + | SMILES c(c(O)5)cc(cc5O)C(O3)=C(C(c(c(O)4)c(cc(O)c4)3)=O)OC(O1)C(C(O)C(C(COC(C2O)OC(C(C(O)2)O)CO)1)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
-2.4755 0.7124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4755 0.0701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9192 -0.2511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3629 0.0701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3629 0.7124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9192 1.0336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8066 -0.2511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2503 0.0701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2503 0.7124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8066 1.0336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8066 -0.7520 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3058 1.0335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8728 0.7061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4398 1.0335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4398 1.6882 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8728 2.0155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3058 1.6882 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0316 1.0335 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9192 -0.8932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8194 1.9074 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3669 -0.3195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0537 -0.8620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6560 -0.6899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2620 -0.8620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5753 -0.3195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9729 -0.4916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8250 -0.3195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2209 -0.0620 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5753 0.4650 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8363 -0.8620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9993 -1.7542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4262 -2.3178 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0410 -2.0787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6342 -2.0723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2031 -1.6411 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5751 -1.8666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5250 -1.8813 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7606 -2.3502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3932 -2.6700 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0316 -1.3840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3739 -0.5261 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8728 2.6700 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8438 -1.5314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0469 -1.3179 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
3 19 1 0 0 0 0
20 15 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
24 30 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 40 1 0 0 0 0
40 30 1 0 0 0 0
22 41 1 0 0 0 0
41 8 1 0 0 0 0
16 42 1 0 0 0 0
36 43 1 0 0 0 0
43 44 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 43 44
M SBL 1 1 47
M SMT 1 ^CH2OH
M SBV 1 47 -7.9776 6.8433
S SKP 8
ID FL5FACGA0008
KNApSAcK_ID C00005399
NAME Quercetin 3-glucosyl-(1->6)-galactoside
CAS_RN 90366-14-6
FORMULA C27H30O17
EXACTMASS 626.148299534
AVERAGEMASS 626.5169000000001
SMILES c(c(O)5)cc(cc5O)C(O3)=C(C(c(c(O)4)c(cc(O)c4)3)=O)OC(O1)C(C(O)C(C(COC(C2O)OC(C(C(O)2)O)CO)1)O)O
M END