Mol:FL5FABGI0023
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 65 70 0 0 0 0 0 0 0 0999 V2000 | + | 65 70 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.9921 0.7638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9921 0.7638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9921 0.1214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9921 0.1214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4358 -0.1998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4358 -0.1998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1205 0.1214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1205 0.1214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1205 0.7638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1205 0.7638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4358 1.0850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4358 1.0850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6768 -0.1998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6768 -0.1998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2331 0.1214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2331 0.1214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2331 0.7638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2331 0.7638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6768 1.0850 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6768 1.0850 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6768 -0.7006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6768 -0.7006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9336 1.2086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9336 1.2086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5006 0.8812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5006 0.8812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0675 1.2086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0675 1.2086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0675 1.8633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0675 1.8633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5006 2.1906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5006 2.1906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9336 1.8633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9336 1.8633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6566 1.1474 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6566 1.1474 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4358 -0.7625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4358 -0.7625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8170 -0.2675 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8170 -0.2675 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3747 -1.3300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3747 -1.3300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9101 -1.6391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9101 -1.6391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7403 -1.0447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7403 -1.0447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9101 -0.4466 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9101 -0.4466 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3747 -0.1374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3747 -0.1374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5446 -0.7319 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5446 -0.7319 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2011 -0.7319 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2011 -0.7319 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5177 -1.8635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5177 -1.8635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9101 -2.1666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9101 -2.1666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2689 -1.3498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2689 -1.3498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7496 0.9258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7496 0.9258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3784 0.4358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3784 0.4358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8439 0.6437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8439 0.6437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3281 0.6493 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3281 0.6493 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7029 1.0241 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7029 1.0241 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1705 0.7773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1705 0.7773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2634 0.6291 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2634 0.6291 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9570 0.4077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9570 0.4077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5376 0.1296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5376 0.1296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4786 -2.1267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4786 -2.1267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8497 -2.6167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8497 -2.6167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3842 -2.4088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3842 -2.4088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9000 -2.4032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9000 -2.4032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5252 -2.0284 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5252 -2.0284 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0576 -2.2752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0576 -2.2752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0353 -2.4234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0353 -2.4234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2711 -2.6448 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2711 -2.6448 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6905 -2.9229 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6905 -2.9229 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4358 1.7271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4358 1.7271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9919 2.0481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9919 2.0481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9919 2.6903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9919 2.6903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4358 3.0113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4358 3.0113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5480 3.0113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5480 3.0113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3323 -2.5887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3323 -2.5887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0883 -3.0113 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0883 -3.0113 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9024 -2.5887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9024 -2.5887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5348 -1.7524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5348 -1.7524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0286 -1.9034 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0286 -1.9034 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5783 -1.9034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5783 -1.9034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9826 -1.4990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9826 -1.4990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8794 -2.4250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8794 -2.4250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5690 1.5615 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5690 1.5615 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7721 1.3480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7721 1.3480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5489 2.2948 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5489 2.2948 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2634 1.8823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2634 1.8823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 19 3 1 0 0 0 0 | + | 19 3 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 26 21 1 1 0 0 0 | + | 26 21 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 21 28 1 0 0 0 0 | + | 21 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 18 1 0 0 0 0 | + | 34 18 1 0 0 0 0 |
| − | 40 41 1 1 0 0 0 | + | 40 41 1 1 0 0 0 |
| − | 41 42 1 1 0 0 0 | + | 41 42 1 1 0 0 0 |
| − | 43 42 1 1 0 0 0 | + | 43 42 1 1 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 40 1 0 0 0 0 | + | 45 40 1 0 0 0 0 |
| − | 40 46 1 0 0 0 0 | + | 40 46 1 0 0 0 0 |
| − | 41 47 1 0 0 0 0 | + | 41 47 1 0 0 0 0 |
| − | 42 48 1 0 0 0 0 | + | 42 48 1 0 0 0 0 |
| − | 43 28 1 0 0 0 0 | + | 43 28 1 0 0 0 0 |
| − | 6 49 1 0 0 0 0 | + | 6 49 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 51 2 0 0 0 0 | + | 50 51 2 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 51 53 1 0 0 0 0 | + | 51 53 1 0 0 0 0 |
| − | 29 54 1 0 0 0 0 | + | 29 54 1 0 0 0 0 |
| − | 54 55 2 0 0 0 0 | + | 54 55 2 0 0 0 0 |
| − | 54 56 1 0 0 0 0 | + | 54 56 1 0 0 0 0 |
| − | 45 57 1 0 0 0 0 | + | 45 57 1 0 0 0 0 |
| − | 57 58 1 0 0 0 0 | + | 57 58 1 0 0 0 0 |
| − | 58 59 1 0 0 0 0 | + | 58 59 1 0 0 0 0 |
| − | 59 60 2 0 0 0 0 | + | 59 60 2 0 0 0 0 |
| − | 59 61 1 0 0 0 0 | + | 59 61 1 0 0 0 0 |
| − | 36 62 1 0 0 0 0 | + | 36 62 1 0 0 0 0 |
| − | 62 63 1 0 0 0 0 | + | 62 63 1 0 0 0 0 |
| − | 15 64 1 0 0 0 0 | + | 15 64 1 0 0 0 0 |
| − | 64 65 1 0 0 0 0 | + | 64 65 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 62 63 | + | M SAL 1 2 62 63 |
| − | M SBL 1 1 67 | + | M SBL 1 1 67 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 67 -6.9345 8.0036 | + | M SBV 1 67 -6.9345 8.0036 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 64 65 | + | M SAL 2 2 64 65 |
| − | M SBL 2 1 69 | + | M SBL 2 1 69 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SBV 2 69 -6.0547 7.6509 | + | M SBV 2 69 -6.0547 7.6509 |
| − | S SKP 8 | + | S SKP 8 |
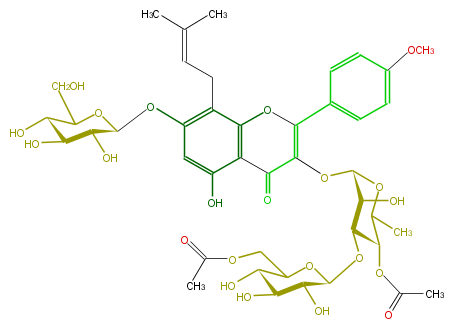
| − | ID FL5FABGI0023 | + | ID FL5FABGI0023 |
| − | KNApSAcK_ID C00006060 | + | KNApSAcK_ID C00006060 |
| − | NAME Epimedokoreanoside I | + | NAME Epimedokoreanoside I |
| − | CAS_RN 130756-11-5 | + | CAS_RN 130756-11-5 |
| − | FORMULA C43H54O22 | + | FORMULA C43H54O22 |
| − | EXACTMASS 922.310673412 | + | EXACTMASS 922.310673412 |
| − | AVERAGEMASS 922.87566 | + | AVERAGEMASS 922.87566 |
| − | SMILES C(C1O)(O)C(Oc(c6)c(CC=C(C)C)c(c(c(O)6)4)OC(c(c5)ccc(c5)OC)=C(C(=O)4)OC(O2)C(O)C(OC(O3)C(C(C(O)C(COC(C)=O)3)O)O)C(OC(C)=O)C2C)OC(C(O)1)CO | + | SMILES C(C1O)(O)C(Oc(c6)c(CC=C(C)C)c(c(c(O)6)4)OC(c(c5)ccc(c5)OC)=C(C(=O)4)OC(O2)C(O)C(OC(O3)C(C(C(O)C(COC(C)=O)3)O)O)C(OC(C)=O)C2C)OC(C(O)1)CO |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
65 70 0 0 0 0 0 0 0 0999 V2000
-0.9921 0.7638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9921 0.1214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4358 -0.1998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1205 0.1214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1205 0.7638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4358 1.0850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6768 -0.1998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2331 0.1214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2331 0.7638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6768 1.0850 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6768 -0.7006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9336 1.2086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5006 0.8812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0675 1.2086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0675 1.8633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5006 2.1906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9336 1.8633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6566 1.1474 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4358 -0.7625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8170 -0.2675 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3747 -1.3300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9101 -1.6391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7403 -1.0447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9101 -0.4466 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3747 -0.1374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5446 -0.7319 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2011 -0.7319 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5177 -1.8635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9101 -2.1666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2689 -1.3498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7496 0.9258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3784 0.4358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8439 0.6437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3281 0.6493 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7029 1.0241 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1705 0.7773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2634 0.6291 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9570 0.4077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5376 0.1296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4786 -2.1267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8497 -2.6167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3842 -2.4088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9000 -2.4032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5252 -2.0284 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0576 -2.2752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0353 -2.4234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2711 -2.6448 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6905 -2.9229 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4358 1.7271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9919 2.0481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9919 2.6903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4358 3.0113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5480 3.0113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3323 -2.5887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0883 -3.0113 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9024 -2.5887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5348 -1.7524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0286 -1.9034 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5783 -1.9034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9826 -1.4990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8794 -2.4250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5690 1.5615 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7721 1.3480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5489 2.2948 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2634 1.8823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
4 3 1 0 0 0 0
1 18 1 0 0 0 0
19 3 1 0 0 0 0
8 20 1 0 0 0 0
22 21 1 1 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 1 0 0 0
26 21 1 1 0 0 0
26 27 1 0 0 0 0
21 28 1 0 0 0 0
22 29 1 0 0 0 0
23 30 1 0 0 0 0
25 20 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 18 1 0 0 0 0
40 41 1 1 0 0 0
41 42 1 1 0 0 0
43 42 1 1 0 0 0
43 44 1 0 0 0 0
44 45 1 0 0 0 0
45 40 1 0 0 0 0
40 46 1 0 0 0 0
41 47 1 0 0 0 0
42 48 1 0 0 0 0
43 28 1 0 0 0 0
6 49 1 0 0 0 0
49 50 1 0 0 0 0
50 51 2 0 0 0 0
51 52 1 0 0 0 0
51 53 1 0 0 0 0
29 54 1 0 0 0 0
54 55 2 0 0 0 0
54 56 1 0 0 0 0
45 57 1 0 0 0 0
57 58 1 0 0 0 0
58 59 1 0 0 0 0
59 60 2 0 0 0 0
59 61 1 0 0 0 0
36 62 1 0 0 0 0
62 63 1 0 0 0 0
15 64 1 0 0 0 0
64 65 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 62 63
M SBL 1 1 67
M SMT 1 ^CH2OH
M SBV 1 67 -6.9345 8.0036
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 64 65
M SBL 2 1 69
M SMT 2 OCH3
M SBV 2 69 -6.0547 7.6509
S SKP 8
ID FL5FABGI0023
KNApSAcK_ID C00006060
NAME Epimedokoreanoside I
CAS_RN 130756-11-5
FORMULA C43H54O22
EXACTMASS 922.310673412
AVERAGEMASS 922.87566
SMILES C(C1O)(O)C(Oc(c6)c(CC=C(C)C)c(c(c(O)6)4)OC(c(c5)ccc(c5)OC)=C(C(=O)4)OC(O2)C(O)C(OC(O3)C(C(C(O)C(COC(C)=O)3)O)O)C(OC(C)=O)C2C)OC(C(O)1)CO
M END