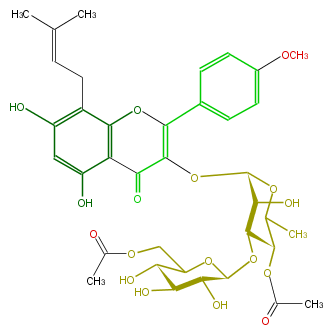
Mol:FL5FABGI0019
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 54 58 0 0 0 0 0 0 0 0999 V2000 | + | 54 58 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.3382 0.8242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3382 0.8242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3382 0.1818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3382 0.1818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7819 -0.1394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7819 -0.1394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2256 0.1818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2256 0.1818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2256 0.8242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2256 0.8242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7819 1.1454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7819 1.1454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6693 -0.1394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6693 -0.1394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1130 0.1818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1130 0.1818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1130 0.8242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1130 0.8242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6693 1.1454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6693 1.1454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6693 -0.6402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6693 -0.6402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5875 1.2690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5875 1.2690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1544 0.9417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1544 0.9417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7214 1.2690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7214 1.2690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7214 1.9237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7214 1.9237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1544 2.2510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1544 2.2510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5875 1.9237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5875 1.9237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0027 1.2078 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0027 1.2078 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7819 -0.7021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7819 -0.7021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4709 -0.2071 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4709 -0.2071 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5236 -1.3108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5236 -1.3108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0590 -1.6199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0590 -1.6199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8891 -1.0255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8891 -1.0255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0590 -0.4275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0590 -0.4275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5236 -0.1183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5236 -0.1183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6935 -0.7128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6935 -0.7128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3500 -0.7128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3500 -0.7128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6611 -1.8241 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6611 -1.8241 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9299 -2.1020 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9299 -2.1020 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4177 -1.3307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4177 -1.3307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7819 1.7875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7819 1.7875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3380 2.1086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3380 2.1086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3380 2.7493 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3380 2.7493 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7831 3.0697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7831 3.0697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8929 3.0697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8929 3.0697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2900 -1.9632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2900 -1.9632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0811 -2.4531 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0811 -2.4531 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6156 -2.2453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6156 -2.2453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1314 -2.2397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1314 -2.2397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7566 -1.8648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7566 -1.8648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2890 -2.1116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2890 -2.1116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8039 -2.2598 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8039 -2.2598 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4975 -2.4813 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4975 -2.4813 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9219 -2.7594 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9219 -2.7594 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2320 -1.5906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2320 -1.5906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7849 -1.7388 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7849 -1.7388 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3072 -1.7388 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3072 -1.7388 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5439 -1.3287 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5439 -1.3287 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6133 -2.2689 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6133 -2.2689 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0997 -2.7360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0997 -2.7360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9071 -3.0697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9071 -3.0697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7403 -2.7360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7403 -2.7360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2882 2.2509 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2882 2.2509 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0027 1.8384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0027 1.8384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 19 3 1 0 0 0 0 | + | 19 3 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 26 21 1 1 0 0 0 | + | 26 21 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 21 28 1 0 0 0 0 | + | 21 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 6 31 1 0 0 0 0 | + | 6 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 37 38 1 1 0 0 0 | + | 37 38 1 1 0 0 0 |
| − | 39 38 1 1 0 0 0 | + | 39 38 1 1 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 36 1 0 0 0 0 | + | 41 36 1 0 0 0 0 |
| − | 36 42 1 0 0 0 0 | + | 36 42 1 0 0 0 0 |
| − | 37 43 1 0 0 0 0 | + | 37 43 1 0 0 0 0 |
| − | 38 44 1 0 0 0 0 | + | 38 44 1 0 0 0 0 |
| − | 39 28 1 0 0 0 0 | + | 39 28 1 0 0 0 0 |
| − | 41 45 1 0 0 0 0 | + | 41 45 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 2 0 0 0 0 | + | 47 48 2 0 0 0 0 |
| − | 47 49 1 0 0 0 0 | + | 47 49 1 0 0 0 0 |
| − | 29 50 1 0 0 0 0 | + | 29 50 1 0 0 0 0 |
| − | 50 51 2 0 0 0 0 | + | 50 51 2 0 0 0 0 |
| − | 50 52 1 0 0 0 0 | + | 50 52 1 0 0 0 0 |
| − | 15 53 1 0 0 0 0 | + | 15 53 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 53 54 | + | M SAL 1 2 53 54 |
| − | M SBL 1 1 57 | + | M SBL 1 1 57 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 57 -4.2216 4.2864 | + | M SBV 1 57 -4.2216 4.2864 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FABGI0019 | + | ID FL5FABGI0019 |
| − | KNApSAcK_ID C00006056 | + | KNApSAcK_ID C00006056 |
| − | NAME Epimedoside | + | NAME Epimedoside |
| − | CAS_RN 106441-31-0 | + | CAS_RN 106441-31-0 |
| − | FORMULA C37H44O17 | + | FORMULA C37H44O17 |
| − | EXACTMASS 760.2578499819999 | + | EXACTMASS 760.2578499819999 |
| − | AVERAGEMASS 760.73506 | + | AVERAGEMASS 760.73506 |
| − | SMILES C(C)(=O)OC(C1C)C(OC(O5)C(C(O)C(O)C5COC(C)=O)O)C(C(OC(C(=O)2)=C(c(c4)ccc(c4)OC)Oc(c(CC=C(C)C)3)c2c(cc(O)3)O)O1)O | + | SMILES C(C)(=O)OC(C1C)C(OC(O5)C(C(O)C(O)C5COC(C)=O)O)C(C(OC(C(=O)2)=C(c(c4)ccc(c4)OC)Oc(c(CC=C(C)C)3)c2c(cc(O)3)O)O1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
54 58 0 0 0 0 0 0 0 0999 V2000
-2.3382 0.8242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3382 0.1818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7819 -0.1394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2256 0.1818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2256 0.8242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7819 1.1454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6693 -0.1394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1130 0.1818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1130 0.8242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6693 1.1454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6693 -0.6402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5875 1.2690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1544 0.9417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7214 1.2690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7214 1.9237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1544 2.2510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5875 1.9237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0027 1.2078 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7819 -0.7021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4709 -0.2071 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5236 -1.3108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0590 -1.6199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8891 -1.0255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0590 -0.4275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5236 -0.1183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6935 -0.7128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3500 -0.7128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6611 -1.8241 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9299 -2.1020 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4177 -1.3307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7819 1.7875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3380 2.1086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3380 2.7493 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7831 3.0697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8929 3.0697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2900 -1.9632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0811 -2.4531 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6156 -2.2453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1314 -2.2397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7566 -1.8648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2890 -2.1116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8039 -2.2598 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4975 -2.4813 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9219 -2.7594 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2320 -1.5906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7849 -1.7388 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3072 -1.7388 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5439 -1.3287 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6133 -2.2689 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0997 -2.7360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9071 -3.0697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7403 -2.7360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2882 2.2509 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0027 1.8384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
4 3 1 0 0 0 0
1 18 1 0 0 0 0
19 3 1 0 0 0 0
8 20 1 0 0 0 0
22 21 1 1 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 1 0 0 0
26 21 1 1 0 0 0
26 27 1 0 0 0 0
21 28 1 0 0 0 0
22 29 1 0 0 0 0
23 30 1 0 0 0 0
25 20 1 0 0 0 0
6 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 2 0 0 0 0
33 34 1 0 0 0 0
33 35 1 0 0 0 0
36 37 1 1 0 0 0
37 38 1 1 0 0 0
39 38 1 1 0 0 0
39 40 1 0 0 0 0
40 41 1 0 0 0 0
41 36 1 0 0 0 0
36 42 1 0 0 0 0
37 43 1 0 0 0 0
38 44 1 0 0 0 0
39 28 1 0 0 0 0
41 45 1 0 0 0 0
45 46 1 0 0 0 0
46 47 1 0 0 0 0
47 48 2 0 0 0 0
47 49 1 0 0 0 0
29 50 1 0 0 0 0
50 51 2 0 0 0 0
50 52 1 0 0 0 0
15 53 1 0 0 0 0
53 54 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 53 54
M SBL 1 1 57
M SMT 1 OCH3
M SBV 1 57 -4.2216 4.2864
S SKP 8
ID FL5FABGI0019
KNApSAcK_ID C00006056
NAME Epimedoside
CAS_RN 106441-31-0
FORMULA C37H44O17
EXACTMASS 760.2578499819999
AVERAGEMASS 760.73506
SMILES C(C)(=O)OC(C1C)C(OC(O5)C(C(O)C(O)C5COC(C)=O)O)C(C(OC(C(=O)2)=C(c(c4)ccc(c4)OC)Oc(c(CC=C(C)C)3)c2c(cc(O)3)O)O1)O
M END