Mol:FL5FABGI0014
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 58 63 0 0 0 0 0 0 0 0999 V2000 | + | 58 63 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.4015 1.2679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4015 1.2679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4015 0.4425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4015 0.4425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6867 0.0298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6867 0.0298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0280 0.4425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0280 0.4425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0280 1.2679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0280 1.2679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6867 1.6806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6867 1.6806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7428 0.0298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7428 0.0298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4576 0.4425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4576 0.4425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4576 1.2679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4576 1.2679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7428 1.6806 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7428 1.6806 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7428 -0.6136 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7428 -0.6136 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1721 1.6805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1721 1.6805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9006 1.2598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9006 1.2598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6291 1.6805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6291 1.6805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6291 2.5216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6291 2.5216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9006 2.9422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9006 2.9422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1721 2.5216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1721 2.5216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1160 1.6805 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1160 1.6805 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3367 0.0034 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3367 0.0034 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6867 -0.7952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6867 -0.7952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6867 2.5056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6867 2.5056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4012 2.9182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4012 2.9182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4012 3.7432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4012 3.7432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6867 4.1556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6867 4.1556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1158 4.1556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1158 4.1556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6411 1.7677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6411 1.7677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1642 1.1381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1642 1.1381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4774 1.4052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4774 1.4052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8149 1.4124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8149 1.4124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2964 1.8940 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2964 1.8940 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8972 1.5769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8972 1.5769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2754 1.5234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2754 1.5234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8400 1.1423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8400 1.1423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8970 0.9702 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8970 0.9702 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0717 -1.4282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0717 -1.4282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7596 -1.8254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7596 -1.8254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5413 -1.0617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5413 -1.0617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7596 -0.2932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7596 -0.2932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0717 0.1040 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0717 0.1040 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2899 -0.6599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2899 -0.6599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2311 -0.7022 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2311 -0.7022 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1945 -2.1923 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1945 -2.1923 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7428 -2.3864 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7428 -2.3864 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0775 -1.3727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0775 -1.3727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0986 -3.1408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0986 -3.1408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9284 -3.7758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9284 -3.7758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4924 -3.4377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4924 -3.4377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1534 -3.4476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1534 -3.4476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3235 -2.8128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3235 -2.8128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7598 -3.1506 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7598 -3.1506 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3193 -4.1556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3193 -4.1556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2624 -3.6809 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2624 -3.6809 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3818 -3.2993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3818 -3.2993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2170 -4.0447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2170 -4.0447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3573 2.9421 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3573 2.9421 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2754 2.4121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2754 2.4121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3937 2.1281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3937 2.1281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7563 3.1241 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7563 3.1241 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 8 19 1 0 0 0 0 | + | 8 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 6 21 1 0 0 0 0 | + | 6 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 2 0 0 0 0 | + | 22 23 2 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 23 25 1 0 0 0 0 | + | 23 25 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 27 28 1 1 0 0 0 | + | 27 28 1 1 0 0 0 |
| − | 29 28 1 1 0 0 0 | + | 29 28 1 1 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 26 1 0 0 0 0 | + | 31 26 1 0 0 0 0 |
| − | 26 32 1 0 0 0 0 | + | 26 32 1 0 0 0 0 |
| − | 27 33 1 0 0 0 0 | + | 27 33 1 0 0 0 0 |
| − | 28 34 1 0 0 0 0 | + | 28 34 1 0 0 0 0 |
| − | 29 18 1 0 0 0 0 | + | 29 18 1 0 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 1 1 0 0 0 | + | 39 40 1 1 0 0 0 |
| − | 40 35 1 1 0 0 0 | + | 40 35 1 1 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 35 42 1 0 0 0 0 | + | 35 42 1 0 0 0 0 |
| − | 36 43 1 0 0 0 0 | + | 36 43 1 0 0 0 0 |
| − | 37 44 1 0 0 0 0 | + | 37 44 1 0 0 0 0 |
| − | 39 19 1 0 0 0 0 | + | 39 19 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 1 1 0 0 0 | + | 46 47 1 1 0 0 0 |
| − | 47 48 1 1 0 0 0 | + | 47 48 1 1 0 0 0 |
| − | 49 48 1 1 0 0 0 | + | 49 48 1 1 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 45 1 0 0 0 0 | + | 50 45 1 0 0 0 0 |
| − | 47 51 1 0 0 0 0 | + | 47 51 1 0 0 0 0 |
| − | 46 52 1 0 0 0 0 | + | 46 52 1 0 0 0 0 |
| − | 45 53 1 0 0 0 0 | + | 45 53 1 0 0 0 0 |
| − | 48 54 1 0 0 0 0 | + | 48 54 1 0 0 0 0 |
| − | 42 49 1 0 0 0 0 | + | 42 49 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 15 55 1 0 0 0 0 | + | 15 55 1 0 0 0 0 |
| − | 57 58 1 0 0 0 0 | + | 57 58 1 0 0 0 0 |
| − | 31 57 1 0 0 0 0 | + | 31 57 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 55 56 | + | M SAL 1 2 55 56 |
| − | M SBL 1 1 61 | + | M SBL 1 1 61 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 61 -0.7283 -0.4205 | + | M SBV 1 61 -0.7283 -0.4205 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 57 58 | + | M SAL 2 2 57 58 |
| − | M SBL 2 1 63 | + | M SBL 2 1 63 |
| − | M SMT 2 ^ CH2OH | + | M SMT 2 ^ CH2OH |
| − | M SBV 2 63 0.4965 -0.5512 | + | M SBV 2 63 0.4965 -0.5512 |
| − | S SKP 5 | + | S SKP 5 |
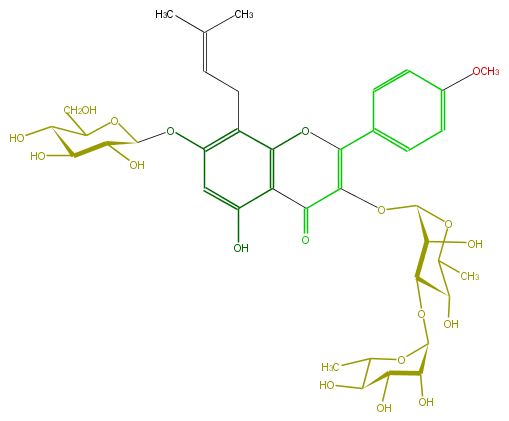
| − | ID FL5FABGI0014 | + | ID FL5FABGI0014 |
| − | FORMULA C39H50O19 | + | FORMULA C39H50O19 |
| − | EXACTMASS 822.294629418 | + | EXACTMASS 822.294629418 |
| − | AVERAGEMASS 822.8029 | + | AVERAGEMASS 822.8029 |
| − | SMILES C(OC(C(O)2)C(O)C(C)OC2OC(C6=O)=C(Oc(c46)c(CC=C(C)C)c(OC(O5)C(O)C(O)C(O)C(CO)5)cc4O)c(c3)ccc(c3)OC)(O1)C(O)C(O)C(O)C(C)1 | + | SMILES C(OC(C(O)2)C(O)C(C)OC2OC(C6=O)=C(Oc(c46)c(CC=C(C)C)c(OC(O5)C(O)C(O)C(O)C(CO)5)cc4O)c(c3)ccc(c3)OC)(O1)C(O)C(O)C(O)C(C)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
58 63 0 0 0 0 0 0 0 0999 V2000
-1.4015 1.2679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4015 0.4425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6867 0.0298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0280 0.4425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0280 1.2679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6867 1.6806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7428 0.0298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4576 0.4425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4576 1.2679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7428 1.6806 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7428 -0.6136 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1721 1.6805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9006 1.2598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6291 1.6805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6291 2.5216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9006 2.9422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1721 2.5216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1160 1.6805 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3367 0.0034 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6867 -0.7952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6867 2.5056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4012 2.9182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4012 3.7432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6867 4.1556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1158 4.1556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6411 1.7677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1642 1.1381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4774 1.4052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8149 1.4124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2964 1.8940 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8972 1.5769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2754 1.5234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8400 1.1423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8970 0.9702 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0717 -1.4282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7596 -1.8254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5413 -1.0617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7596 -0.2932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0717 0.1040 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2899 -0.6599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2311 -0.7022 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1945 -2.1923 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7428 -2.3864 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0775 -1.3727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0986 -3.1408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9284 -3.7758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4924 -3.4377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1534 -3.4476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3235 -2.8128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7598 -3.1506 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3193 -4.1556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2624 -3.6809 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3818 -3.2993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2170 -4.0447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3573 2.9421 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.2754 2.4121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3937 2.1281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7563 3.1241 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
8 19 1 0 0 0 0
3 20 1 0 0 0 0
6 21 1 0 0 0 0
21 22 1 0 0 0 0
22 23 2 0 0 0 0
23 24 1 0 0 0 0
23 25 1 0 0 0 0
26 27 1 1 0 0 0
27 28 1 1 0 0 0
29 28 1 1 0 0 0
29 30 1 0 0 0 0
30 31 1 0 0 0 0
31 26 1 0 0 0 0
26 32 1 0 0 0 0
27 33 1 0 0 0 0
28 34 1 0 0 0 0
29 18 1 0 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
38 39 1 0 0 0 0
39 40 1 1 0 0 0
40 35 1 1 0 0 0
40 41 1 0 0 0 0
35 42 1 0 0 0 0
36 43 1 0 0 0 0
37 44 1 0 0 0 0
39 19 1 0 0 0 0
45 46 1 0 0 0 0
46 47 1 1 0 0 0
47 48 1 1 0 0 0
49 48 1 1 0 0 0
49 50 1 0 0 0 0
50 45 1 0 0 0 0
47 51 1 0 0 0 0
46 52 1 0 0 0 0
45 53 1 0 0 0 0
48 54 1 0 0 0 0
42 49 1 0 0 0 0
55 56 1 0 0 0 0
15 55 1 0 0 0 0
57 58 1 0 0 0 0
31 57 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 55 56
M SBL 1 1 61
M SMT 1 OCH3
M SBV 1 61 -0.7283 -0.4205
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 57 58
M SBL 2 1 63
M SMT 2 ^ CH2OH
M SBV 2 63 0.4965 -0.5512
S SKP 5
ID FL5FABGI0014
FORMULA C39H50O19
EXACTMASS 822.294629418
AVERAGEMASS 822.8029
SMILES C(OC(C(O)2)C(O)C(C)OC2OC(C6=O)=C(Oc(c46)c(CC=C(C)C)c(OC(O5)C(O)C(O)C(O)C(CO)5)cc4O)c(c3)ccc(c3)OC)(O1)C(O)C(O)C(O)C(C)1
M END