Mol:FL5FABGI0013
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 58 63 0 0 0 0 0 0 0 0999 V2000 | + | 58 63 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.6545 0.6711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6545 0.6711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6545 0.0287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6545 0.0287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0982 -0.2925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0982 -0.2925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5419 0.0287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5419 0.0287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5419 0.6711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5419 0.6711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0982 0.9922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0982 0.9922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0144 -0.2925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0144 -0.2925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5707 0.0287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5707 0.0287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5707 0.6711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5707 0.6711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0144 0.9922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0144 0.9922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0144 -0.7933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0144 -0.7933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2712 1.1159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2712 1.1159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8382 0.7885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8382 0.7885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4052 1.1159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4052 1.1159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4052 1.7706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4052 1.7706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8382 2.0979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8382 2.0979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2712 1.7706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2712 1.7706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0982 -0.9346 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0982 -0.9346 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1494 0.9922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1494 0.9922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1340 -0.3808 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1340 -0.3808 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0982 1.6344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0982 1.6344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4085 0.6176 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -4.4085 0.6176 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -3.9816 0.0540 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.9816 0.0540 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.3668 0.2931 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.3668 0.2931 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.7736 0.2995 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.7736 0.2995 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.2046 0.7307 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2046 0.7307 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8326 0.5052 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -3.8326 0.5052 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -4.9575 0.6657 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9575 0.6657 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3846 -0.4765 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3846 -0.4765 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0145 -0.2983 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0145 -0.2983 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6543 1.9554 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6543 1.9554 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6543 2.5976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6543 2.5976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2104 2.9186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2104 2.9186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0982 2.9186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0982 2.9186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9599 -1.4746 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 1.9599 -1.4746 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 2.5881 -1.8373 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.5881 -1.8373 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.3887 -1.1398 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.3887 -1.1398 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.5881 -0.4381 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5881 -0.4381 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9599 -0.0753 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.9599 -0.0753 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.1591 -0.7729 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.1591 -0.7729 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.9295 -0.7729 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9295 -0.7729 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1212 -2.0768 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1212 -2.0768 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4365 -2.4030 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4365 -2.4030 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0090 -1.4979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0090 -1.4979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8367 -1.9902 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 3.8367 -1.9902 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 4.4650 -2.3529 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 4.4650 -2.3529 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 4.2656 -1.6554 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 4.2656 -1.6554 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 4.4650 -0.9537 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4650 -0.9537 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8367 -0.5909 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.8367 -0.5909 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 4.0360 -1.2885 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 4.0360 -1.2885 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 4.8064 -1.2885 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8064 -1.2885 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9981 -2.5924 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9981 -2.5924 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3134 -2.9186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3134 -2.9186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8859 -2.0135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8859 -2.0135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0120 2.1209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0120 2.1209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7264 1.7084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7264 1.7084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0679 1.2310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0679 1.2310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0459 1.4396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0459 1.4396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 1 1 0 0 0 0 | + | 19 1 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 6 21 1 0 0 0 0 | + | 6 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 25 19 1 0 0 0 0 | + | 25 19 1 0 0 0 0 |
| − | 21 31 1 0 0 0 0 | + | 21 31 1 0 0 0 0 |
| − | 31 32 2 0 0 0 0 | + | 31 32 2 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 1 1 0 0 0 | + | 39 40 1 1 0 0 0 |
| − | 40 35 1 1 0 0 0 | + | 40 35 1 1 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 35 42 1 0 0 0 0 | + | 35 42 1 0 0 0 0 |
| − | 36 43 1 0 0 0 0 | + | 36 43 1 0 0 0 0 |
| − | 37 44 1 0 0 0 0 | + | 37 44 1 0 0 0 0 |
| − | 39 20 1 0 0 0 0 | + | 39 20 1 0 0 0 0 |
| − | 46 45 1 1 0 0 0 | + | 46 45 1 1 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 1 1 0 0 0 | + | 49 50 1 1 0 0 0 |
| − | 50 45 1 1 0 0 0 | + | 50 45 1 1 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 45 52 1 0 0 0 0 | + | 45 52 1 0 0 0 0 |
| − | 46 53 1 0 0 0 0 | + | 46 53 1 0 0 0 0 |
| − | 47 54 1 0 0 0 0 | + | 47 54 1 0 0 0 0 |
| − | 41 49 1 0 0 0 0 | + | 41 49 1 0 0 0 0 |
| − | 15 55 1 0 0 0 0 | + | 15 55 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 27 57 1 0 0 0 0 | + | 27 57 1 0 0 0 0 |
| − | 57 58 1 0 0 0 0 | + | 57 58 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 57 58 | + | M SAL 2 2 57 58 |
| − | M SBL 2 1 62 | + | M SBL 2 1 62 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 62 -4.0679 1.231 | + | M SVB 2 62 -4.0679 1.231 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 55 56 | + | M SAL 1 2 55 56 |
| − | M SBL 1 1 60 | + | M SBL 1 1 60 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 60 3.012 2.1209 | + | M SVB 1 60 3.012 2.1209 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FABGI0013 | + | ID FL5FABGI0013 |
| − | KNApSAcK_ID C00005830 | + | KNApSAcK_ID C00005830 |
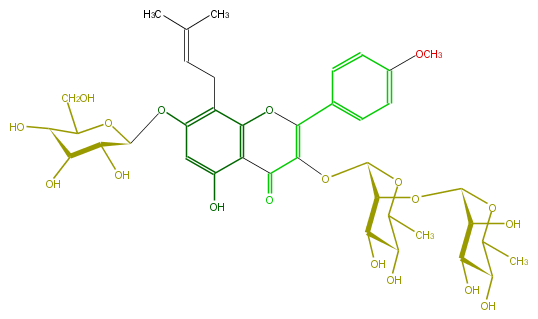
| − | NAME Epimedin C | + | NAME Epimedin C |
| − | CAS_RN 110642-44-9 | + | CAS_RN 110642-44-9 |
| − | FORMULA C39H50O19 | + | FORMULA C39H50O19 |
| − | EXACTMASS 822.294629418 | + | EXACTMASS 822.294629418 |
| − | AVERAGEMASS 822.8029 | + | AVERAGEMASS 822.8029 |
| − | SMILES C(O1)([C@H]([C@@H](O)[C@H](O)[C@H]1O[C@H]([C@H]2OC(C3=O)=C(c(c6)ccc(c6)OC)Oc(c4CC=C(C)C)c3c(cc4O[C@H](O5)[C@H]([C@@H](O)[C@@H](O)C5CO)O)O)[C@@H]([C@@H](C(C)O2)O)O)O)C | + | SMILES C(O1)([C@H]([C@@H](O)[C@H](O)[C@H]1O[C@H]([C@H]2OC(C3=O)=C(c(c6)ccc(c6)OC)Oc(c4CC=C(C)C)c3c(cc4O[C@H](O5)[C@H]([C@@H](O)[C@@H](O)C5CO)O)O)[C@@H]([C@@H](C(C)O2)O)O)O)C |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
58 63 0 0 0 0 0 0 0 0999 V2000
-1.6545 0.6711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6545 0.0287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0982 -0.2925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5419 0.0287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5419 0.6711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0982 0.9922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0144 -0.2925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5707 0.0287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5707 0.6711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0144 0.9922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0144 -0.7933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2712 1.1159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8382 0.7885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4052 1.1159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4052 1.7706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8382 2.0979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2712 1.7706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0982 -0.9346 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1494 0.9922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1340 -0.3808 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0982 1.6344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4085 0.6176 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-3.9816 0.0540 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.3668 0.2931 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.7736 0.2995 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.2046 0.7307 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8326 0.5052 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-4.9575 0.6657 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3846 -0.4765 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0145 -0.2983 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6543 1.9554 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6543 2.5976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2104 2.9186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0982 2.9186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9599 -1.4746 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
2.5881 -1.8373 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.3887 -1.1398 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.5881 -0.4381 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9599 -0.0753 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.1591 -0.7729 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.9295 -0.7729 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1212 -2.0768 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4365 -2.4030 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0090 -1.4979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8367 -1.9902 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
4.4650 -2.3529 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
4.2656 -1.6554 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
4.4650 -0.9537 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8367 -0.5909 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
4.0360 -1.2885 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
4.8064 -1.2885 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9981 -2.5924 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3134 -2.9186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8859 -2.0135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0120 2.1209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7264 1.7084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0679 1.2310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.0459 1.4396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 1 1 0 0 0 0
20 8 1 0 0 0 0
4 3 1 0 0 0 0
6 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
25 19 1 0 0 0 0
21 31 1 0 0 0 0
31 32 2 0 0 0 0
32 33 1 0 0 0 0
32 34 1 0 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
38 39 1 0 0 0 0
39 40 1 1 0 0 0
40 35 1 1 0 0 0
40 41 1 0 0 0 0
35 42 1 0 0 0 0
36 43 1 0 0 0 0
37 44 1 0 0 0 0
39 20 1 0 0 0 0
46 45 1 1 0 0 0
46 47 1 0 0 0 0
47 48 1 0 0 0 0
48 49 1 0 0 0 0
49 50 1 1 0 0 0
50 45 1 1 0 0 0
50 51 1 0 0 0 0
45 52 1 0 0 0 0
46 53 1 0 0 0 0
47 54 1 0 0 0 0
41 49 1 0 0 0 0
15 55 1 0 0 0 0
55 56 1 0 0 0 0
27 57 1 0 0 0 0
57 58 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 57 58
M SBL 2 1 62
M SMT 2 CH2OH
M SVB 2 62 -4.0679 1.231
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 55 56
M SBL 1 1 60
M SMT 1 OCH3
M SVB 1 60 3.012 2.1209
S SKP 8
ID FL5FABGI0013
KNApSAcK_ID C00005830
NAME Epimedin C
CAS_RN 110642-44-9
FORMULA C39H50O19
EXACTMASS 822.294629418
AVERAGEMASS 822.8029
SMILES C(O1)([C@H]([C@@H](O)[C@H](O)[C@H]1O[C@H]([C@H]2OC(C3=O)=C(c(c6)ccc(c6)OC)Oc(c4CC=C(C)C)c3c(cc4O[C@H](O5)[C@H]([C@@H](O)[C@@H](O)C5CO)O)O)[C@@H]([C@@H](C(C)O2)O)O)O)C
M END