Mol:FL5FABGI0007
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 48 52 0 0 0 0 0 0 0 0999 V2000 | + | 48 52 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.0670 0.3823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0670 0.3823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0670 -0.2600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0670 -0.2600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5107 -0.5812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5107 -0.5812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0456 -0.2600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0456 -0.2600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0456 0.3823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0456 0.3823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5107 0.7035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5107 0.7035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6019 -0.5812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6019 -0.5812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1582 -0.2600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1582 -0.2600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1582 0.3823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1582 0.3823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6019 0.7035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6019 0.7035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6019 -1.0821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6019 -1.0821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7143 0.7034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7143 0.7034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2813 0.3760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2813 0.3760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8482 0.7034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8482 0.7034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8482 1.3581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8482 1.3581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2813 1.6854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2813 1.6854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7143 1.3581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7143 1.3581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5107 -1.2234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5107 -1.2234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7143 -0.5811 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7143 -0.5811 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6779 0.5808 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6779 0.5808 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5107 1.3456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5107 1.3456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0668 1.6667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0668 1.6667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0668 2.3088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0668 2.3088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6229 2.6299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6229 2.6299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5107 2.6299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5107 2.6299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8245 0.3587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8245 0.3587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4533 -0.1313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4533 -0.1313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9188 0.0766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9188 0.0766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4031 0.0822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4031 0.0822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7778 0.4570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7778 0.4570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2454 0.2102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2454 0.2102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3383 0.0620 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3383 0.0620 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0319 -0.1594 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0319 -0.1594 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6126 -0.4375 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6126 -0.4375 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7742 -1.7015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7742 -1.7015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4024 -2.0642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4024 -2.0642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2031 -1.3667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2031 -1.3667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4024 -0.6650 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4024 -0.6650 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7742 -0.3022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7742 -0.3022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9735 -0.9997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9735 -0.9997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7438 -0.9997 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7438 -0.9997 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9356 -2.3037 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9356 -2.3037 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2508 -2.6299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2508 -2.6299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8233 -1.7248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8233 -1.7248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6239 1.7379 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6239 1.7379 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3383 1.3254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3383 1.3254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3195 0.6087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3195 0.6087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1164 0.8222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1164 0.8222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 8 19 1 0 0 0 0 | + | 8 19 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 6 21 1 0 0 0 0 | + | 6 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 2 0 0 0 0 | + | 22 23 2 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 23 25 1 0 0 0 0 | + | 23 25 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 27 28 1 1 0 0 0 | + | 27 28 1 1 0 0 0 |
| − | 29 28 1 1 0 0 0 | + | 29 28 1 1 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 26 1 0 0 0 0 | + | 31 26 1 0 0 0 0 |
| − | 26 32 1 0 0 0 0 | + | 26 32 1 0 0 0 0 |
| − | 27 33 1 0 0 0 0 | + | 27 33 1 0 0 0 0 |
| − | 28 34 1 0 0 0 0 | + | 28 34 1 0 0 0 0 |
| − | 29 20 1 0 0 0 0 | + | 29 20 1 0 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 1 1 0 0 0 | + | 39 40 1 1 0 0 0 |
| − | 40 35 1 1 0 0 0 | + | 40 35 1 1 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 35 42 1 0 0 0 0 | + | 35 42 1 0 0 0 0 |
| − | 36 43 1 0 0 0 0 | + | 36 43 1 0 0 0 0 |
| − | 37 44 1 0 0 0 0 | + | 37 44 1 0 0 0 0 |
| − | 39 19 1 0 0 0 0 | + | 39 19 1 0 0 0 0 |
| − | 15 45 1 0 0 0 0 | + | 15 45 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 31 47 1 0 0 0 0 | + | 31 47 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 45 46 | + | M SAL 1 2 45 46 |
| − | M SBL 1 1 49 | + | M SBL 1 1 49 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 49 -4.3710 5.0109 | + | M SBV 1 49 -4.3710 5.0109 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 47 48 | + | M SAL 2 2 47 48 |
| − | M SBL 2 1 51 | + | M SBL 2 1 51 |
| − | M SMT 2 ^CH2OH | + | M SMT 2 ^CH2OH |
| − | M SBV 2 51 -5.2207 5.0295 | + | M SBV 2 51 -5.2207 5.0295 |
| − | S SKP 8 | + | S SKP 8 |
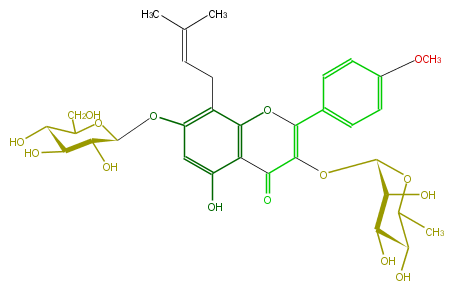
| − | ID FL5FABGI0007 | + | ID FL5FABGI0007 |
| − | KNApSAcK_ID C00005824 | + | KNApSAcK_ID C00005824 |
| − | NAME Icariin | + | NAME Icariin |
| − | CAS_RN 489-32-7 | + | CAS_RN 489-32-7 |
| − | FORMULA C33H40O15 | + | FORMULA C33H40O15 |
| − | EXACTMASS 676.23672061 | + | EXACTMASS 676.23672061 |
| − | AVERAGEMASS 676.6617 | + | AVERAGEMASS 676.6617 |
| − | SMILES C(C5O)(OC(C(O)C5O)CO)Oc(c(CC=C(C)C)1)cc(O)c(C3=O)c1OC(=C3OC(O4)C(O)C(O)C(C(C)4)O)c(c2)ccc(c2)OC | + | SMILES C(C5O)(OC(C(O)C5O)CO)Oc(c(CC=C(C)C)1)cc(O)c(C3=O)c1OC(=C3OC(O4)C(O)C(O)C(C(C)4)O)c(c2)ccc(c2)OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
48 52 0 0 0 0 0 0 0 0999 V2000
-1.0670 0.3823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0670 -0.2600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5107 -0.5812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0456 -0.2600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0456 0.3823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5107 0.7035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6019 -0.5812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1582 -0.2600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1582 0.3823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6019 0.7035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6019 -1.0821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7143 0.7034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2813 0.3760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8482 0.7034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8482 1.3581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2813 1.6854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7143 1.3581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5107 -1.2234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7143 -0.5811 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6779 0.5808 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5107 1.3456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0668 1.6667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0668 2.3088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6229 2.6299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5107 2.6299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8245 0.3587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4533 -0.1313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9188 0.0766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4031 0.0822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7778 0.4570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2454 0.2102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3383 0.0620 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0319 -0.1594 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6126 -0.4375 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7742 -1.7015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4024 -2.0642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2031 -1.3667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4024 -0.6650 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7742 -0.3022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9735 -0.9997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7438 -0.9997 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9356 -2.3037 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2508 -2.6299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8233 -1.7248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6239 1.7379 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3383 1.3254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3195 0.6087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1164 0.8222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
8 19 1 0 0 0 0
1 20 1 0 0 0 0
6 21 1 0 0 0 0
21 22 1 0 0 0 0
22 23 2 0 0 0 0
23 24 1 0 0 0 0
23 25 1 0 0 0 0
26 27 1 1 0 0 0
27 28 1 1 0 0 0
29 28 1 1 0 0 0
29 30 1 0 0 0 0
30 31 1 0 0 0 0
31 26 1 0 0 0 0
26 32 1 0 0 0 0
27 33 1 0 0 0 0
28 34 1 0 0 0 0
29 20 1 0 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
38 39 1 0 0 0 0
39 40 1 1 0 0 0
40 35 1 1 0 0 0
40 41 1 0 0 0 0
35 42 1 0 0 0 0
36 43 1 0 0 0 0
37 44 1 0 0 0 0
39 19 1 0 0 0 0
15 45 1 0 0 0 0
45 46 1 0 0 0 0
31 47 1 0 0 0 0
47 48 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 45 46
M SBL 1 1 49
M SMT 1 OCH3
M SBV 1 49 -4.3710 5.0109
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 47 48
M SBL 2 1 51
M SMT 2 ^CH2OH
M SBV 2 51 -5.2207 5.0295
S SKP 8
ID FL5FABGI0007
KNApSAcK_ID C00005824
NAME Icariin
CAS_RN 489-32-7
FORMULA C33H40O15
EXACTMASS 676.23672061
AVERAGEMASS 676.6617
SMILES C(C5O)(OC(C(O)C5O)CO)Oc(c(CC=C(C)C)1)cc(O)c(C3=O)c1OC(=C3OC(O4)C(O)C(O)C(C(C)4)O)c(c2)ccc(c2)OC
M END