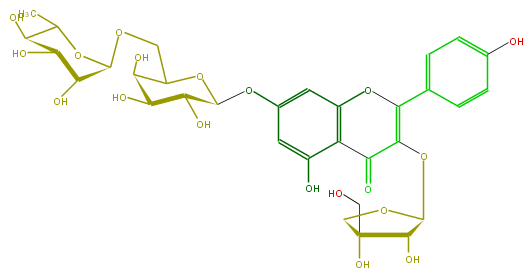
Mol:FL5FAAGS0092
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 51 56 0 0 0 0 0 0 0 0999 V2000 | + | 51 56 0 0 0 0 0 0 0 0999 V2000 |
| − | 3.9531 1.9798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9531 1.9798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9531 1.1548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9531 1.1548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6675 0.7423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6675 0.7423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3820 1.1548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3820 1.1548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3820 1.9798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3820 1.9798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6675 2.3923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6675 2.3923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2386 0.7423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2386 0.7423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5241 1.1548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5241 1.1548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8096 0.7423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8096 0.7423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8096 -0.0827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8096 -0.0827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5241 -0.4952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5241 -0.4952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2386 -0.0827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2386 -0.0827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0952 1.1548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0952 1.1548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3807 0.7423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3807 0.7423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3807 -0.0827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3807 -0.0827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0952 -0.4952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0952 -0.4952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5241 -1.2134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5241 -1.2134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3338 1.1548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3338 1.1548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8449 -0.4327 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8449 -0.4327 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9827 2.3267 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9827 2.3267 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0952 -1.1947 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0952 -1.1947 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2997 -2.3288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2997 -2.3288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2997 -3.0063 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2997 -3.0063 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4834 -2.8802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4834 -2.8802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3159 -1.5901 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3159 -1.5901 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8511 -1.9688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8511 -1.9688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9346 -1.9688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9346 -1.9688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4834 -2.3288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4834 -2.3288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8935 -1.7103 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8935 -1.7103 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8219 -1.3048 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8219 -1.3048 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0968 1.5174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0968 1.5174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6843 0.8029 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6843 0.8029 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8862 1.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8862 1.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0930 0.7852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0930 0.7852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5055 1.4998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5055 1.4998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3036 1.2908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3036 1.2908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5296 2.2165 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5296 2.2165 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4371 2.5465 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4371 2.5465 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9851 1.9344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9851 1.9344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3607 0.9842 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3607 0.9842 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4987 0.3409 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4987 0.3409 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9065 2.6632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9065 2.6632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6756 2.3649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6756 2.3649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9153 2.0445 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9153 2.0445 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4244 1.3814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4244 1.3814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6552 1.6797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6552 1.6797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4155 2.0001 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4155 2.0001 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9180 0.8879 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9180 0.8879 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7505 2.0445 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7505 2.0445 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.9827 2.8967 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.9827 2.8967 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5008 3.0063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5008 3.0063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 5 20 1 0 0 0 0 | + | 5 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 28 22 1 1 0 0 0 | + | 28 22 1 1 0 0 0 |
| − | 27 22 1 1 0 0 0 | + | 27 22 1 1 0 0 0 |
| − | 26 28 1 1 0 0 0 | + | 26 28 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 28 24 1 0 0 0 0 | + | 28 24 1 0 0 0 0 |
| − | 29 26 1 0 0 0 0 | + | 29 26 1 0 0 0 0 |
| − | 25 30 1 0 0 0 0 | + | 25 30 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 22 25 1 0 0 0 0 | + | 22 25 1 0 0 0 0 |
| − | 19 26 1 0 0 0 0 | + | 19 26 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 31 39 1 0 0 0 0 | + | 31 39 1 0 0 0 0 |
| − | 32 40 1 0 0 0 0 | + | 32 40 1 0 0 0 0 |
| − | 33 41 1 0 0 0 0 | + | 33 41 1 0 0 0 0 |
| − | 34 18 1 0 0 0 0 | + | 34 18 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 44 1 1 0 0 0 | + | 43 44 1 1 0 0 0 |
| − | 45 44 1 1 0 0 0 | + | 45 44 1 1 0 0 0 |
| − | 46 45 1 1 0 0 0 | + | 46 45 1 1 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 42 1 0 0 0 0 | + | 47 42 1 0 0 0 0 |
| − | 45 48 1 0 0 0 0 | + | 45 48 1 0 0 0 0 |
| − | 44 49 1 0 0 0 0 | + | 44 49 1 0 0 0 0 |
| − | 43 50 1 0 0 0 0 | + | 43 50 1 0 0 0 0 |
| − | 42 51 1 0 0 0 0 | + | 42 51 1 0 0 0 0 |
| − | 38 46 1 0 0 0 0 | + | 38 46 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAAGS0092 | + | ID FL5FAAGS0092 |
| − | FORMULA C32H38O19 | + | FORMULA C32H38O19 |
| − | EXACTMASS 726.200729034 | + | EXACTMASS 726.200729034 |
| − | AVERAGEMASS 726.6327200000001 | + | AVERAGEMASS 726.6327200000001 |
| − | SMILES C(C1O)(O)C(Oc(c6)cc(O4)c(c(O)6)C(C(=C4c(c5)ccc(O)c5)OC(C(O)3)OCC3(CO)O)=O)OC(COC(C2O)OC(C)C(C(O)2)O)C1O | + | SMILES C(C1O)(O)C(Oc(c6)cc(O4)c(c(O)6)C(C(=C4c(c5)ccc(O)c5)OC(C(O)3)OCC3(CO)O)=O)OC(COC(C2O)OC(C)C(C(O)2)O)C1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
51 56 0 0 0 0 0 0 0 0999 V2000
3.9531 1.9798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9531 1.1548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6675 0.7423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3820 1.1548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3820 1.9798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6675 2.3923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2386 0.7423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5241 1.1548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8096 0.7423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8096 -0.0827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5241 -0.4952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2386 -0.0827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0952 1.1548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3807 0.7423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3807 -0.0827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0952 -0.4952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5241 -1.2134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3338 1.1548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8449 -0.4327 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.9827 2.3267 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0952 -1.1947 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2997 -2.3288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2997 -3.0063 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4834 -2.8802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3159 -1.5901 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8511 -1.9688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9346 -1.9688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4834 -2.3288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8935 -1.7103 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8219 -1.3048 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0968 1.5174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6843 0.8029 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8862 1.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0930 0.7852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5055 1.4998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3036 1.2908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5296 2.2165 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4371 2.5465 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9851 1.9344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3607 0.9842 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4987 0.3409 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9065 2.6632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.6756 2.3649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9153 2.0445 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4244 1.3814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6552 1.6797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4155 2.0001 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9180 0.8879 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.7505 2.0445 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.9827 2.8967 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.5008 3.0063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
5 20 1 0 0 0 0
16 21 1 0 0 0 0
28 22 1 1 0 0 0
27 22 1 1 0 0 0
26 28 1 1 0 0 0
22 23 1 0 0 0 0
28 24 1 0 0 0 0
29 26 1 0 0 0 0
25 30 1 0 0 0 0
27 29 1 0 0 0 0
22 25 1 0 0 0 0
19 26 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
31 39 1 0 0 0 0
32 40 1 0 0 0 0
33 41 1 0 0 0 0
34 18 1 0 0 0 0
42 43 1 0 0 0 0
43 44 1 1 0 0 0
45 44 1 1 0 0 0
46 45 1 1 0 0 0
46 47 1 0 0 0 0
47 42 1 0 0 0 0
45 48 1 0 0 0 0
44 49 1 0 0 0 0
43 50 1 0 0 0 0
42 51 1 0 0 0 0
38 46 1 0 0 0 0
S SKP 5
ID FL5FAAGS0092
FORMULA C32H38O19
EXACTMASS 726.200729034
AVERAGEMASS 726.6327200000001
SMILES C(C1O)(O)C(Oc(c6)cc(O4)c(c(O)6)C(C(=C4c(c5)ccc(O)c5)OC(C(O)3)OCC3(CO)O)=O)OC(COC(C2O)OC(C)C(C(O)2)O)C1O
M END